原則上如果您的配備夠好,不需要看這篇,我電腦配備是 Intel i6, 18g RAM, 880g Hard disk, Nvidia RTX 4060 8g RAM, Ubuntu 24.01, CUDA 12.04, 所以guassian splatting以及會用到他的submodules的專案,像Sugar,4DGaussians等等都裝不起來.你可以參考這篇
0.更改.bashr下CUDA設定,讓RAM不碎片
export PYTORCH_CUDA_ALLOC_CONF=expandable_segments:True #https://docs.pytorch.org/docs/stable/notes/cuda.html#environment-variables
export PYTORCH_CUDA_ALLOC_CONF=max_split_size_mb:32 #https://blog.csdn.net/MirageTanker/article/details/127998036
export MAX_JOBS=2
1.裝gsplat要先安裝nerfstudio,我直接pip gsplat或從gsplat github source build,在ChatGPT/Gemini/Claud.ai幫忙下一直鬼打牆在修錯誤,修理好一個會跑出第二個問題,所以還是乖乖先裝nerfstudio
conda create -n nerfstudio python=3.10
conda activate nerfstudio
conda install pytorch torchvision torchaudio pytorch-cuda=12.1 -c pytorch -c nvidia --yes
pip install nerfstudio
你還是可以考慮下載測試用資料集,不過這些檔案都很大,8g vram測試沒辦法跑,跑是有跑但沒存任何結果檔
git clone https://github.com/nerfstudio-project/nerfstudio.git
cd nerfstudio
ns-download-data nerfstudio --capture-name=poster#Train model
ns-train nerfacto --data data/nerfstudio/poster
#其他檔案可以看 https://drive.google.com/drive/folders/19TV6kdVGcmg3cGZ1bNIUnBBMD-iQjRbG 列表
我是下載tank, 解壓縮,其中只要train/下面的檔案copy到 ./gsplat/examples/data/tank_dataset/train
再執行完整路徑指定處理後的數據位置
ns-process-data images
--data ~/gsplat/examples/data/tank_dataset/train/images
--output-dir ~/nerfstudio/outputs/tank_nerfstudio/
--colmap-model-path ~/gsplat/examples/data/new_tank/sparse/0
--skip-colmap
--verbose#然後訓練
ns-train nerfacto --data ~/nerfstudio/output/tank_nerfstudio/#在viser(我的在執行時,照螢幕顯示,在web開http://localhost:7007/), 照它操作, 但還是沒辦法render出mp4
torch.OutOfMemoryError: CUDA out of memory. Tried to allocate 320.00 MiB. GPU 0 has a total capacity of 7.74 GiB...#所以ns-viewer也沒用
2.好消息是gsplat可以裝起來,但要先安裝nerfstudio才不會缺一堆dependence,但因為我的被裝成nerfstudio 1.1.5,所以gsplat只能裝到1.4.0
git clone https://github.com/nerfstudio-project/gsplat.git
#確保您在 gsplat 倉庫的根目錄
cd gsplat
#清理所有未追蹤的文件和目錄 (非常重要!)
git clean -fdx
#重置所有本地更改,使其與當前分支或標籤一致
git reset --hard
#再次切換到 v1.4.0 (確保狀態正確)
git checkout v1.4.0
#再次初始化並更新子模組 (以防萬一,確保所有第三方庫都存在)
git submodule update --init --recursive
pip install -e .
#確保這兩個也已安裝且兼容
pip install fused-ssim pycolmap
#下面這2個我跳過
#cd examples
#pip install -r requirements.txt
下載測試檔案
Mip-NeRF 360對8g vram還是太大,如果你的gpu不陽春,照gsplat就好,或者下載Mip-NeRF 360,解壓縮到 gsplat/examples/data/360_v2/下
我是到https://www.kaggle.com/datasets/thnhdg/tandt-db 下載tank,解壓縮,其中只要train/下面的檔案copy到 ~/gsplat/examples/data/tank_dataset/train
修改 gsplat/examples/benchmarks/basic.sh成這個
SCENE_DIR="data"
RESULT_DIR="results/tank_benchmark"
SCENE_LIST="tank" # treehill flowers
RENDER_TRAJ_PATH="ellipse"
for SCENE in $SCENE_LIST;
do
# 例如:
if [ "$SCENE" = "tank" ]; then
DATA_FACTOR=1 # <--- 確保是 1
else
DATA_FACTOR=4 # 或者其他默認值
fiecho "Running $SCENE"
#train without eval
CUDA_VISIBLE_DEVICES=0 python simple_trainer.py default --eval_steps -1 --disable_viewer
--data_factor 1
--render_traj_path $RENDER_TRAJ_PATH
--data_dir data/tank_dataset/train/
--result_dir $RESULT_DIR/
#run eval and render
for CKPT in $RESULT_DIR/ckpts/*;
do
CUDA_VISIBLE_DEVICES=0 python simple_trainer.py default --disable_viewer
--data_factor 1
--render_traj_path $RENDER_TRAJ_PATH
--data_dir data/tank_dataset/train/
--result_dir $RESULT_DIR/
--ckpt $CKPT
done
done
for SCENE in $SCENE_LIST;
do
echo "=== Eval Stats ==="
for STATS in $RESULT_DIR/stats/val*.json;
do
echo $STATS
cat $STATS;
echo
done
echo "=== Train Stats ==="
for STATS in $RESULT_DIR/stats/train*_rank0.json;
do
echo $STATS
cat $STATS;
echo
done
done
修改colmap.py檔,
gedit gsplat/examples/datasets/colmap.py
查找 images_ 的構建邏輯,特別是導致 images_4 出現的那一行。通常會在 init 函數中。 您可能會看到類似這樣的部分(具體行數可能不同):
將 if data_factor > 1: 的邏輯修改為總是指向 images:
找到下面這段程式碼 (大約在第 153 行左右):, 將 for d in [image_dir, colmap_image_dir]: 這一行修改為:
# Load images.
# 簡化圖像資料夾處理邏輯
#確保 colmap_image_dir 和 image_dir 都指向原始的 'images' 資料夾
colmap_image_dir = os.path.join(data_dir, "images") # <--- 確保這行存在並正確
image_dir = colmap_image_dir # <--- image_dir 也指向同一個位置
#檢查圖像資料夾是否存在
if not os.path.exists(image_dir):
raise ValueError(f"Image folder {image_dir} does not exist.")
然後執行
bash benchmarks/basic_tank.sh
在gsplat/examples/results/tank_benchmark/videos有mp4
3.我另外找一個youtube影片,這影片中的010-014sec跟037-042sec,我用kazam得兩段mp4,再用ffmepg轉出jpg,存在~/gsplat/examples/data/liver/images下,
ffmpeg -i input1.mp4 -vf fps=20 image1%04d.jpg
ffmpeg -i input2.mp4 -vf fps=20 image2%04d.jpg
然後依樣修改benchmarks/basic.sh中的2段datadir改成--data_dir data/liver/,SCENE名為tank改成liver,讓gsplat跑看看,一樣執行bash benchmarks/basic.sh
看螢幕左邊可以看到兩隻器械可是分不出ligasure跟grasp,而右手器械不見.看螢幕左邊的肝臟S2,3跟右邊的S4並不漂亮,慘的是中間的falciform ligament和mesentery變得亂七八糟
4.再來就是安裝colmap從影像來萃取出自己要的,如果是用sudo apt install colmap安裝得到是CPU版本,我本來是照這個安裝,結果卻發覺conda環境有很大的問題,一大堆的錯誤,問了ChatGPT/Gemini/Claido.ai,解決一個錯誤又得到一個新的錯誤,一直在鬼打牆,半小時的安裝花了我10天,系統還被改得亂七八糟,所以我反而建議照colmap官網就好,不要在conda環境下compile colmap GPU版
#以下是修正被莫名其妙安裝不存在的檔案
#Glog 0.8.0 尚未正式發布,修正sudo nano /usr/lib/x86_64-linux-gnu/cmake/glog/glog-targets-noconfig.cmakels -l /usr/lib/x86_64-linux-gnu/libglog.so*
#其中2行有/lib/x86_64-linux-gnu/libglog.so.0.8.0改為0.6.0#只留系統apt裝的libglog.so.0.6.0
sudo rm /usr/local/lib/libglog.so*
sudo rm -rf /usr/local/include/glog
sudo rm -rf /usr/local/lib/cmake/glog
sudo ldconfig # 更新庫緩存#確認你有 libglog.so.0.6.0 和 libglog.so.1
ls -l /usr/lib/x86_64-linux-gnu/libglog.so*#降級G++ 版本
sudo apt update
sudo apt install gcc-11 g++-11
sudo update-alternatives --install /usr/bin/gcc gcc /usr/bin/gcc-11 110
--slave /usr/bin/g++ g++ /usr/bin/g++-11
sudo update-alternatives --install /usr/bin/gcc gcc /usr/bin/gcc-12 120
--slave /usr/bin/g++ g++ /usr/bin/g++-12
sudo update-alternatives --install /usr/bin/gcc gcc /usr/bin/gcc-13 130
--slave /usr/bin/g++ g++ /usr/bin/g++-13
#如果你有更高版本,也一樣添加,數值越大優先級越高
sudo update-alternatives --config gcc
#在列表中選擇 gcc-11 的對應數字export CUDAHOSTCXX=/usr/bin/g++
開始安裝
sudo apt-get install \
git \
cmake \
ninja-build \
build-essential \
libboost-program-options-dev \
libboost-graph-dev \
libboost-system-dev \
libeigen3-dev \
libflann-dev \
libfreeimage-dev \
libmetis-dev \
libgoogle-glog-dev \
libgtest-dev \
libgmock-dev \
libsqlite3-dev \
libglew-dev \
qtbase5-dev \
libqt5opengl5-dev \
libcgal-dev \
libceres-dev
sudo apt-get install -y \
nvidia-cuda-toolkit \
nvidia-cuda-toolkit-gcc
git clone https://github.com/colmap/colmap.git
cd colmap
mkdir build
cd build
rm -rf CMakeCache.txt CMakeFiles/
cmake .. -GNinja -DCUDA_ENABLED=ON #不需要設定-DCUDA_ARCH_BIN="8.9"
ninja -j8
sudo ninja install
sudo ninja install
自己從tank image建 spares/下的3個.bin
#colmap -h
colmap automatic_reconstructor \
--workspace_path ~/gsplat/examples/data/new_tank_gpu \
--image_path ~/gsplat/examples/data/tank_dataset/train/images
另外一個terminal
nvidia-smi -l 1
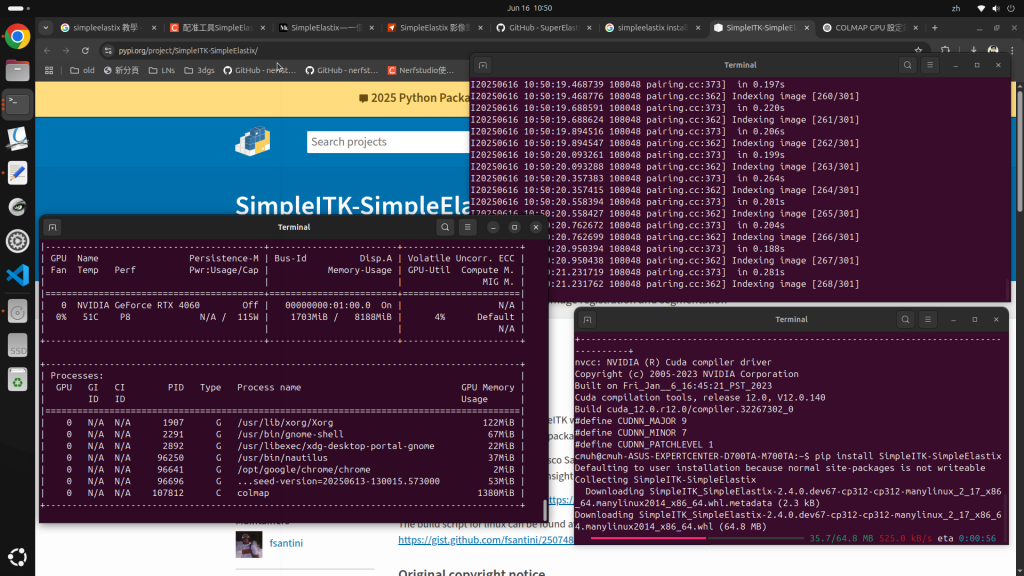
理論上COLMAP GPU 支援主要集中在以下步驟:Feature extraction(SIFT),Feature matching(matching),但Structure-from-Motion(SfM)和Bundle Adjustment(BA)仍然是 CPU-based。可是我實測只有在matching一小段時間有用到GPU, 可以看到colmap有佔了GPU vram 1380M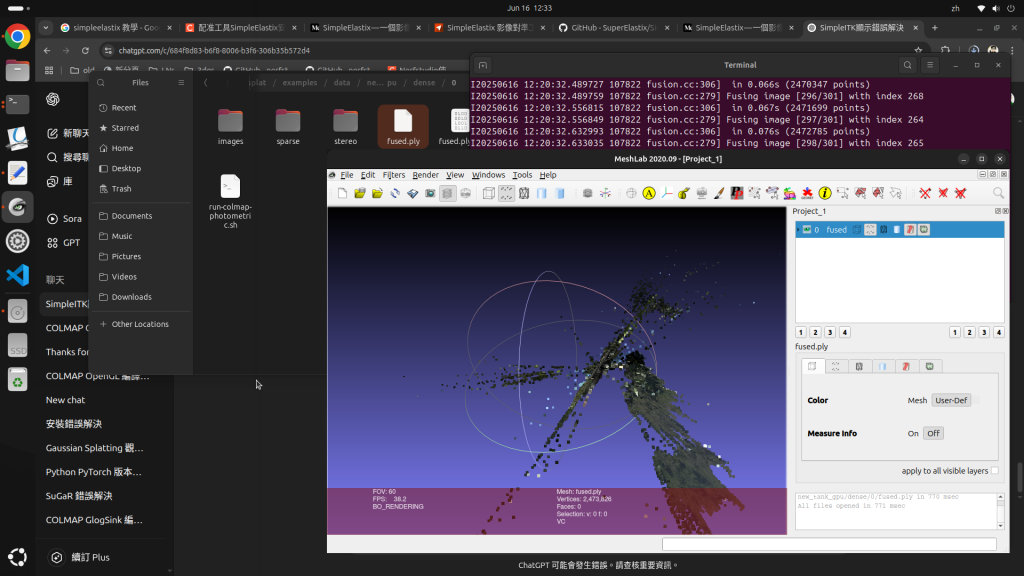
5.因為MITI dataset的干擾比較大,在肝臟表面都可以看到反光,所以我另外找一個youtube影片,這影片中的010-014sec跟037-042sec,我用kazam得兩段mp4,再用ffmepg轉出jpg,存在~/gsplat/examples/data/liver/images下
再試看看colmap
colmap automatic_reconstructor \
--workspace_path ~/gsplat/examples/data/liver \
--image_path ~/gsplat/examples/data/liver/images
在~/gsplat/examples/data/liver/dense/1/fused.ply長這樣子,效果不好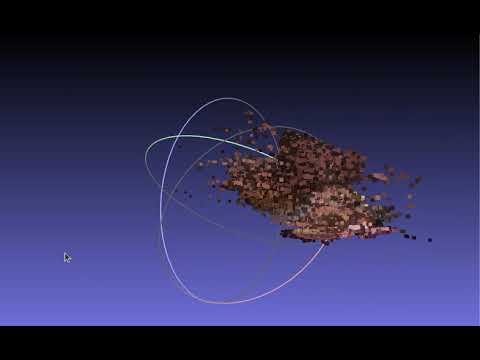
原來軟組織真的難搞,難怪一堆研究報告的各種評分都只有到6成或更慘,不像segmentation的paper的DICE都可以到90以上.
