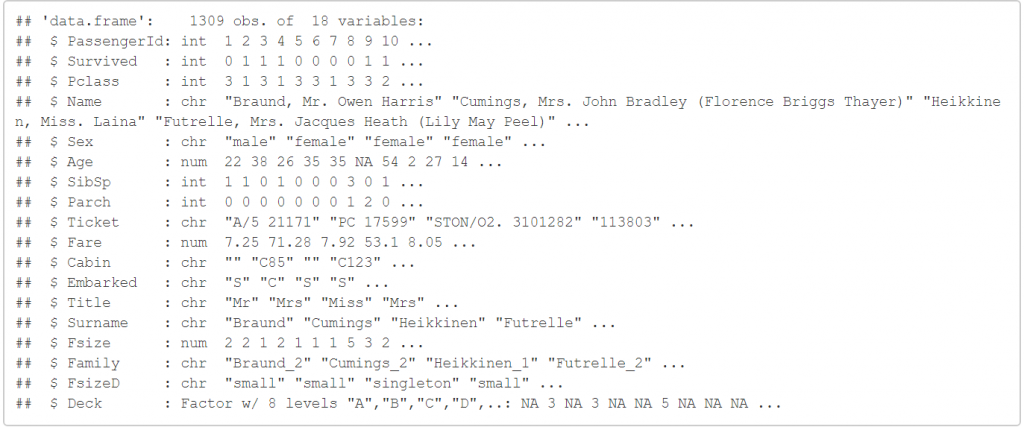
接續上次的文章,今天我們會針對 缺失值 做處理,我們先來看上次的資料最後的型態。
str(full)
我們先用 table 來看一下 Embarked 有幾種不同的資料。
table(full$Embarked)

會發現有兩個空白的值,接著我們來實際找出他們。
filter(full,full$Embarked=="")

我們可以看到在第62跟830筆資料是空缺的。那我們有很多種方法可以處理缺失值,可以把他移除,或是幫助他補值,在決定怎麼做之前,先來看看其他資料長的怎麼樣吧!我們使用 Embarked(上船港口) 作為x軸,票價為y軸,並且根據不同票艙的等級各自做出三個盒狀圖。
# Get rid of our missing passenger IDs
embark_fare <- full %>%
filter(PassengerId != 62 & PassengerId != 830)
# Use ggplot2 to visualize embarkment, passenger class, & median fare
ggplot(embark_fare, aes(x = Embarked, y = Fare, fill = factor(Pclass))) +
geom_boxplot() +
geom_hline(aes(yintercept=80),
colour='red', linetype='dashed', lwd=2) +
scale_y_continuous(labels=dollar_format()) +
theme_few()
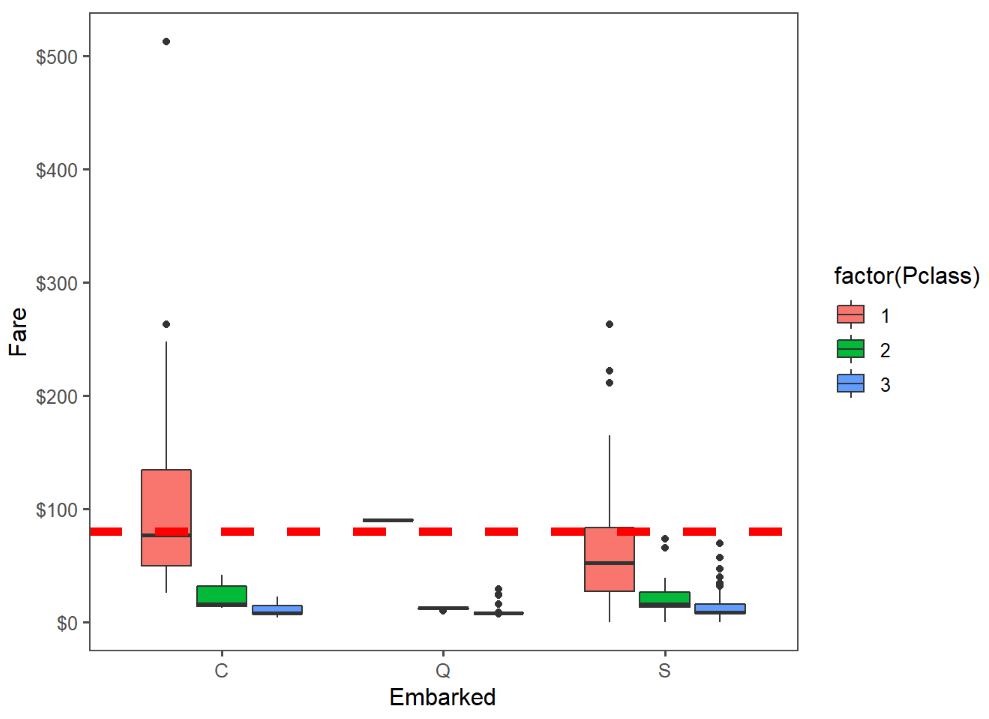
又因為他們的Fare都是$80 且 Pclass都是1,所以編號是62跟830的乘客有較高機率是從C港上船的,所以我們或許可以將他們的上傳資料改為"C"。
# Since their fare was $80 for 1st class, they most likely embarked from 'C'
full$Embarked[c(62, 830)] <- 'C'
接著,我們來看看 Fare 有沒有缺失值。
filter(full,is.na(full$Fare))
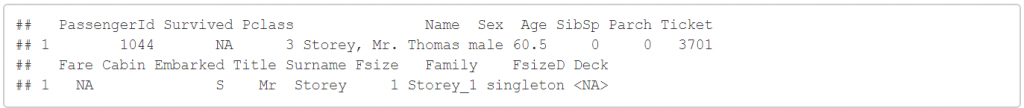
那我們就直接來看看1044筆資料的形式。
full[1044, ]

因為1044筆資料的Pclass=3,且Embarked=S,根據剛剛的圖形,比較安全的設置是把他的票價訂在Pclass=3,且Embarked=S的中位數。再畫一個圖形來確認我們的想法。
ggplot(full[full$Pclass == '3' & full$Embarked == 'S', ],
aes(x = Fare)) +
geom_density(fill = '#99d6ff', alpha=0.4) +
geom_vline(aes(xintercept=median(Fare, na.rm=T)),
colour='red', linetype='dashed', lwd=1) +
scale_x_continuous(labels=dollar_format()) +
theme_few()
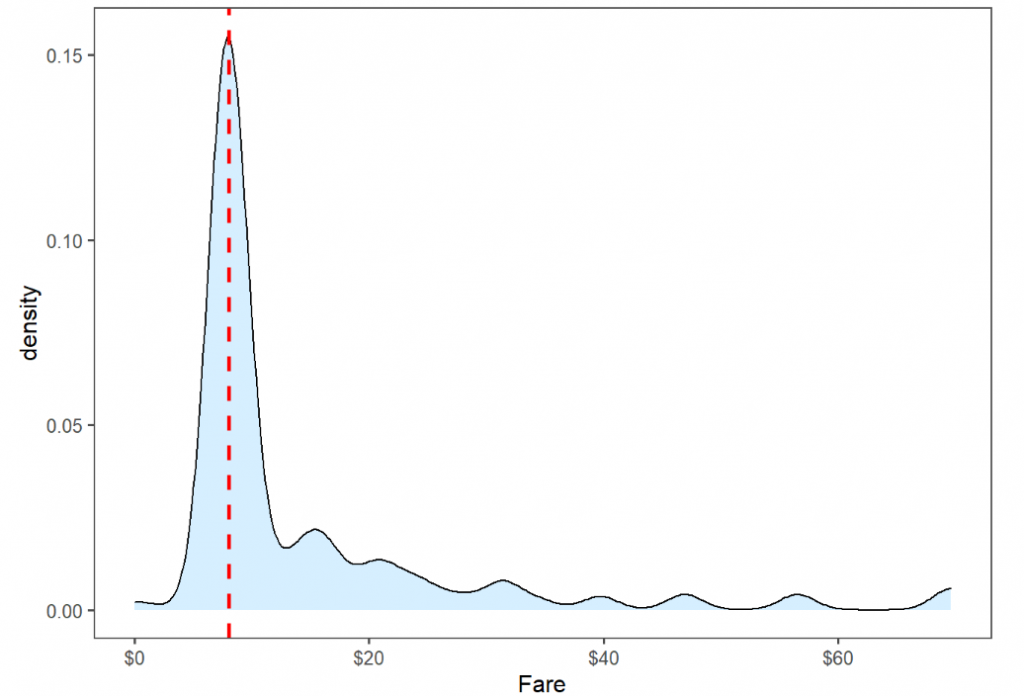
確實的,在Pclass=3且Embarked=S的情況下,在中位數(紅線)附近是比較密集的,所以我們可以較為安心的把1044筆資料的票價設為中位數。
# Replace missing fare value with median fare for class/embarkment
full$Fare[1044] <- median(full[full$Pclass == '3' & full$Embarked == 'S', ]$Fare, na.rm = TRUE)
接著 Megan L. Risdal 做了一個比較特別的預測。
因為Age的缺失值也相當多,不過從這些資料當中,要預測存活率的話,Age的資料似乎是不可刪減的,先來看看到底缺了多少資料吧!
# Show number of missing Age values
sum(is.na(full$Age))

竟然缺了263筆年齡的資料,所以在預測存活率之前,Megan決定要先來預測年齡,這邊他選擇使用 mice package 去做多重預測,在捨棄一些無關變數之後,進行多個變數的預測,如下:
# Make variables factors into factors
factor_vars <- c('PassengerId','Pclass','Sex','Embarked',
'Title','Surname','Family','FsizeD')
full[factor_vars] <- lapply(full[factor_vars], function(x) as.factor(x))
# Set a random seed
set.seed(129)
# Perform mice imputation, excluding certain less-than-useful variables:
mice_mod <- mice(full[, !names(full) %in% c('PassengerId','Name','Ticket','Cabin','Family','Surname','Survived')], method='rf')

# Save the complete output
mice_output <- complete(mice_mod)
預測的結果包含了以上11個變數,不過我們只需要預測出來的Age的部分。
但是會有疑問,這樣的預測真的好嗎?那我們來看看預測前後的分布圖。
# Plot age distributions
par(mfrow=c(1,2))
hist(full$Age, freq=F, main='Age: Original Data',
col='darkgreen', ylim=c(0,0.04))
hist(mice_output$Age, freq=F, main='Age: MICE Output',
col='lightgreen', ylim=c(0,0.04))
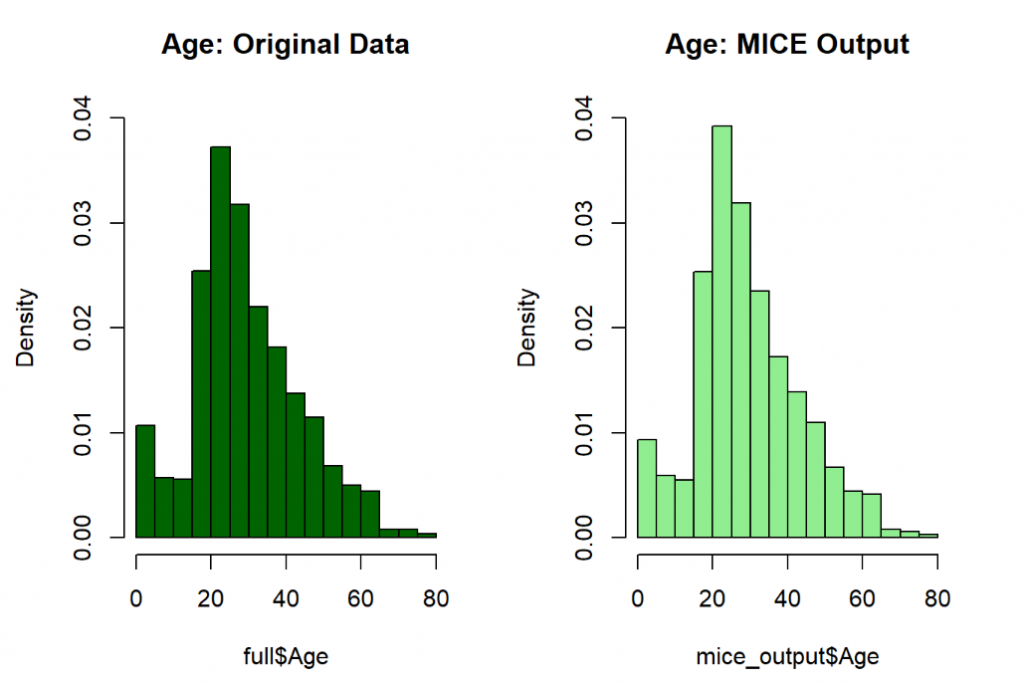
從這兩張圖可以發現,分佈的情況非常相似,所以這似乎是好的預測模型。因此我們就只擷取Age的部分,因為其他部份的預測結果可能很糟,且不是我們想要的。
# Replace Age variable from the mice model.
full$Age <- mice_output$Age
# Show new number of missing Age values
sum(is.na(full$Age))

最後我們就把預測好的 Age 存取致我們原來的資料,今天的缺失值處理就先到這裡,下一篇我章我們會把我們現在的資料再做一次特徵工程,最終再來預測我們要的存活率。今天的文章到這裡,謝謝大家。
原文網址:[https://www.kaggle.com/mrisdal/exploring-survival-on-the-titanic]
