https://yourfreetemplates.com/free-machine-learning-diagram/
專案中,常會遇到 Imbalanced Data 不平衡資料。
如:乳癌患者、恐怖份子查驗、詐欺犯預測...等,我們關注的是"少數"樣本是否能被準確預測?
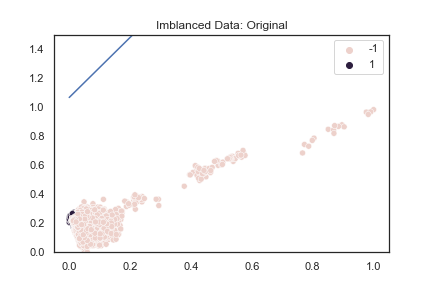
以蛋白質範例,可以發現決策邊界完全無法將少數資料分離。
sns.set(style='white')
ds = fetch_datasets()['protein_homo']
X = PCA(n_components=2).fit_transform(ds.data)
X = MinMaxScaler().fit_transform(X)
y = ds.target
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=42, shuffle=True)
# print('X shape:', X.shape)
# print('y shape:', y.shape)
# print('Positive Ratio:', np.count_nonzero(y==1) / y.shape[0])
lr = LogisticRegression().fit(X_train, y_train)
y_pred = lr.predict(X_test)
# print('Report :', classification_report(y_test, y_pred))
# print('ROC :', roc_auc_score(y_test, y_pred))
plot_x = np.linspace(0, 1, 1000)
plot_y = (-lr.coef_[0][0] * plot_x - lr.intercept_) / lr.coef_[0][1]
# 作圖
sns.scatterplot(X_train[:, 0], X_train[:, 1], hue=y_train)
plt.plot(plot_x, plot_y)
plt.title('Imblanced Data: Original')
plt.ylim(0, 1.5)
plt.show()
一般來說 Accuracy 準確度是一個直覺性高的指標。
但單純的準確度並沒辦法精準衡量模型是好是壞,因此這裡介紹幾種更常見的評估方式:
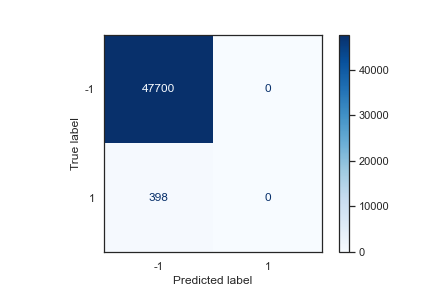
下圖為上述資料的混淆矩陣,可發現準確率達到 47700 / (47700+398) = 99.2%
from sklearn.metrics import plot_confusion_matrix
cm = plot_confusion_matrix(
lr,
X_test, y_test,
cmap=plt.cm.Blues
)
plt.show()
甚麼是精確率?
Precision 精確率:被"預測正確樣本"中,是"實際正確樣本"有多少比例。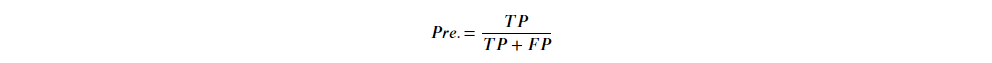
Recall 召回率:"實際正確樣本"中,被"預測正確樣本"有多少比例。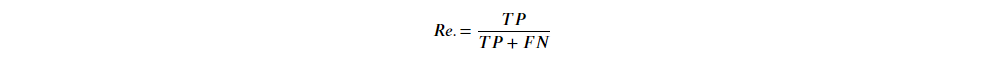
有了精確率與召回率,統計學家進一步定義了:
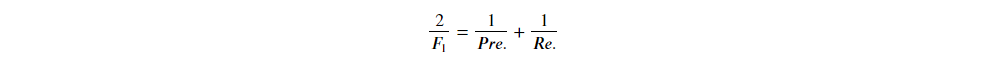
化簡後可得: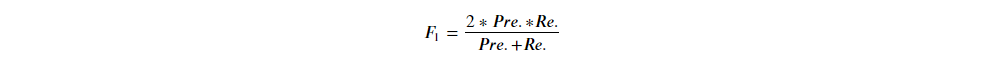
也有學者提出精確率 & 召回率不同權重的算法: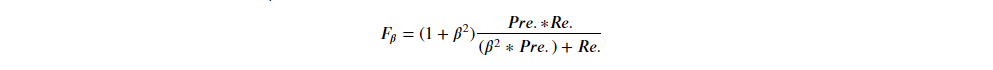
全名 Receiver Operating Characteristic,而曲線下面積稱 Area Under Curve (AUC)。
首先定義: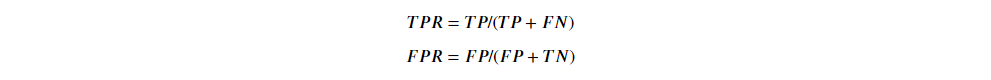
有了這兩個指標,再配合模型的"閾值",我們可以畫出一條 ROC 曲線。
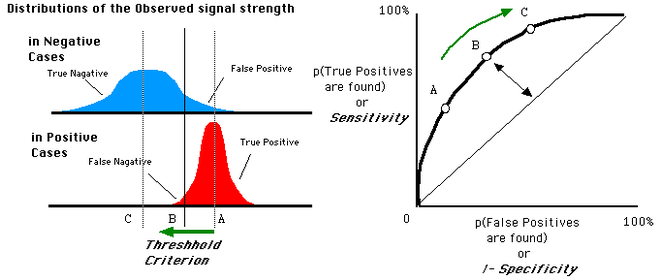
圖左:
藍色為負樣本,紅色為正樣本,橫軸則是模型預測的機率。
很直覺的理解:正樣本集中於預測機率高的分段上,負樣本則較低。
此時我們可以設一個"閾值"(通常預設 0.5),以上判定為正,以下判定為負。
圖右:
將橫軸設為 FPR,縱軸設為 TPR,配上不同閾值可畫出一條曲線。
若選 A 點作閾值,則大部分負樣本都被剔除,但正樣本也留下較少(TPR/FPR 皆下降)。
約有一半的正樣本被判定為正,TPR ≈ 0.5 ,少部分負樣本被判定為正,FPR ≈ 0.2 ,
最終得到 A 點在右圖曲線上的位置。

蛋白質範例的 ROC(AUC):
from sklearn.metrics import roc_curve, roc_auc_score
fpr, tpr, threshold = roc_curve(y_test, y_pred)
auc = roc_auc_score(y_test, y_pred)
plt.title('Receiver Operating Characteristic')
plt.plot(fpr, tpr, c='b', label=f'AUC = {auc:0.2f}')
plt.plot([0, 1], [0, 1], 'r--')
plt.ylabel('True Positive Rate')
plt.xlabel('False Positive Rate')
plt.legend(loc='best')
plt.show()
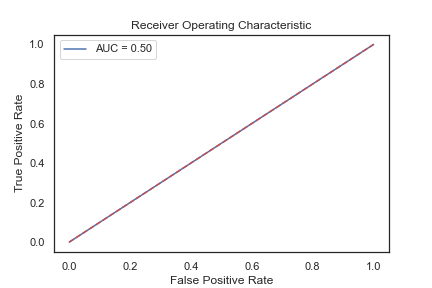
但問題仍沒解決:樣本比例差異過大的情況下,總會使訓練的模型判斷能力差。
因此,接下來我們會嘗試透過採樣技術來克服它。
將少數樣本用某種方式重複抽樣或合成新樣本,稱過採樣。
相反,將多數樣本中較不具代表性的移除以免造成雜訊,稱欠採樣。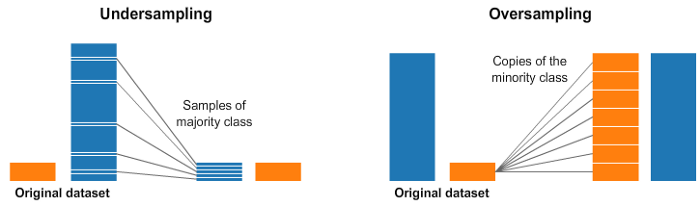
全名 Synthetic Minority Oversampling Technique 合成少數過採樣技術。
概念是在少數樣本位置近的地方,人工合成一些樣本。
A. 挑一個少數派(紅點),並將鄰近的 k 個(k=3)點找出。(Pic1)
B. 從 k 個近鄰點中隨機選取一個,透過公式合成 N 個(N=3)樣本點。(Pic2)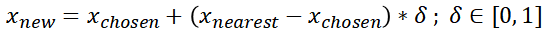
C. 接著對所有的少數點做同樣的操作。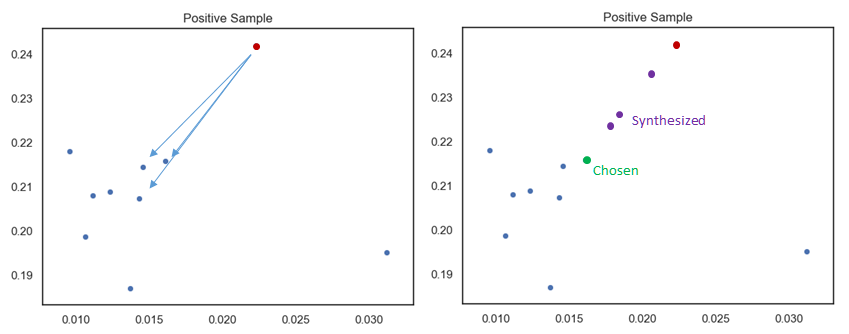
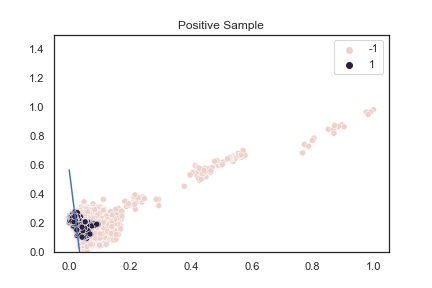
蛋白質範例操作 SMOTE:
from imblearn.over_sampling import SMOTE
from sklearn.metrics import roc_auc_score, classification_report
X_re, y_re = SMOTE(random_state=42).fit_resample(X_train, y_train)
lr = LogisticRegression().fit(X_re, y_re)
y_pred = lr.predict(X_test)
plot_x = np.linspace(0, 1, 1000)
plot_y = (-lr.coef_[0][0] * plot_base - lr.intercept_) / lr.coef_[0][1]
# 作圖
sns.scatterplot(X_re[:, 0], X_re[:, 1], hue=y_re)
plt.plot(plot_x, plot_y)
plt.title('Positive Sample')
plt.ylim(0, 1.5)
plt.show()
結合剛剛的 ROC(AUC)曲線:
from sklearn.metrics import roc_curve, roc_auc_score
fpr, tpr, threshold = roc_curve(y_test, y_pred)
auc = roc_auc_score(y_test, y_pred)
plt.title('Receiver Operating Characteristic')
plt.plot(fpr, tpr, c='b', label=f'AUC = {auc:0.2f}')
plt.plot([0, 1], [0, 1], 'r--')
plt.ylabel('True Positive Rate')
plt.xlabel('False Positive Rate')
plt.legend(loc='best')
plt.show()
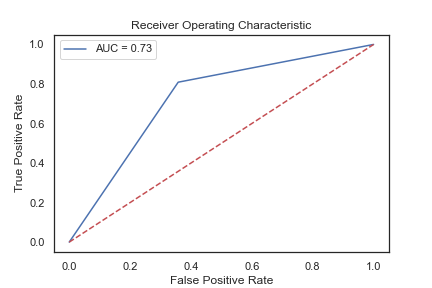
print(auc)
>> 0.7254869736523286
SMOTE 雖不錯,但有一個明顯缺點:對"所有少數樣本"都做過採樣。
大多時候並不是所有少數樣本都無鑑別度,真正無鑑別度的是與多數樣本混合在一起的少數樣本。
靠近邊界的少數樣本因與多數樣本混合在一起,易產生雜訊。若對邊界樣本學習,可能將多數樣本誤判為少數。
因此,對 SMOTE 算法做出改進的算法,即 SMOTE Border Line。
from imblearn.over_sampling import BorderlineSMOTE
blsmote = BorderlineSMOTE(random_state=42, kind=’borderline-2')
X_re, y_re = blsmote.fit_resample(X_train, y_train)
實作上,其實還是更常使用 SMOTE,畢竟 Borderline 方法的計算複雜,且閾值設定也缺乏公定的標準。
需要花更多時間調參,然而跑分進步幅度卻不大。
相對過採樣,欠採樣是將多數樣本進行 Scale Down,使模型的權重改變,少考慮一些多數樣本。
最簡單的做法是隨機排除掉一些多數樣本。但有可能排除掉邊界樣本,
使沒鑑別度少數樣本也被模型考慮,雖使鑑別度上升,卻增加過擬合風險。
會針對所有樣本去遍歷一次。
令兩個樣本點 x, y 分屬不同的 class,一個為多數樣本,另一為少數,可計算樣本間距 d(x, y)。
若找不到第三個樣本點 z,使得任一樣本點到 z 的距離比樣本點間距還小,則刪去其。
核心理念:找出邊界鑑別度不高的樣本,認為這些樣本屬雜訊應該剔除(類似 Borderline SMOTE)。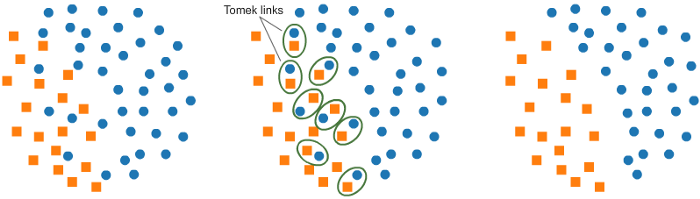
蛋白質範例操作 SMOTE + TomekLinks:
X_re, y_re = SMOTE(random_state=42).fit_resample(X_train, y_train)
X_rere, y_rere = TomekLinks().fit_resample(X_re, y_re)
lr = LogisticRegression().fit(X_rere, y_rere)
y_pred = lr.predict(X_test)
from sklearn.metrics import roc_curve, roc_auc_score
fpr, tpr, threshold = roc_curve(y_test, y_pred)
auc = roc_auc_score(y_test, y_pred)
print(auc)
>> 0.7332799742949548
與 Tomek Links 觀念相同,也是透過某種方式來剔除鑑別度低的樣本。
ENN 改成對多數樣本尋找 K 個近鄰點,若一半以上(門檻可自設)不屬於多數樣本,就將該樣本剔除。
實作上,其實很常同時使用過採樣 + 欠採樣來做資料重組。如下圖: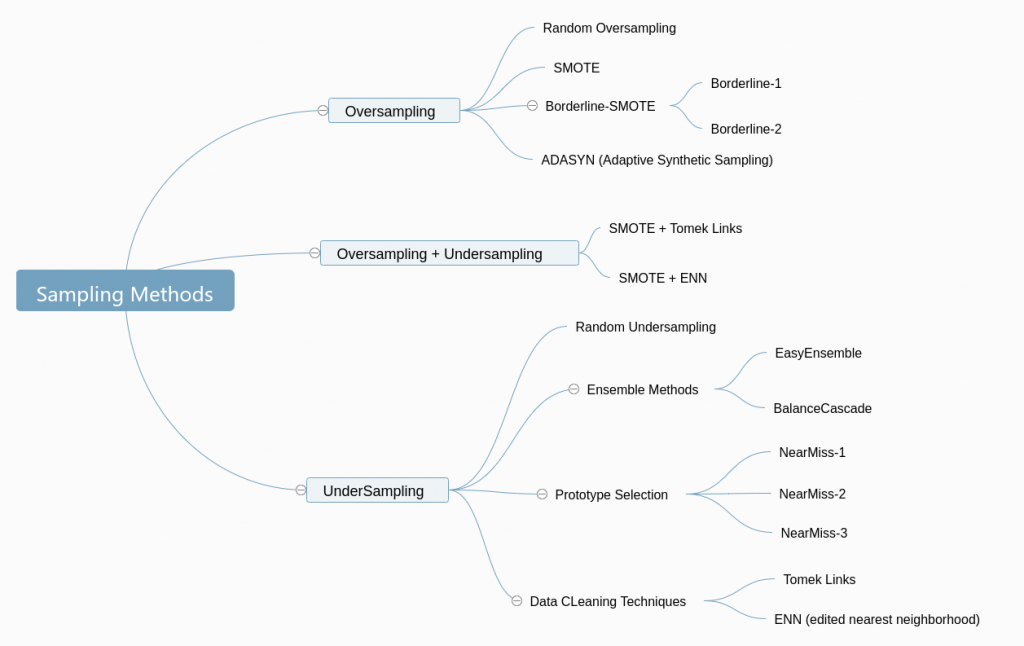
重新採樣的目的是讓模型產生鑑別度,而不是讓模型學習錯誤資訊。若先採樣才切分,可能使測試資料偏離了原資料,導致模型學習到一堆雜訊。
不管哪種採樣,都會大幅增加過擬合程度(如:樣本數少,又做欠採樣)。
即使模型區分出來,由於欠採樣後多數樣本過少,導致模型只側重學習某部分樣本,無法反映資料全貌。
此時,交叉驗證、建立多模型做集成學習,都會是好的解決方式。
蛋白質範例是因為少數樣本與多數樣本看上去還能分離,實際運行很有可能碰到完全分不開的例子。
若少數樣本雜亂地散落在多數樣本之間,此時就不要考慮採樣問題。
可以優先評估是否資料本身的分佈有問題,像是一開始回收數據錯誤,或樣本並非歐幾里得分布等情況。
使用內建 wine,試著用 pipeline、Cross Validation,寫個迴圈以操作演示過的演算法。
import numpy as np
import pandas as pd
from sklearn.datasets import load_wine
from sklearn.model_selection import train_test_split
ds = load_wine()
X = pd.DataFrame(ds.data, columns=ds.feature_names)
y = pd.DataFrame(ds.target, columns=['Wine'])
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=0)
# 把要用的 model 整理出
from sklearn.linear_model import LogisticRegression
from sklearn.naive_bayes import GaussianNB
from sklearn.neighbors import KNeighborsClassifier
from sklearn.tree import DecisionTreeClassifier
from sklearn.svm import SVC
from sklearn.neural_network import MLPClassifier
from sklearn.ensemble import RandomForestClassifier
models = []
models.append(("Logistic Regression", LogisticRegression()))
models.append(("Naive Bayes", GaussianNB()))
models.append(("K-Nearest Neighbour", KNeighborsClassifier(n_neighbors=3)))
models.append(("Decision Tree", DecisionTreeClassifier()))
models.append(("Support Vector Machine-linear", SVC(kernel="linear")))
models.append(("Support Vector Machine-rbf", SVC(kernel="rbf")))
models.append(("Random Forest", RandomForestClassifier(n_estimators=7)))
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import StratifiedKFold
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
scores = []
names = []
for name, model in models:
kfold = StratifiedKFold(n_splits=10).split(X_train, y_train)
rfc_PL = make_pipeline(
StandardScaler(),
PCA(n_components=2),
model
)
cv = cross_val_score(rfc_PL, X_train, y_train, cv=kfold, scoring = "accuracy")
names.append(name)
scores.append(cv)
for i in range(len(names)):
print(f'{names[i]:<30}: {scores[i].mean()*100:.3f}')
>> Logistic Regression : 96.090
Naive Bayes : 96.090
K-Nearest Neighbour : 92.949
Decision Tree : 94.551
Support Vector Machine-linear : 95.321
Support Vector Machine-rbf : 96.090
Random Forest : 96.090
