本文重點:細針活體切片、乳房腫瘤、Sklearn模型、預測
完整代碼+csv+model 在GitHub
(一)、認識資料集:
Breast cancer Datasets有多個版本,我們先來認識一下資料集內容是什麼。
安裝完Sklearn后,隨附的Breast cancer.csv 有569筆資料,30個特徵欄位,1個診斷結果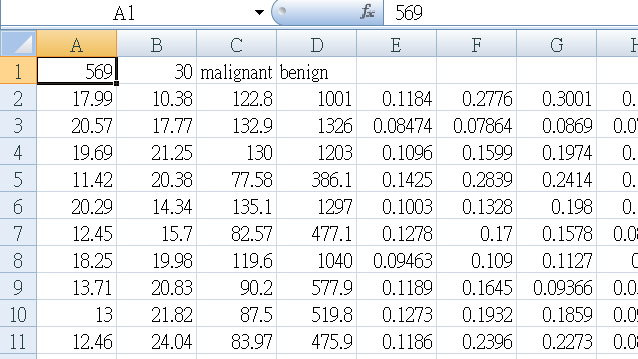
今天我們取用的是另外一個版本,Kaggle dataset,這是已經標上欄位名稱的CSV檔,特徵資料內容看起來和上一個檔是一樣的,也是569筆。
第一欄位增加了”id”,第二欄是target ”診斷”是文字(M 代表惡性 Malignant ,B代表良性Benign) ,其餘30個欄位是”特徵數據”。
“特徵”所代表的意義:UCI machine learning center 有解說 link here
這些資料是從乳房腫瘤細針穿刺(Fine needle aspiration)樣本取得的,特徵欄位所記錄的是細胞核的多項數據資料:radius、texture、perimeter、area…等等。 資料集是這樣子的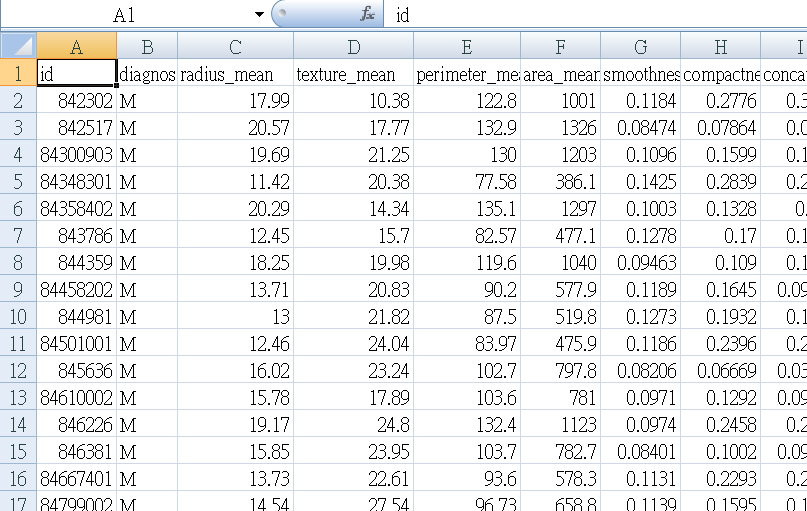
Kaggle討論區有人提到說,Cancer其實就是”惡性”的專有名稱,並不包含”良性”,不應該把資料集名稱叫xxCancer,因此,我們也從善如流,將資料集改名為 BStumorKaggle.csv
Tumor腫瘤(或腫塊)包含了”良性”與”惡性”。
(二)、本文目的: 訓練-->建立模型-->預測
我們今天的重點在於建模、預測,而在Sklearn、Kaggle有多個範例是使用Seaborne畫出華麗的圖(violin、box、heatmap、swarm…),這些畫圖的部份,我們就略過不提了,以免失焦、糢糊了重點。
有興趣者請參考此連結
(三)、建立模型
Step 1.以dataframe 讀取csv 上一篇 以ndarray讀取CSV的方式
Step 2.資料前處理
Step 3.Split data
Step 4.Build the Model
Step 5.評估模型準確度
Step 6.儲存模型
以下直接貼代碼,各段說明在代碼內
# BStumor_detect.py 2021/9/5 neoCaffe
import numpy as np
import pandas as pd
from sklearn.preprocessing import StandardScaler
from sklearn.model_selection import train_test_split
''' Step 1. 以dataframe 讀取csv '''
print('以dataframe 讀取 csv')
df = pd.read_csv('BStumorKaggle.csv')
print(f'df.shape : {df.shape}')
print(f'欄位: {df.columns}')
print(f'前5筆記錄: {df.head()}')
# 節省篇幅,也因為欄位內容都沒有缺損,所以這裡沒有寫nan之處理
''' Step 2. 資料前處理 '''
# 設定 X ,y
y = df['diagnosis'] # y 診斷結果=diagnosis 欄位
''' 去除 id diagnosis 兩個分類欄位,剩下特徵欄位-->X '''
df2 = df.drop(['id', 'diagnosis'], axis = 1)
print(df2.shape)
X = df2
print(X.head()) # X is a dataframe
print(y[:5]) # y is a Series
''' Step 3. Split data 分成 training 、testing 兩組 '''
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.2, random_state = 42)
''' Step 4. Build the Model : training'''
# model logistic regression
from sklearn.linear_model import LogisticRegression
logis = LogisticRegression(solver='lbfgs',max_iter=400)
logis.fit(X_train, y_train)
# model random forest
from sklearn.ensemble import RandomForestClassifier
fores = RandomForestClassifier()
fores.fit(X_train, y_train)
''' 測試 抓一筆資料,試試看。此筆資料的 diagnosis 是 M '''
newX =[[13.73,22.61,93.6,578.3,0.1131,0.2293,0.2128,0.08025,
0.2069,0.07682,0.2121,1.169,2.061,19.21,0.006429,0.05936,
0.05501,0.01628,0.01961,0.008093,15.03,32.01,108.8,697.7,
0.1651,0.7725,0.6943,0.2208,0.3596,0.1431]]
newP_logis = logis.predict(newX) # 預測值
newP_fores = fores.predict(newX)
print(f'真正的診斷: M LogisticRegression 預測診斷: {newP_logis}')
print(f'真正的診斷: M RandomForest 預測診斷: {newP_fores}')
''' Step 5. 評估模型準確度 兩種model的accuracy '''
from sklearn.metrics import accuracy_score
pred_logis = logis.predict(X_test)
pred_fores = fores.predict(X_test)
a1 = accuracy_score(pred_logis, y_test)
a2 = accuracy_score(pred_fores, y_test)
print(f'兩種模型的準確率 accuracy: logis: {a1} fores: {a2}')
''' Step 6. 儲存模型 '''
import pickle
mdl_BSlr = 'BSlr.model'
pickle.dump(logis, open(mdl_BSlr, 'wb'))
mdl_BSfs = 'BSfs.model'
pickle.dump(fores, open(mdl_BSfs, 'wb'))
print('models saved ')
# --- 建模完成 ---
(四)、使用模型做預測
上面程式,將兩個模型存檔成 BSlr.model BSfs.model
另寫個py 使用這兩個模型 代碼如下
test1 test2 是從csv檔摘出來的兩筆特徵資料,當測試。
''' 以兩種模型進行預測 predict with loaded model '''
mdl_logis = pickle.load(open('BSlr.model', 'rb'))
predict1 = mdl_logis.predict(test1)
mdl_fores = pickle.load(open('BSfs.model', 'rb'))
predict2 = mdl_fores.predict(test2)
print(f'test1 真正診斷是: B 以logis模型預測是: {predict1}')
print(f'test2 真正診斷是: M 以fores模型預測是: {predict2}')
