題目連結:https://rosalind.info/problems/revc/
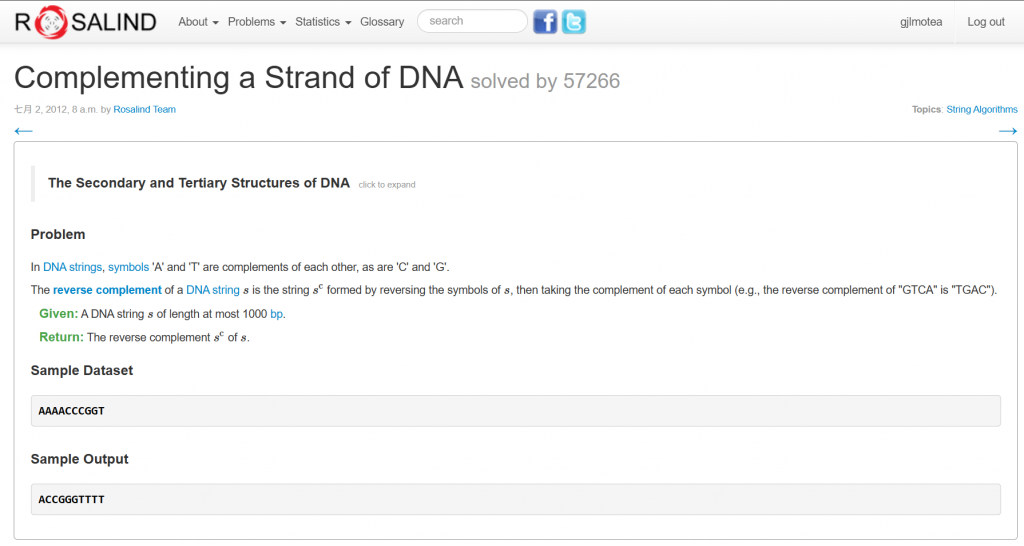
DNA 的每一條鏈都有一個方向:5' → 3'(由糖-磷酸骨架的 5' 碳到 3' 碳)
在雙股 DNA 中,兩條鏈是反向平行 (antiparallel) 的:
一條是 5' → 3'
一條是 3' → 5'
但是我們閱讀的方向,都是從5' → 3'
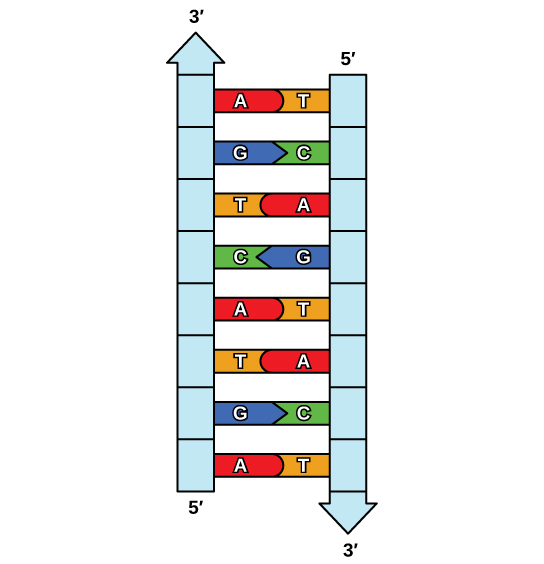
這一題要計算DNA字串的反向互補(reverse complement)
輸入
AAAACCCGGT
輸出
ACCGGGTTTT
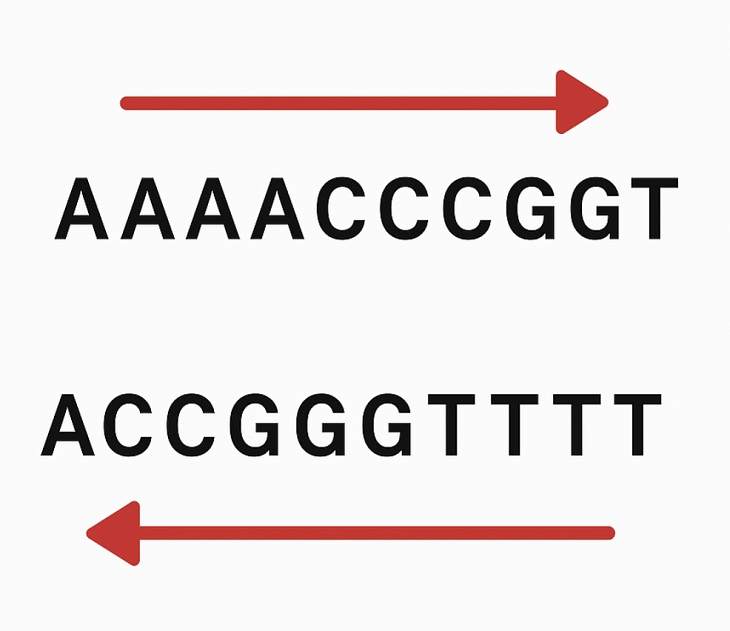
此題有兩個核心要點
1:反向。序列方向相反(首尾顛倒)
2:互補。DNA是雙股螺旋,四種鹼基互補規則為:A配T、C配G
所以給一段序列 AAAACCCGGT
需要輸出 ACCGGGTTTT
直觀說明
兩端3'->5'、5'->3'互補,就有點像我們的書本:
中文字都是由左向右排列,人類閱讀是從左邊讀到右邊

但是書本闔上、閉起來的時候,兩者就頭尾重疊了:
左邊的頭對到右邊尾、右邊頭對到左邊尾

不會都從閉闔的書本同一側讀進去、或者讀出來

(除非你的眼睛特別牛B)

所以我們朗讀的時候,都是從頭部(5')讀到尾部(3')
照固定的順序來
程式碼:
s = "AAAACCCGGT"
rev_s = s[::-1]
# 或者
# rev_s = ''.join(reversed(s))
for base in rev_s:
match base:
case 'A':
print('T', end='')
case 'C':
print('G', end='')
case 'G':
print('C', end='')
case 'T':
print('A', end='')
或者用dict存table配對
def reverse_complement(dna):
table = {'A': 'T', 'T': 'A', 'C': 'G', 'G': 'C'}
result = str()
for base in reversed(dna):
result += table[base]
return result
s = "AAAACCCGGT"
print(reverse_complement(s))
或者沿用前一篇maketrans()
def reverse_complement(s):
# 先互補(替換),再反轉
trans_table = str.maketrans('ATCG', 'TAGC')
rev_s = s.translate(trans_table)
return rev_s[::-1]
s = "AAAACCCGGT"
print(reverse_complement(s))
