題目連結:https://rosalind.info/problems/gc/
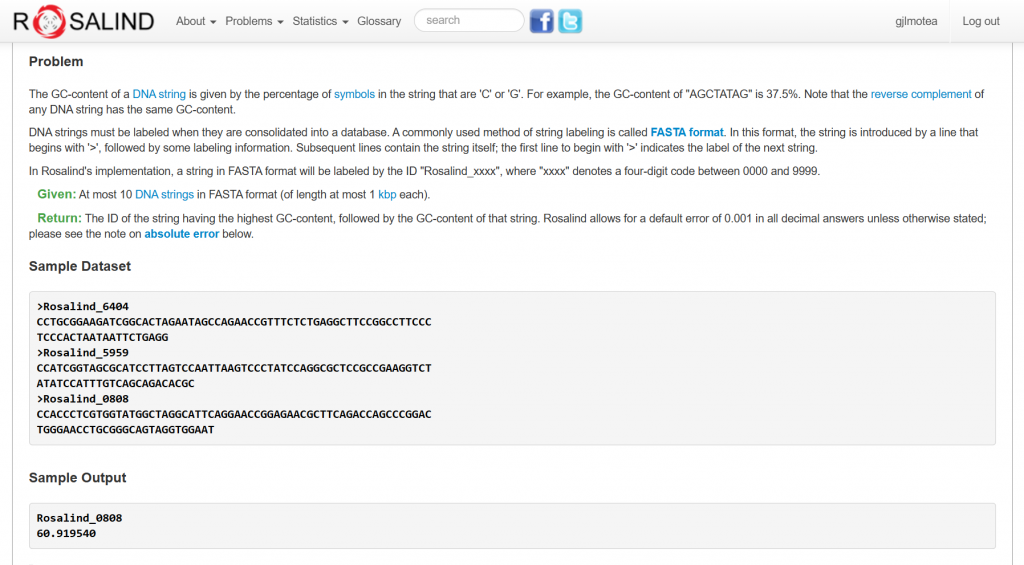
這題要我們讀入多個DNA序列(FASTA格式)
從中找出GC含量最高的那一段序列,並輸出GC含量(保留小數到第六位)
普遍稱呼「GC」而非「CG」,我也不知道為什麽 ╮(╯_╰)╭
但讓我聯想到,GC在程式語言專指垃圾回收(Garbage Collection)
首先要了解FASTA格式(又稱Pearson格式)
這是生物資訊中最常見的一種序列資料的儲存格式
FASTA格式非常松散,它的規則非常簡單:
> 開頭,表示一段新序列的標籤(描述行)>我的序列01
AAAAAAAA
>我的序列02
CCCCCCCC
標籤(描述行)又有很多種說法:
ID/Description/Header/Name/Title/Label Line(也太松散了吧!)
通常以 ID/Description/Header 為最佳實踐
資料本體(序列內容)又稱爲 :Sequence/Sequence Lines
輸入
>Rosalind_6404
CCTGCGGAAGATCGGCACTAGAATAGCCAGAACCGTTTCTCTGAGGCTTCCGGCCTTCCC
TCCCACTAATAATTCTGAGG
>Rosalind_5959
CCATCGGTAGCGCATCCTTAGTCCAATTAAGTCCCTATCCAGGCGCTCCGCCGAAGGTCT
ATATCCATTTGTCAGCAGACACGC
>Rosalind_0808
CCACCCTCGTGGTATGGCTAGGCATTCAGGAACCGGAGAACGCTTCAGACCAGCCCGGAC
TGGGAACCTGCGGGCAGTAGGTGGAAT
輸出
Rosalind_0808
60.919540
程式碼
data = \
""">Rosalind_6404
CCTGCGGAAGATCGGCACTAGAATAGCCAGAACCGTTTCTCTGAGGCTTCCGGCCTTCCC
TCCCACTAATAATTCTGAGG
>Rosalind_5959
CCATCGGTAGCGCATCCTTAGTCCAATTAAGTCCCTATCCAGGCGCTCCGCCGAAGGTCT
ATATCCATTTGTCAGCAGACACGC
>Rosalind_0808
CCACCCTCGTGGTATGGCTAGGCATTCAGGAACCGGAGAACGCTTCAGACCAGCCCGGAC
TGGGAACCTGCGGGCAGTAGGTGGAAT
"""
# 讀入資料,將sequence存入dict
sequences = dict()
label = ""
for line in data.splitlines(): # 等同做了 str.split("\n"), \r, \n\r...
if line[0] == ">":
label = line[1:] # 去除">"
sequences[label] = ""
else:
sequences[label] += line
# 針對每一個序列進行GC計算
max_gc_label, max_gc_ratio = "", 0
for label, seq in sequences.items():
at_count = 0
gc_count = 0
for base in seq:
if base == "A" or base == "T":
at_count += 1
if base == "C" or base == "G":
gc_count += 1
gc_ratio = gc_count / (at_count + gc_count) * 100
if gc_ratio > max_gc_ratio:
max_gc_label, max_gc_ratio = label, gc_ratio
print(max_gc_label)
print(f"{max_gc_ratio:.6f}")
對於變數名稱感到糾結 >< 很想維持一致性
到底要 cg 還 gc
因為一下子 at_count、另一邊 gc_count 又怪怪的,沒有照字母順序排
最後還是統一改成 gc_count、gc_ratio
要“保留小數到第六位”,如果直接用Python來做很省事
因為Python預設float就是double精度,處理格式化輸出也非常容易
其他程式語言就不一定了
另外,因平台每次出題都會有變化,只能知道正確/錯誤,沒有除錯機會
可先用此連結(Endmemo)進行驗算
BTW,在生物意義上
GC含量可作為基因體比較、演化分析的參考指標之一
因為不同種類的物種,通常擁有不同比例的「GC含量」
(當然,只是大約數字、並非精確,畢竟每個物種都存在個體差異以及突變)
人與人之間的DNA相似度為99.9%
黑猩猩與黑猩猩之間的DNA相似度為99.7~99.9%
在植物界,同一種植物之間,DNA相似度也都在98%以上
雖然不能只透過GC含量精準判斷物種,但可以大幅縮小範圍
題外話
我有一個自然而然的疑惑是
既然知其一就能推得另一個,那為何大家都是說「GC」而非「AT」?
=> 原因是CG的結構更穩定、不易變性,更能提供演化的線索
所以主要就是觀察「GC含量」
CG穩定的原因:C≡G(三個氫鍵)、A=T(兩個氫鍵)
如果你和我一樣有強迫癥 (工匠精神),可以繼續往下閱讀
在讀入序列時,有幾個坑要注意
要預設「使用者可能輸入任何格式」,有空白、特殊字元、不規則的換行符號等情況。
一個好的程式寫法都要可以支援
Description Line 只能有一行。中間要不要空格都可以,能不能有特殊字元都可以。
Sequence Lines 與下一個 Description Line之間要不要換行(回車)都可以。
若使用者不小心將資料縮排,也要有防呆功能。

ex: 地獄級測資
>我的序列01
AAAAA
A
A
A
> 我的序列02
CCCC
CCCC
> 我的序列 03
GGGGGGGG
> 神的序列>>>
ATCGATCG
>鬼的序列
>>>
>不小心縮排
TTTT
>
ATGC
(空白標籤不合法,捨棄這段序列)
最終要讀成
"我的序列01"=AAAAAAAA
"我的序列02"=CCCCCCCC
"我的序列 03"=GGGGGGGG
"神的序列>>>"=ATCGATCG
"鬼的序列"=(空)
">>"=(空)
"不小心縮排"=TTTT
健壯的parse程式碼(地獄測資):
def parse_fasta(data: str) -> dict:
sequences = {}
label = ""
for line in data.strip().splitlines():
line = line.strip() # 縮排防呆
if not line: # 跳過空行
continue
if line.startswith(">"):
label = line[1:].strip() # 去除label頭尾空白
if not label: # 跳過空標籤(不合法)
label = ""
continue
sequences[label] = ""
elif label:
sequences[label] += line
return sequences
data = \
"""
>我的序列01
AAAAA
A
A
A
> 我的序列02
CCCC
CCCC
> 我的序列 03
GGGGGGGG
> 神的序列>>>
ATCGATCG
>鬼的序列
>>>
>不小心縮排
TTTT
>
ATGC
(空白標籤不合法,應捨棄這段序列)
"""
seqs = parse_fasta(data)
print(seqs)
完整程式碼(改寫成function)
def parse_fasta(data: str) -> dict:
sequences = {}
label = ""
for line in data.strip().splitlines():
line = line.strip() # 縮排防呆
if not line: # 跳過空行
continue
if line.startswith(">"):
label = line[1:].strip() # 去除label頭尾空白
if not label: # 跳過空標籤(不合法)
label = ""
continue
sequences[label] = ""
elif label:
sequences[label] += line
return sequences
def get_gc_content(seq):
total = seq.count("A") + seq.count("C") + seq.count("G") + seq.count("T")
if total == 0:
return 0
gc_content = (seq.count("G") + seq.count("C")) / total * 100
return gc_content
def find_highest_gc(fasta_str):
sequences = parse_fasta(fasta_str)
max_gc_label, max_gc_ratio = "", 0
for label, sequence in sequences.items():
gc_content = get_gc_content(sequence)
if gc_content > max_gc_ratio:
max_gc_label, max_gc_ratio = label, gc_content
return max_gc_label, max_gc_ratio
data = """
>Rosalind_6404
CCTGCGGAAGATCGGCACTAGAATAGCCAGAACCGTTTCTCTGAGGCTTCCGGCCTTCCC
TCCCACTAATAATTCTGAGG
>Rosalind_5959
CCATCGGTAGCGCATCCTTAGTCCAATTAAGTCCCTATCCAGGCGCTCCGCCGAAGGTCT
ATATCCATTTGTCAGCAGACACGC
>Rosalind_0808
CCACCCTCGTGGTATGGCTAGGCATTCAGGAACCGGAGAACGCTTCAGACCAGCCCGGAC
TGGGAACCTGCGGGCAGTAGGTGGAAT
"""
label, gc_ratio = find_highest_gc(data)
print(label)
print(f"{gc_ratio:.6f}")
