生物資訊研究中,研究者時常需要自己編寫客製化的程式碼腳本。不論是視覺化或是統計分析的功能,通常都是引入前人寫好的套件 (package) 中的函式庫來達成,如先前介紹的 biopython or seaborn 都是套件。從另一個角度來看,一個腳本要成功執行的話,它的執行環境需要安裝好那些需要使用到的套件,以及特定的套件版本,這就叫做一個腳本或一個軟體的 ‘dependencies’。問題就出在每個使用者電腦中已經安裝的套件不同,而導致相同的腳本無法在不同的人手中成功執行。
成熟的套件或軟體都會註明需要哪些 dependencies,比如說要先在系統中安裝好哪些其它套件、套件版本、甚至是作業系統等等。但是畢竟所有的現成的套件都是人寫的,有時候工程師為了讓東西運作起來,可能會使用各種不同的奇技淫巧而有意想不到的漏洞,因此即使都按照使用說明安裝 dependencies 了還是會無法成功執行。這樣的情況在生物資訊、計算生物學領域中有多常見呢?
UCLA 的研究團隊搜集了 2005-2017 年間的發表的 36,702 個 omics 軟體,其中 28% 已經失聯找不到下載方式。他們挑選了 98 個軟體來測試安裝,有 49% 並不容易安裝,其中 28% 因為內部程式碼撰寫方式的關係根本無法安裝。另外值得一提的是,那些好安裝的軟體之發表文章的引用情形比不好安裝的軟體更常被引用。
確保研究的可再現性,不只是根本上對學術的尊重,更有可能直接影響到研究的影響力。我個人碩班期間想要嘗試比較不同的轉錄體新組裝軟體,相關研究顯示 Bridger 的組裝結果比引用數最高的 Trinity 更好,花了一整個月嘗試安裝 Bridger 卻一直缺少一個 C 語言套件,相較之下 Trinity 一下子就安裝成功了,我也放棄去追求可能更好的新組裝軟體。
對應用端的生物資訊研究者來說,還不需要去擔心軟體開發之後要給其他人使用的問題,只需要確保自己寫的腳本換台電腦也可以執行就好。除了使用 Anaconda 設定虛擬環境以外,現在還流行用 docker 來把所有執行的環境放進容器中,但是這兩種方式的技術門檻稍微高一點點。NextJournal 提供的則是可以線上執行的程式筆記本,就像是 google colab 一樣,使用體驗類似 Jupyter 與 Notion 筆記軟體的結合。
NextJournal 是來自柏林的團隊所開發,該團隊靠著一款持續營運十年的卡牌遊戲的資金來啟動 NextJournal 計畫,因此可以自主由衷地決定項目的走向,而不用為了錢而放棄自由...阿不是...我是說為了盈利而收費使經費拮据的學術機構難以入手使用他們的服務。一般使用者都可以免費使用NextJournal 上的運算資源,包括 4 central processing units (CPUs), 1 graphical processing unit (GPU), 15 GB memory 和 500 GB of storage 的資源,比一般的小筆電還好了,對於簡單的生物資訊後期分析及視覺化綽綽有餘。
馬上使用吧!
註冊與登錄過程不需要準備密碼
只要提供 email 之後在 email 中點選連結就可以登錄

個人頁面主要控制板就是各個程式筆記本的縮圖,底下也可以看官方的大量使用範例,並且可以 remix 直接由別人的筆記本改造為自己所用,減少從頭寫作的心理負擔
以下連結是我簡單嘗試使用 SRA Toolkit 的示範頁面
https://nextjournal.com/a/La9B7RSN6KjVg7yvc6qJc?token=5HctkYMkZnd4aMnwriKqTA
頁面中也包括了如何在該環境中安裝 SRA Toolkit 的 Ruby dependencies 的指令,但是其實 NextJournal 不大適合這樣的工作。其提供的虛擬主機每隔一天左右就會還原成初始狀態,雖然執行過程與系統的輸出結果會保留下來以純文字模式在筆記本中給所有人看,但是過程中儲存成在系統中的檔案會消失,使用上這點可能要稍微注意。筆記本也可以開啟編輯權限給同事一起編輯,或是僅開啟閱讀權限供讀者瀏覽。
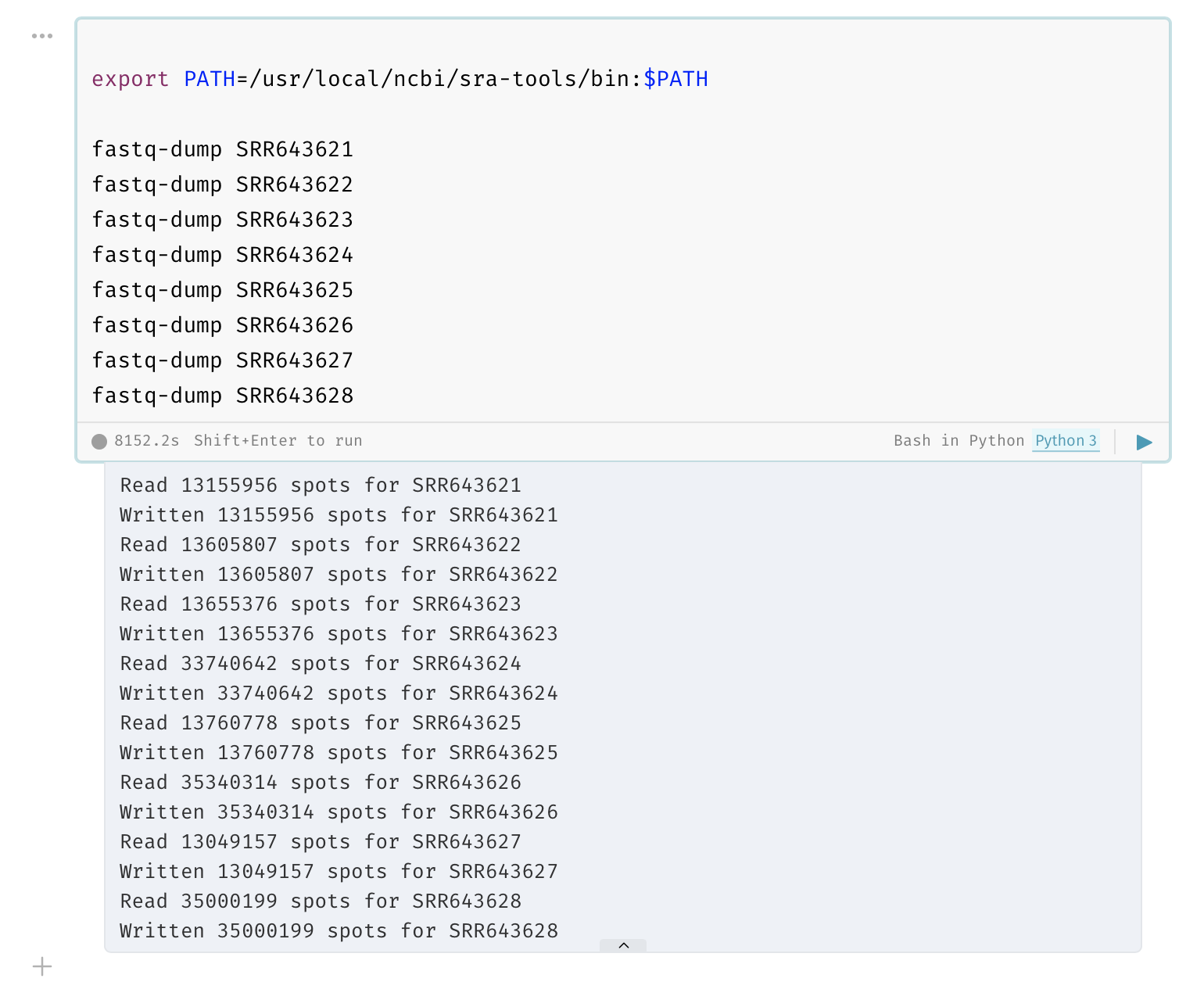
總地來說,NextJournal 實在是非常深得我心,很期待未來他們的發展~希望可以更廣泛地出現在各種學術期刊的發表中,促進整體學術圈可再現性的精神。如果對相關工具有什麼創意的使用方式,或是想了解的地方請留言告訴我~
A “petting zoo for code” makes studies easier to reproduce
關於作者
謝晨 (Chen Hsieh),臺大園藝暨景觀學系研究所碩士。讀碩士前的興趣是懷著寫點程式妄圖解決農業問題的夢想參加比賽,拿了幾個黑客松與 Open Data 創新應用競賽的獎,卻都沒有勇氣將項目經營下去;研究所期間的興趣轉換成讀學術期刊的出刊電子報。靠著這些興趣當選 107 學年的臺大優秀青年,畢業後卻成了無業的實驗室居民。現在在農場旁的研究館辦公室寫點東西,希望可以跟世界分享生物資訊與園藝的樂趣!
感謝選擇匿名的朋友協助校閱初稿與提供意見,也敬請各位讀者不吝指教!
