Alpha Diversity 強調的是組內的量化數據,
接下來就來看看 Alpha Diversity 呈現的兩種常見圖表,
稀疏曲線(rarefaction curve)、箱形圖 (Box plot) :
檔案 : alpha-rarefaction.qzv
稀疏曲線用於生態學中時,
可以確認樣本中的序列數是否足夠代表整個樣本,
也可以用來確認這個樣本採樣時是否來自同一批群體 (有無汙染或是來自兩個群體)。
(Whether a group of samples are from the same community)
簡單來說,想像在同一樣本中多次隨機抽樣,
每次抽樣的序列數逐漸增加,並觀察每次抽樣所含有的OTU數量,
隨著抽樣序列數變多,OTU 數量逐漸飽和,
即可以推論樣本中的序列數足夠代表整個樣本。
alpha-rarefaction.qzv
調整到 Metric : observed_features 及 Index 組 :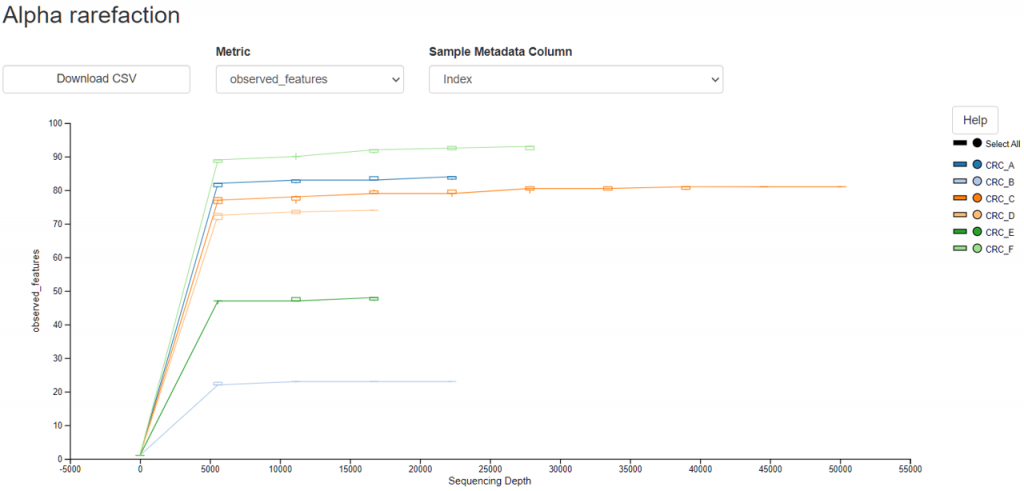
橫軸為抽樣的序列數,範圍為0~55000
([Day 16],設定--p-max-depth 為 50131 的緣故)。
縱軸為觀察到的 OTU 數量(observed_features),
屬於物種豐富度指數 (Species richness),
可以發現在約莫6000條的位置,所有樣本都趨於平緩。
推論樣本中的序列數足夠代表整個樣本。
若發現你的樣本長這樣一直往上跑停不下來,無法收斂 (紅色),
就要特別注意這個樣本的豐富度是否被低估 :
Reference : cd-genomics.com
也可以用箱形圖方式呈現樣本多樣性情況,
此一方式是可以將各組別放在一起比較,
並帶有檢定統計。
資料視覺化輸出 - observed_features_vector
方便與稀疏曲線比較,同樣使用 observed_features
qiime diversity alpha-group-significance \
--i-alpha-diversity core-metrics-results/observed_features_vector.qza \
--m-metadata-file sample-metadata.tsv \
--o-visualization observed_features_vector.qzv
放到 QIIME2 VIEW 檢視結果圖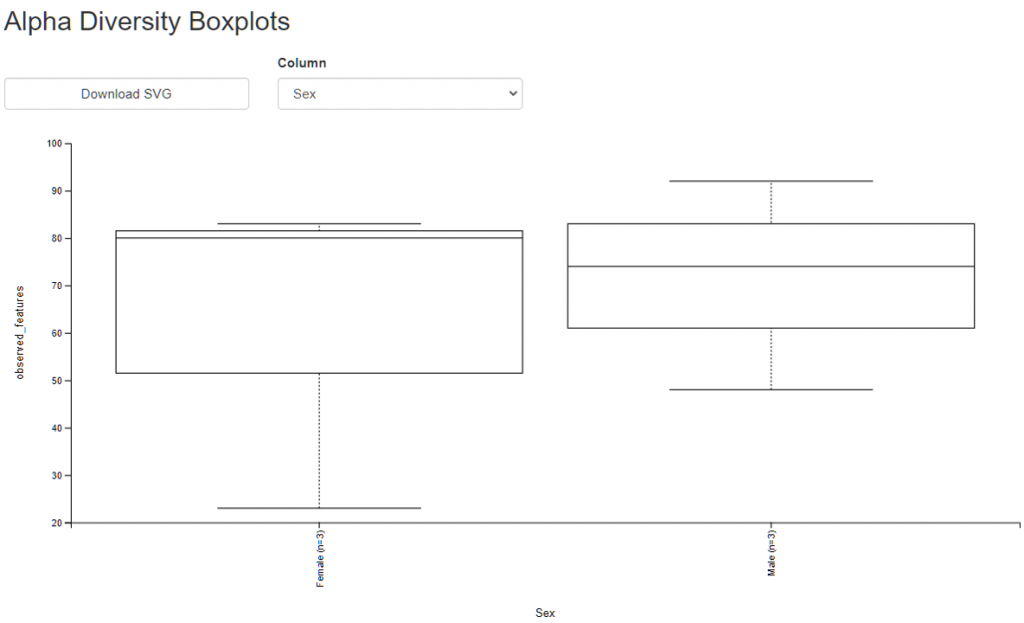
橫軸是女性、男性,縱軸則一樣是 observed_features,
無論是稀疏曲線還是箱形圖都可以根據不同多樣性指數繪製,
我們可以仔細看,女性中最小值是不是與稀疏曲線CRC_B相同。
上述的圖表方式其 縱軸(統計方式) 都是可以替換的,
[Day 16] 有提到輸出了一坨的檔案,不過有些是明天會說到的 Beta diversity,
下面列出各類統計方式供區分與參考(照字母順序) :
| 檔名 | 方法 |
|---|---|
| bray_curtis_* | Beta diversity |
| evenness_* | Alpha diversity |
| faith_pd_* | Alpha diversity |
| jaccard_* | Beta diversity |
| observed_features_* | Alpha diversity |
| shannon_* | Alpha diversity |
| unweighted_unifrac_* | Beta diversity |
| weighted_unifrac_* | Beta diversity |
雖然看起來很複雜,但其實可以將 Alpha Diversity 統計分為下列三類 :
上述指數其實網路上蠻好搜尋到的,我們來看看一個其中一個酷酷的 faith_pd,
Faith Phylogenetic Diversity 是根據親緣關係樹 (branch-based) 的物種多樣性指數,
如下圖,若兩群集間物種種類相同 (皆為4種)、物種數量的分配相同 (皆各佔25%),
無論是豐富度、均勻度,甚至是 shannon 多樣性指數都會相同,
但仔細看可以發現,
Community A 樹、蝴蝶、花、狐狸彼此親緣差異大,
Community B 僅有樹與狐狸的親緣差異較大,
因此 Community A 物種間親緣關係差距明顯較 Community B 大 :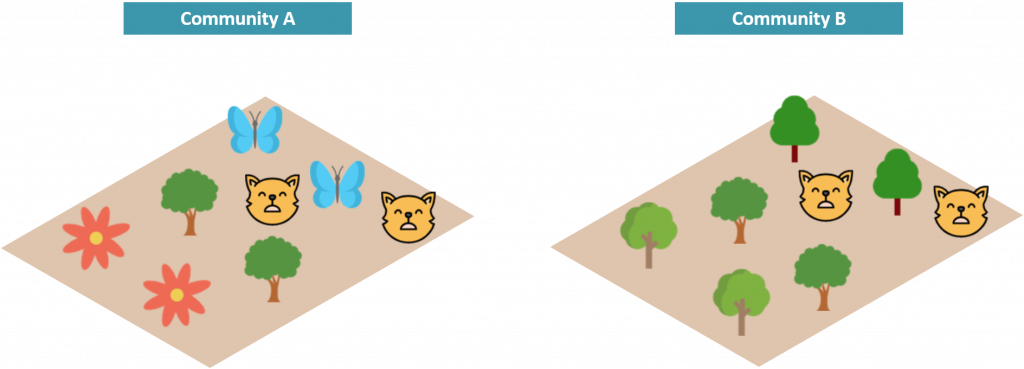
Icon : iconpacks.net
為了能夠呈現這樣子的差異,Faith Phylogenetic Diversity 以關係樹節點的量化方式,
示意圖中左圖彼此的親緣關係較遠, 右圖則其中三個物種較為接近 :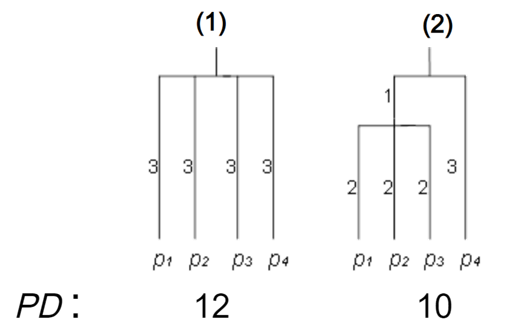
僅示意圖,非真實情況
Reference : 邱春火 清華大學統計研究所、SAM' NOTE
因此,將每個節點數值相加後,PD : A > B,
我們將這個概念套用在菌相中,
也可以得知組內的菌相親緣關係的情況。
本篇使用到的輸入/輸出檔案 :Input : core-metrics-results/observed_features_vector.qza、sample-metadata.tsvOutput: observed_features_vector.qzv、alpha-rarefaction.qzv、core-metrics-results (folder)
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
