Beta Diversity 強調的是組與組之間的量化數據,
從範例檔案來看,每個樣本有約莫100種菌 (確切來說是OTU),
如果想要繪製一個立體圖來表示每個樣本的位置,
每一種菌都有一個對應的豐富度,
兩種菌可以在 X-Y 平面圖(2D)中放置一個點代表該樣本,
三種菌可以在 X-Y-Z 三維圖(3D)中放置一個點代表該樣本,
四種...五種...一百種該怎麼辦呢??
難不成要畫一個 100D圖嗎?
降維(reduction of dimensionality) 就是很常見處理方式,
將多維度資料透過投影 (Projection) 與旋轉 (Rotation) 留下主要特徵,
還記得高中地理學過將地球(3D)投影成平面地圖(2D)嗎?
這就是日常最常見的降維處理,
而我們也知道不同投影方式會犧牲面積、距離等因素 :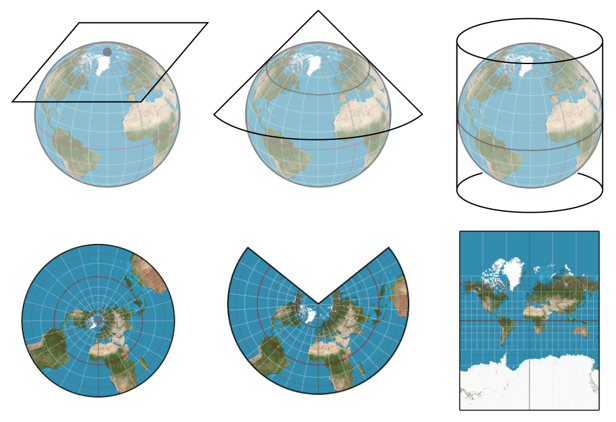
Reference : UCGIS
在生態學角度,要比較組與組之間的差異考量的因素有三 :
投影與距離計算方式
必須將兩組或以上的多維度資料中找出差異大的數據,
重新繪製於二維或三維座標系上,
常見的方式包含 PCA, PCoA, NMDS 等 (可參考 Biotools 文章)。
(可在[Day 16]檔案發現含有 pcoa 的名稱)
權重 (weight)
有無加入權重(weighted)也是考量的因素之一,
就像是國高中成績單總成績主科會加權個2倍 3倍 QQ,
若存在一些菌佔了較高比例(如優勢菌等),
可以考慮使用 Weighted (Bray curtis、Weighted unifrac),
菌種分布平均或欲觀察整體分布,
適合使用 Unweighted (Jaccard、Unweighted unifrac),實務上就是兩者都拿出來看看,哪個符合預期目標。
(可在[Day 16]檔案發現含有 unweighted weighted 的名稱)
親緣關係樹 (Phylogenetic tree)
與 [Day 17] 中所描述的 faith pd 概念類似,
加入了親緣關係的因素。
推薦參考 盛夏不等式 文章,
(可在[Day 16]檔案發現含有 bray_curtis jaccard unifrac 的名稱)
其實在 [Day 16] QIIME2已經分析好了,
| 檔名 | 方法 |
|---|---|
| bray_curtis_* | Beta diversity |
| evenness_* | Alpha diversity |
| faith_pd_* | Alpha diversity |
| jaccard_* | Beta diversity |
| observed_features_* | Alpha diversity |
| shannon_* | Alpha diversity |
| unweighted_unifrac_* | Beta diversity |
| weighted_unifrac_* | Beta diversity |
只是需要轉換為 qzv ,並告訴軟體所想要比較的組別 (範例為 Sex):
範例 : unweighted_unifrac_distance_matrix.qza
qiime diversity beta-group-significance \
--i-distance-matrix core-metrics-results/unweighted_unifrac_distance_matrix.qza \
--m-metadata-file sample-metadata.tsv \
--m-metadata-column Sex \
--o-visualization core-metrics-results/unweighted_unifrac_Sex_site_betasig.qzv \
--p-pairwise
--i-distance-matrix : 可自行變換表格上所述不同量化方式的
Betadiversity .qza
--m-metadata-column : 想要比較的組別 (進階學習)
--p-pairwise : 成對樣本檢定
完成後會顯示 :
'
Saved Visualization to: core-metrics-results/unweighted_unifrac_Sex_site_betasig.qzv
'
拖曳到 QIIME2 VIEW,根據檔名不同會有兩種的呈現方式 :
箱形圖 (帶有 betasig 名稱的.qzv)
範例 : unweighted_unifrac_Sex_site_betasig.qzv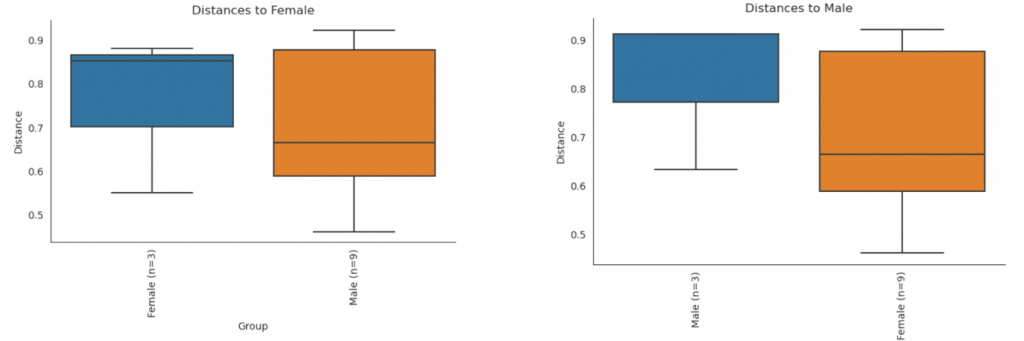
3D Plot (帶有 emperor 名稱的.qzv)
範例 : weighted_unifrac_emperor.qzv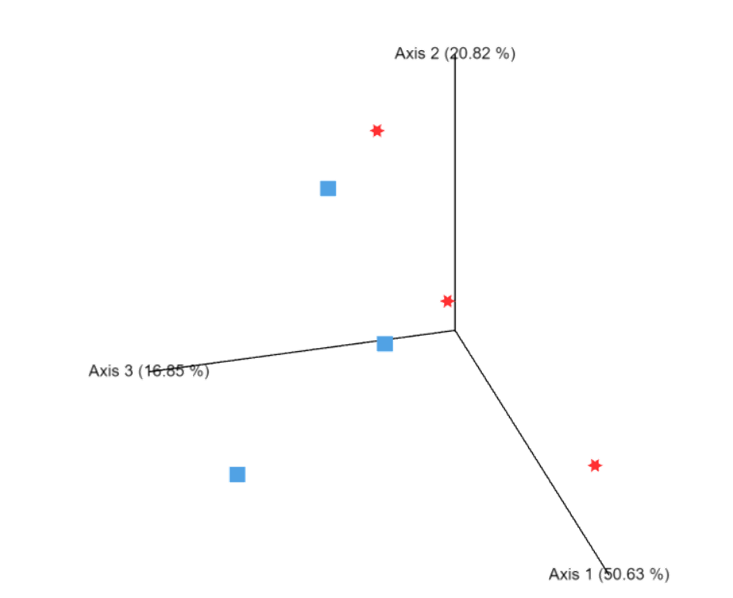
本篇使用到的輸入/輸出檔案 :Input : sample-metadata.tsv、core-metrics-results/unweighted_unifrac_distance_matrix.qzaOutput: alpha-rarefaction.qzv、core-metrics-results (folder)
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
