假設你走路走著撞見了一片森林,
想知道森林中樹種多樣化程度,要如何進行量化分析呢?
在生態學角度,
「多樣」這件事情可以分成
物種豐富度 (Species richness)、物種均勻度 (Species evenness) :
- 物種豐富度 指的是物種的數量,數量越多豐富度越大。
- 物種均勻度 指的是群集(community)中物種數量的分配情況,
物種彼此數量越接近,均勻度越高。
Reference : 科學 Online
森林1 與 森林2 進行比較,
兩者物種豐富度 相同,都是16顆樹,
但 森林1 4個不同物種數量接近(均勻),物種均勻度 森林1 > 森林2 :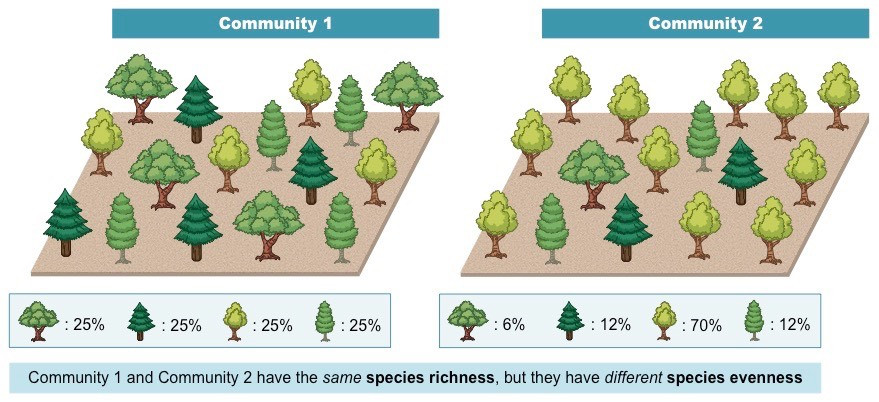
Reference : bioninja
我們把森林1、森林2... 想像成範例檔案中 Female、Male 兩組,
想知道女性與男性組內的菌相多樣性如何? 並量化成數字做成圖
我們可以用 組內多樣性 Alpha diversity 找到答案,
「組內」指的是樣本內的意思,觀察樣本內的多樣性情況,
QIIME2 提供三種指數進行計算 (嗅到生物統計學的味道了 :
並提供兩種視覺化呈現方式 :
先把程式跑完,後面慢慢解讀~
先啟動 qiime2-2022.8 環境
conda activate qiime2-2022.8
會使用到rooted-tree.qza、table-dada2-240.qza、sample-metadata.tsv
帶上[Day 15] 的便條紙,這邊要告訴 QIIME2 最大的序列條數是多少 :
qiime diversity alpha-rarefaction \
--i-table table-dada2-240.qza \
--i-phylogeny rooted-tree.qza \
--p-max-depth 50131 \
--m-metadata-file sample-metadata.tsv \
--o-visualization alpha-rarefaction.qzv
--p-max-depth : 可視為所有樣本中,最大的序列條數
完成後會顯示 :
'
Saved Visualization to: alpha-rarefaction.qzv
'
同一張便條紙,這邊要告訴 QIIME2 統計的取樣深度是多少 :
qiime diversity core-metrics-phylogenetic \
--i-phylogeny rooted-tree.qza \
--i-table table-dada2-240.qza \
--p-sampling-depth 19914 \
--m-metadata-file sample-metadata.tsv \
--output-dir core-metrics-results
--p-sampling-depth : 取樣深度
--output-dir : 輸出的資料夾,若原本無此資料夾會自動創一個,
因為此次產出檔案眾多,用一個資料夾裝他們。
完成後會顯示 :
'
Saved FeatureTable[Frequency] to: core-metrics-results/rarefied_table.qza
Saved SampleData[AlphaDiversity] to: core-metrics-results/faith_pd_vector.qza
Saved SampleData[AlphaDiversity] to: core-metrics-results/observed_features_vector.qza
Saved SampleData[AlphaDiversity] to: core-metrics-results/shannon_vector.qza
Saved SampleData[AlphaDiversity] to: core-metrics-results/evenness_vector.qza
Saved DistanceMatrix to: core-metrics-results/unweighted_unifrac_distance_matrix.qza
Saved DistanceMatrix to: core-metrics-results/weighted_unifrac_distance_matrix.qza
Saved DistanceMatrix to: core-metrics-results/jaccard_distance_matrix.qza
Saved DistanceMatrix to: core-metrics-results/bray_curtis_distance_matrix.qza
Saved PCoAResults to: core-metrics-results/unweighted_unifrac_pcoa_results.qza
Saved PCoAResults to: core-metrics-results/weighted_unifrac_pcoa_results.qza
Saved PCoAResults to: core-metrics-results/jaccard_pcoa_results.qza
Saved PCoAResults to: core-metrics-results/bray_curtis_pcoa_results.qza
Saved Visualization to: core-metrics-results/unweighted_unifrac_emperor.qzv
Saved Visualization to: core-metrics-results/weighted_unifrac_emperor.qzv
Saved Visualization to: core-metrics-results/jaccard_emperor.qzv
Saved Visualization to: core-metrics-results/bray_curtis_emperor.qzv
'
沒錯,就是這麼多,跑出了滿滿的統計圖,真的是很方便。
本篇使用到的輸入/輸出檔案 :Input : table-dada2-240.qza、rooted-tree.qza、sample-metadata.tsvOutput: alpha-rarefaction.qzv、core-metrics-results (folder)
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
