科學毛怪定序報告請下滑至 STAMP 圖形化介面視覺化軟體 章節開始閱讀。
PICRUSt2 強大之處可以藉由菌相資料預測獲得 Enzyme Commission (E.C number)、KEGG Orthology (KO)、MetaCyc pathway 豐富度資料,用比較輕鬆的說法就是 : PICRUSt2 可以預測菌群中的酵素及代謝途徑豐富度。
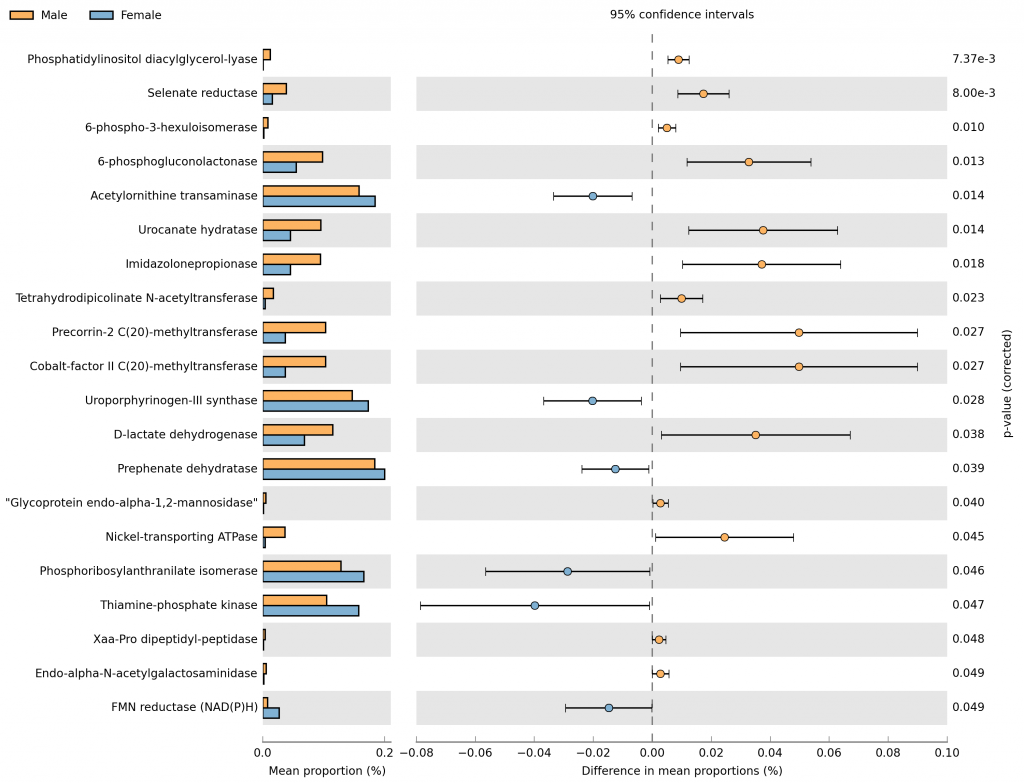
利用 PICRUSt2 的 E.C number 預測結果繪製的組間酵素豐富度比較。
PICRUSt2 分析後主要利用下列的檔案解壓縮後視覺化,
分別是以E.C、KO、Pathway :
現在並沒有上述的檔案,需要先進行以下步驟。
由於PICRUSt2原始輸出時僅含有酵素及代謝途徑編號,為方便人類判讀,會使用 PICRUSt2 Add descriptions 功能替編號加上註釋 :
操作下列指令時需先啟動 PICRUSt2 環境並 cd 到
picrust2_out_pipeline資料夾 。
add_descriptions.py \
-i EC_metagenome_out/pred_metagenome_unstrat.tsv.gz \
-m EC \
-o EC_metagenome_out/pred_metagenome_unstrat_descrip.tsv.gz
add_descriptions.py \
-i pathways_out/path_abun_unstrat.tsv.gz \
-m METACYC \
-o pathways_out/path_abun_unstrat_descrip.tsv.gz
path_abun_unstrat_descrip.tsv.gz、pred_metagenome_unstrat_descrip.tsv.gz拉到本機端並解壓縮,可以獲得分別含有 KO/EC/Pathway、註釋、樣本名、豐富度的 tsv 三個檔案。
注意 KO 與 EC 出來的檔名相同,避免混淆可以重新命名
EC_metagenome_out/pred_metagenome_unstrat_descrip.tsv.gzdescription CRC_A CRC_B CRC_C CRC_D ...
Alcohol dehydrogenase 18794.29 19032.78 31185.93 17137.23 ...
3-oxoacyl-[acyl-carrier-protein] reductase 43579.57 32083.94 60889.85 31771.04
.
.
.
sample-metadata.tsv拉到本機端,並刪除#q2:types 那一行,完成後如下 :
sample_name Index Sex
CRC_A CRC_A Female
CRC_B CRC_B Female
CRC_C CRC_C Female
CRC_D CRC_D Male
CRC_E CRC_E Male
CRC_F CRC_F Male
做研究已經很辛苦了,這邊採用使用者友善的菌相功能預測視覺化軟體 STAMP :
There are many possible ways to analyze PICRUSt2 output. STAMP is one tool that can be used that requires no background in scripting languages.
Reference : picrust
純指令的 Linux 系統 是開不起來的,需使用圖形化作業系統。
Ctrl + O 載入檔案,
Profile file 是‵pred_metagenome_unstrat_descrip.tsv,
Group metadata file 是 sample-metadata.tsv
或
Profile file 是pred_metagenome_unstrat.tsv,
Group metadata file 是 sample-metadata.tsv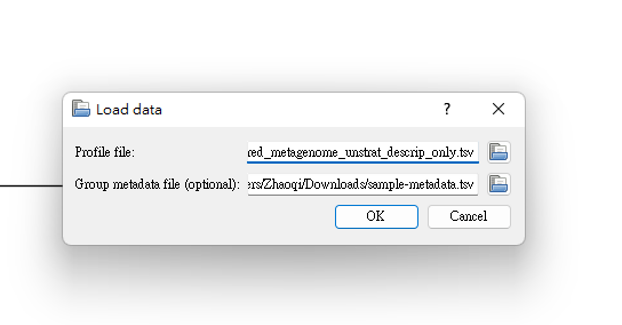
使用
pred_metagenome_unstrat_descrip.tsv可能因檔案中的酵素代號不同但名稱相同,出現錯誤而無法載入,(Data does not form a strict hierarchy...),建議改用pred_metagenome_unstrat.tsv。
將組別設定切換進來,範例我們調整到 Sex 組 (因為Index一人一組在這裡無意義),若有不同組別可之後再調整 :
STAMP 提供三種類別,多重組別、兩組比較、兩樣本比較,
先選 Two samples (左圖),再選擇產圖方式,這邊我們選 Profile bat plot (右圖):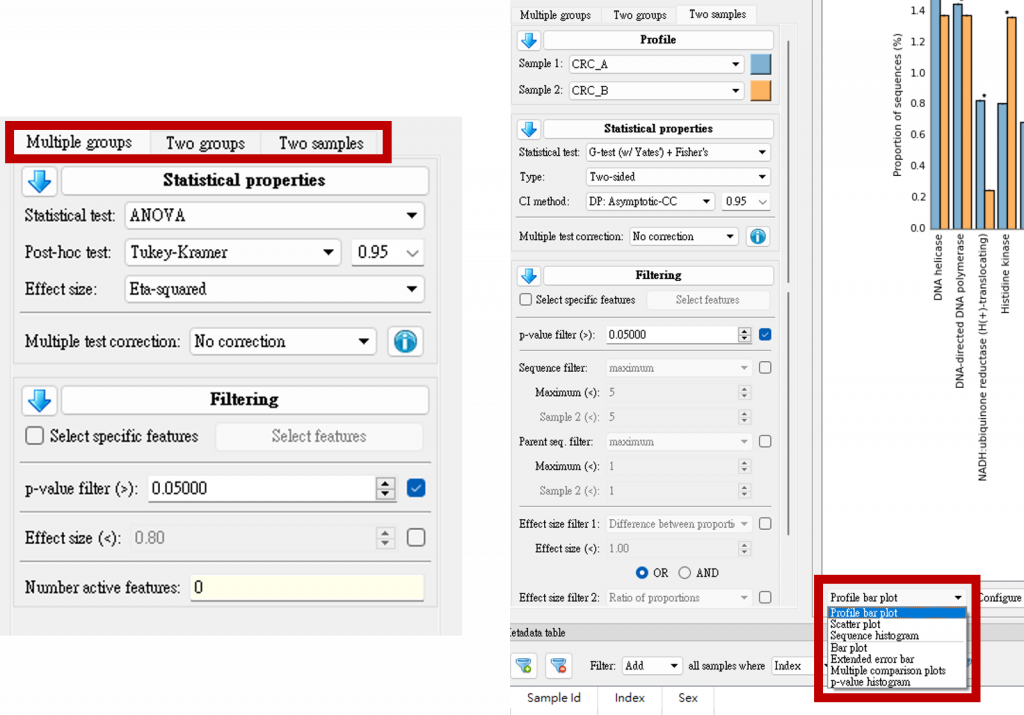
仔細看軟體有很多參數可以調整(選擇樣本、統計檢定方式、p value等),
也能在 Configure plot 調整圖片參數。調整完後可以按Ctrl + s存圖,檔名用英文,中文會報錯!
以下皆使用軟體預設的統計方式 :
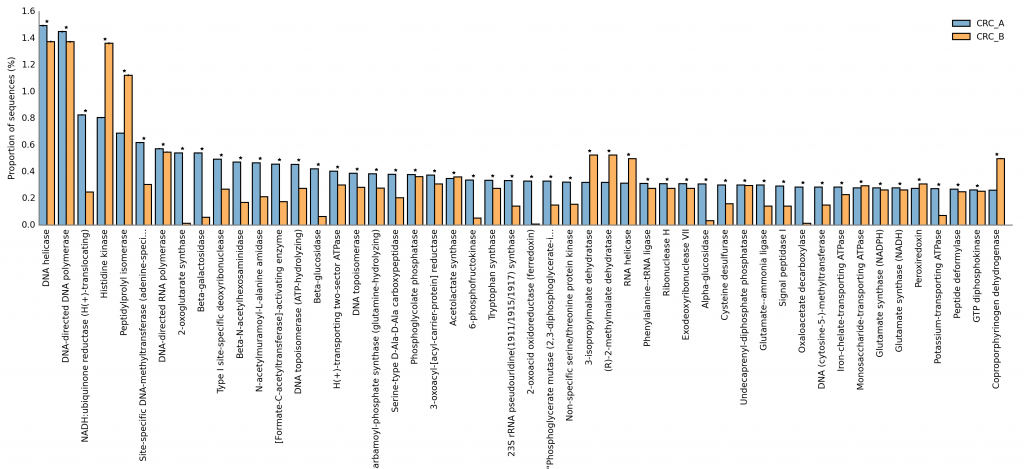
E.C. number abundance / Two samples / CRC_A & CRC_B / Profile bar plot

E.C. number abundance / Two groups / Female & Male / Extended error bar
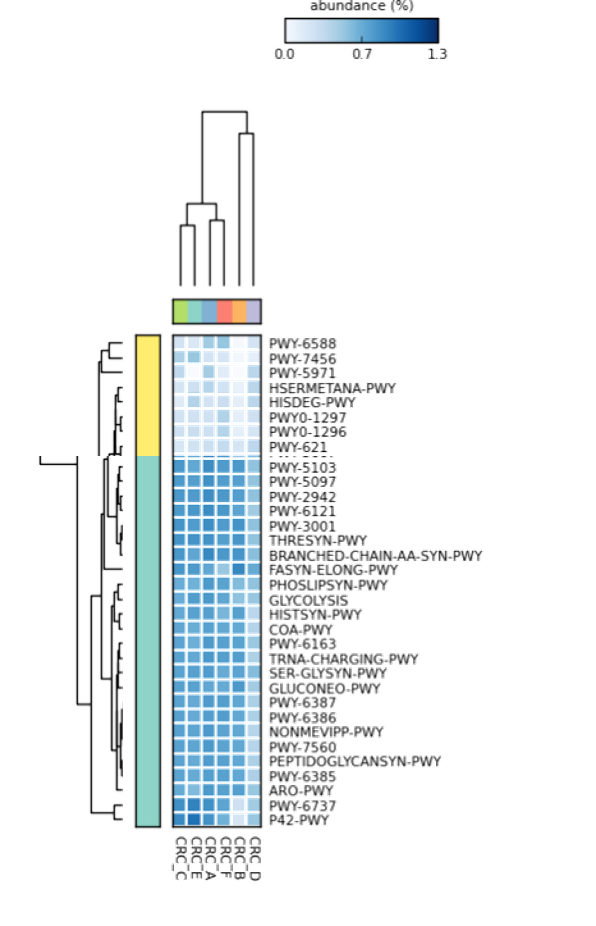
Pathway abundance / Mutiple groups / Heatmap plot /
Width : 12, Height : 50 in Configure plot (原圖過長已裁切)
本篇使用到的輸入檔案 :
EC_metagenome_out/pred_metagenome_unstrat.tsv.gz
pathways_out/path_abun_unstrat.tsv.gz
經歷觀念介紹、檔案製備、品質管制、視覺化資料、多樣性統計、功能性預測等,
如果你趁著一股熱血做到這裡,差不多表情長這樣 :![]()
最難的永遠不是產圖,而是找出數據背後的生物意義。
喘一口氣,
下篇整理過去點點滴滴,最後幾篇迎接更炫炮的第三代定序 (TGS) 資料分析 !!
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
