接下來我們在這裡要介紹一些比較雜的演算法像是Floyd Warshall algorithm, minimum spanning tree(MST), Kruskal algorithm 還有 Prim’s algorithm。
Floyd Warshall algorithm 可以用來計算任意兩點間的最短距離,它跟Bellman-Ford一樣無法用在有negative cycle的圖,但它也無法偵測圖是否有negative cycle。如果你看它的程式碼,它有點像是用窮舉的方法不斷做比較,見以下圖說明: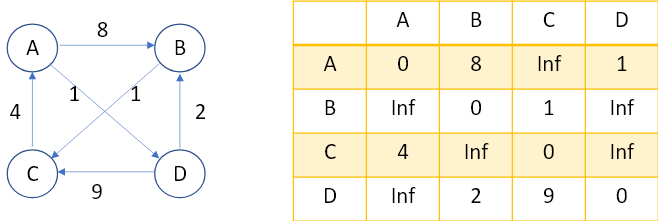
圖1,假設今天我們有個路徑如左圖,我們想用Floyd-Warshall找到任意兩點間的最短距離,首先我們將左圖改寫成如右圖的陣列。若兩點間無直接通往的箭頭者,我們用Inf代替; 自己到自己距離為0,故對角線為0。
接著我們要試著藉由更新陣列,算出兩兩點間的最短距離,舉例來說: AB=8,會拿他和AA+AB=8, AB+BB=8,AC+CB=Inf,AD+DB=3做比較,這裡我們發現3小於8,所以我們Array[A][B]更新為3。以此類推,直到整個陣列更新完畢,時間複雜度O(V^3)(三層loop),空間複雜度O(V^2)(創了VxV)的陣列。我們直接來看個程式碼:
import numpy as np
def FloydWarshall(graph):
numV = len(graph)
for i in range(numV):
for j in range(numV):
for k in range(numV):
graph[j][k] = min(graph[j][k], graph[j][i]+graph[i][k])
return graph
G = np.array([[0, 8, float('Inf'), 1], [float('Inf'), 0, 1, float('Inf')], [
4, float('Inf'), 0, float('Inf')], [float('Inf'), 2, 9, 0]])
print(FloydWarshall(G))
>> [[0. 3. 4. 1.]
[5. 0. 1. 6.]
[4. 7. 0. 5.]
[7. 2. 3. 0.]]
最小生成樹為樹的一個子集合,其邊(edges)是互相連結節點、沒有方向性、有權重,所有vertices連在一起、不會形成一個cycle(封閉循環),然後所有邊的權重加起來為最小。實際的例子譬如說,有五個獨立的小島,你要在島和島中間蓋橋,是得五個島相連,但蓋橋成本最低。(課程中的例子)那在那之前,要先介紹一下disjoint set(併查集): Disjoint set 為一資料結構,用來幫助追蹤元素集,這些元素集被劃分為許多不相交且不重疊的集,並且每個集都有代表(幫助識別該集),常用來檢測圖是否相連。它有幾個功能:
MakeSet(x):創建一個包含單個元素x的新集合。
Union(x,y): 將包含x的集合與包含y的集合合併。
Find(x): 確定特定元素x屬於哪個幾何並返回屬於x所在集合的代表元素。
class DisjointSet:
def __init__(self, vertices):
self.vertices = vertices
self.parent = {}
for i in vertices:
self.parent[i] = i
self.rank = dict.fromkeys(vertices, 0)
# 尋找你要找的element屬於那個子集合
def find(self, x):
if self.parent[x] == x:
return x
else:
return self.parent[x]
def union(self, x, y):
xroot = self.parent[x]
yroot = self.parent[y]
# 比較他們的rank,比較大的併別人
if self.rank[xroot] > self.rank[yroot]:
self.parent[yroot] = xroot
elif self.rank[xroot] < self.rank[yroot]:
self.parent[xroot] = yroot
else:
# 一開始沒有大小差別讓xroot變mother
self.parent[yroot] = xroot
self.rank[xroot] += 1
if __name__ == '__main__':
vertices = ['A', 'B', 'C', 'D', 'E', 'F']
ds = DisjointSet(vertices)
ds.union('A', 'B')
ds.union('A', 'E')
# 來看E屬於哪個集合
print(ds.find('E'))
print('===check the parent dictionary===')
print(ds.parent)
# E的集合
>> 'A'
===check the parent dictionary===
{'A': 'A', 'B': 'A', 'C': 'C', 'D': 'D', 'E': 'A', 'F': 'F'}
好惹,接著我們來看怎麼用Kruskal和Prim algorithm解最小生成樹。
Krusal algorithm是一種貪婪算法(greedy algorithm),用來算加權無向圖(weighted undirected graph)的最小生成樹。首先將每個節點用disjointset變成一個個set,然後將他們根據權重(weight)由小到大排序,然後由小到大union兩兩節點N-1次(當他們不屬於同一個union時),即可得解,我們來看程式碼可能比較快:
import DisjointSet as dst
class Graph:
def __init__(self):
self.numV = 0
self.graph = []
self.MST = []
self.vertices = []
def add_edge(self, s, d, w):
self.graph.append([s, d, w])
def add_node(self, v):
self.vertices.append(v)
self.numV += 1
def KruskalAlgo(self):
i, c = 0, 0
ds = dst.DisjointSet(self.vertices)
self.graph = sorted(self.graph, key=lambda x: x[2])
while c < self.numV-1:
s, d, w = self.graph[i]
i += 1
x = ds.find(s)
y = ds.find(d)
if x != y:
self.MST.append([s, d, w])
ds.union(x, y)
c += 1
res = ''
for s, d, w in self.MST:
res += f'{s}-{d}:{w}'
res += '\n'
print(res)
g = Graph()
g.add_node('A')
g.add_node('B')
g.add_node('C')
g.add_node('D')
g.add_node('E')
g.add_edge('A', 'B', 5)
g.add_edge('A', 'C', 13)
g.add_edge('A', 'E', 15)
g.add_edge('B', 'A', 5)
g.add_edge('B', 'C', 10)
g.add_edge('B', 'D', 8)
g.add_edge('C', 'E', 20)
g.add_edge('C', 'A', 13)
g.add_edge('C', 'B', 10)
g.add_edge('C', 'D', 6)
g.add_edge('D', 'C', 6)
g.add_edge('D', 'B', 8)
g.add_edge('E', 'A', 15)
g.add_edge('E', 'C', 20)
g.KruskalAlgo()
Prism’s algorithm也是一種解加權無向圖(weighted undirected graph)的最小生成樹的貪婪演算法。首先,先任意選擇一個節點作為起始點,並將之標為visited,觀察周圍的邊,找最小權重者,選擇最小邊到達的點,標為visited,將此段存於MST list中,再找下一個連結的最小邊,再將之到達的點標為visited,以此類推直到所有節點都visited。這個影片解釋得不錯,才兩分鐘,可以參考一下: https://www.youtube.com/watch?v=cplfcGZmX7I
直接來看程式碼:
import sys
class graph:
def __init__(self, node, edges):
self.node = node
self.edges = edges
self.MST = []
self.vnum = len(self.node)
# time complexity: O(V^3), space complexity: O(V)
def primsalgo(self):
visited = [0]*self.vnum
visited[0] = True
c = 0
while c < self.vnum-1:
min = sys.maxsize
for i in range(self.vnum):
if visited[i]:
for j in range(self.vnum):
if ((not visited[j]) and (self.edges[i][j])):
if min > self.edges[i][j]:
min = self.edges[i][j]
s = i
d = j
self.MST.append([self.node[s], self.node[d], self.edges[s][d]])
visited[d] = True
c += 1
res = ''
for s, d, w in self.MST:
res += f'{s}->{d}:{w}\n'
return res
edges = [[0, 10, 20, 0, 0],
[10, 0, 30, 5, 0],
[20, 30, 0, 15, 6],
[0, 5, 15, 0, 8],
[0, 0, 6, 8, 0]]
nodes = ['A', 'B', 'C', 'D', 'E']
g = graph(nodes, edges)
print(g.primsalgo())
>>
A->B:10
B->D:5
D->E:8
E->C:6
參考資料:
http://alrightchiu.github.io/SecondRound/minimum-spanning-treeintrojian-jie.html
The Complete Data Structures and Algorithms Course in Python (from Udemy)
