在昨天開啟了copy number variation系列文的篇章之後,簡單地介紹了CNV是什麼,以及CNV的生物意義,今天我將來介紹目前常見的CNV檢測方法有哪些及背後的實驗原理 [1]。如果我的時間夠且精神狀況良好的話,我會繼續介紹一些用來檢測CNV的生物資訊方法。
FISH是一種1980年代早期由一群生物醫學專家所發展出來的方法,可用來偵測、定位染色體上某段DNA序列是否存在。到了現在,這個方法當然不只是用來檢測DNA片段,也可被用來檢測某些特定的RNA分子,其檢測原理(見下圖)皆是基於偵測螢光信號強弱的方式 [2]。一開始必須先建構出所使用的探針(probe),這個探針的大小取決於這個探針是否能專一地找到它的目標與之配對雜交。這些探針同時還會加入標定物,在雜交反應開始後,再使用可辨識那些標定物的螢光抗體與之結合。此抗體會專一性結合上樣本中已經被標定過的核酸,因此這些被螢光標定的抗體便可在螢光顯微鏡下被偵測到。

aCGH是一種基於比較兩個不同情況/樣本之間相對倍數關係來分析CNV的分子細胞遺傳方法(molecular cytogenetic method),這個方法透過晶片來實行,藉由晶片上數萬到上百萬個DNA探針與來自樣品的DNA片段雜交,進行全基因體(genome-wide)的遺傳分析,見下圖 [3]。與傳統的染色體檢查方法對比,像是G-banding,aCGH的解析度勝過數百倍以上,因此除了大片段DNA變異(如aneuploidy等),皆屬於aCGH分析的範疇。

qPCR是藉由PCR原理 [4] 將DNA信號放大,同時又可利用螢光染劑偵測每次PCR循環後產物總量的一種方法。qPCR是基於偵測樣品DNA在反應前後其產物的量化關係,越早探測到指定螢光值表示目標DNA量越多。相對於傳統PCR方法,qPCR有許多優點:其結果可以較快取得且穩定,且操作污染少。qPCR分析亦適於更具挑戰性的應用,如多重偵測與高通量分析。依其標定螢光物的不同,qPCR又可分成非專一性化學標定(SYBR Green)與專一性化學標定(TaqMan probe)兩大類 [5]:
SYBR Green I這種螢光染劑會與雙股DNA的minor groove結合,崁入DNA中,因此PCR的過程中,在每個循環結束時偵測螢光強弱,就能推算出有多少PCR的產物,但因為是非專一性的結合,所以SYBR Green I除了結合到目標產物上,同時也會結合到其他的DNA中。
透過人工合成的
TaqMan probe(oligonucleotid),在oligonucleotid兩端分別標定上不同螢光物質,而這兩螢光物質彼此間距離太近時會遮蔽掉彼此的螢光,因此無法偵測到螢光,但是當DNA進行複製時,oligonucleotid會被切割水解,讓兩個螢光物質分開,因此能偵測到螢光,螢光越強代表PCR產物越多。其中使用的probe是特異性與Taq結合,因此不會偵測到非特異性的反應。
以圖來表示,則向下圖所示 [6]:
NGS是近十年來最為重要的生物技術之一,堪稱當代生命科學領域的顯學。NGS的原理是先將樣品DNA打碎成為小片段之後,加上adapter序列滴入晶片中,這些adapter序列會與晶片上的probe互補,因此可以將DNA片段固定到晶片上。再利用前面提到的PCR方法將DNA的數量提昇數倍,重複合成DNA、偵測螢光訊號、沖洗這些步驟,便可得知樣品DNA的序列 [7]。由於這個方法可以直接獲得DNA序列,因此可以直接透過序列比對的方式以及不同的統計分析方法得知哪些區域有CNV發生。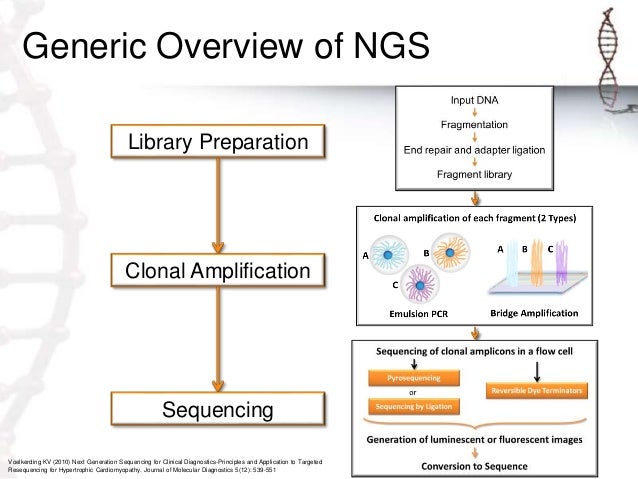
....
