比起 R 語言,python 的使用場合更多樣化,因此時常要自行引入相關功能的函式庫。在資料視覺化上最常聽到的 python 繪圖函式庫包括 matplotlib、seaborn、以及 plotly,matplotlib 是 MATLAB + plot + library 的縮寫,如其名寫作風格有點 MATLAB 的味道;如果嫌 MATLAB 寫法囉唆的話,Seaborn 是建立在 matplotlib 上的工具,且參數的資料結構與 pandas 相整合,所以如果已經習慣使用相關資料科學套件的話,seaborn 是個不卑不亢的好選擇;plotly 以及 bokeh 等等其他套件具有更多樣化的互動功能,我個人沒有使用到,以下選擇 seaborn 來簡介~

(截圖來自怪化貓海坊主)
沿用上一篇的順式作用元件 (cis-acting element) 分析結果來進行視覺化,這種分析的視覺化方式很多樣化,熱圖、直方圖、或是長條圖都可以,端看想透過圖表達的概念是什麼。我想要保留序列線性的方向關係 (-1500bp ⇒ TSS),在代表位置的橫軸上標出所在位置,並且用顏色呈現出不同生理調控的順式作用元件。
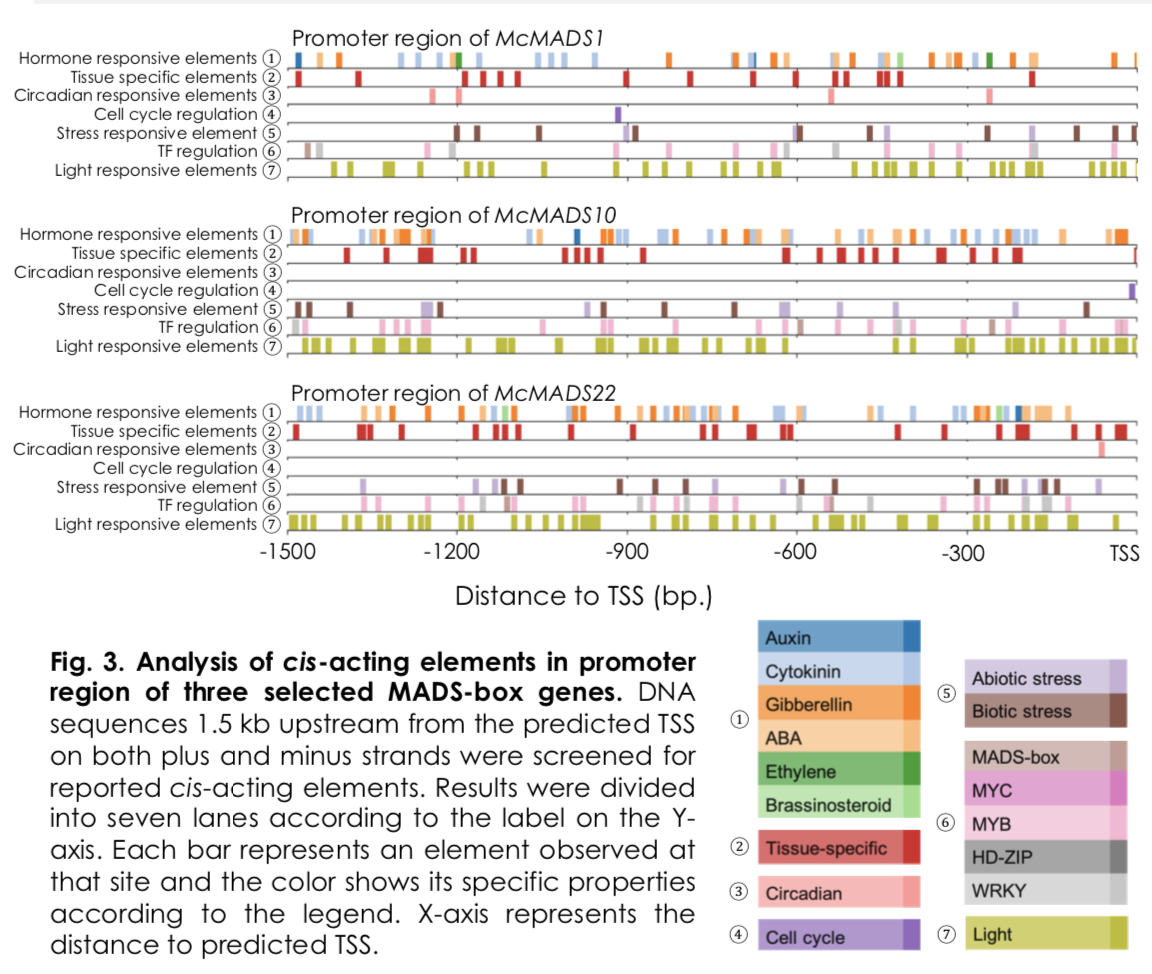
(謝晨、杜宜殷. 2019. Genome-wide Identification and Analysis of MADS-box Gene Family in Bitter Gourd.)
檢視 PLACE 輸出的純文字檔案,分成標題、時間、fasta 格式序列、執行結果說明、序列註解區、以及元件列表區,只有元件列表區整齊的格式適合我們使用,因此僅擷取元件列表區的文字另存新檔案,經過一番針對空格的調教變成 tab seperated values file。
標題、時間

fasta 格式序列
執行結果說明
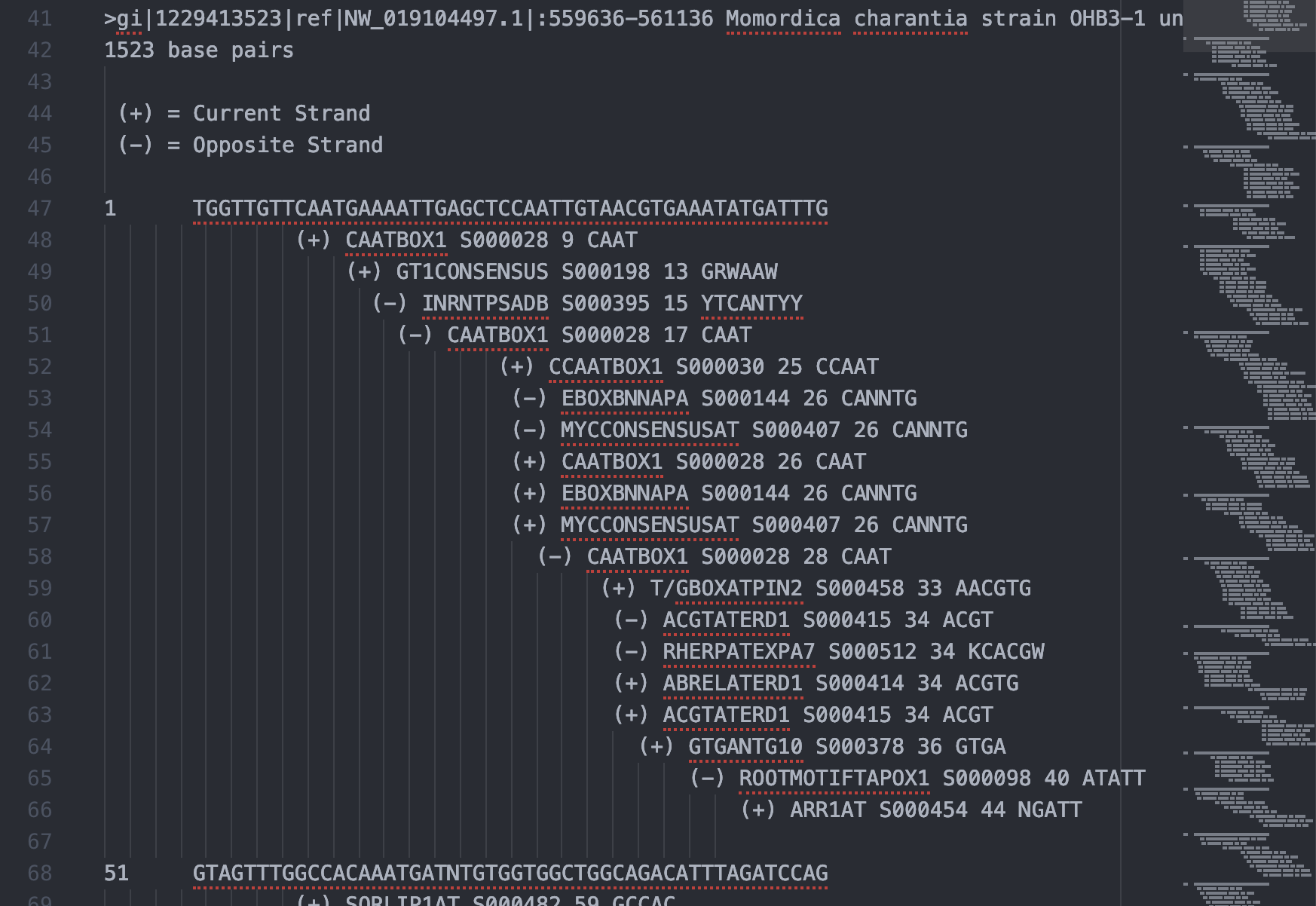
序列註解區
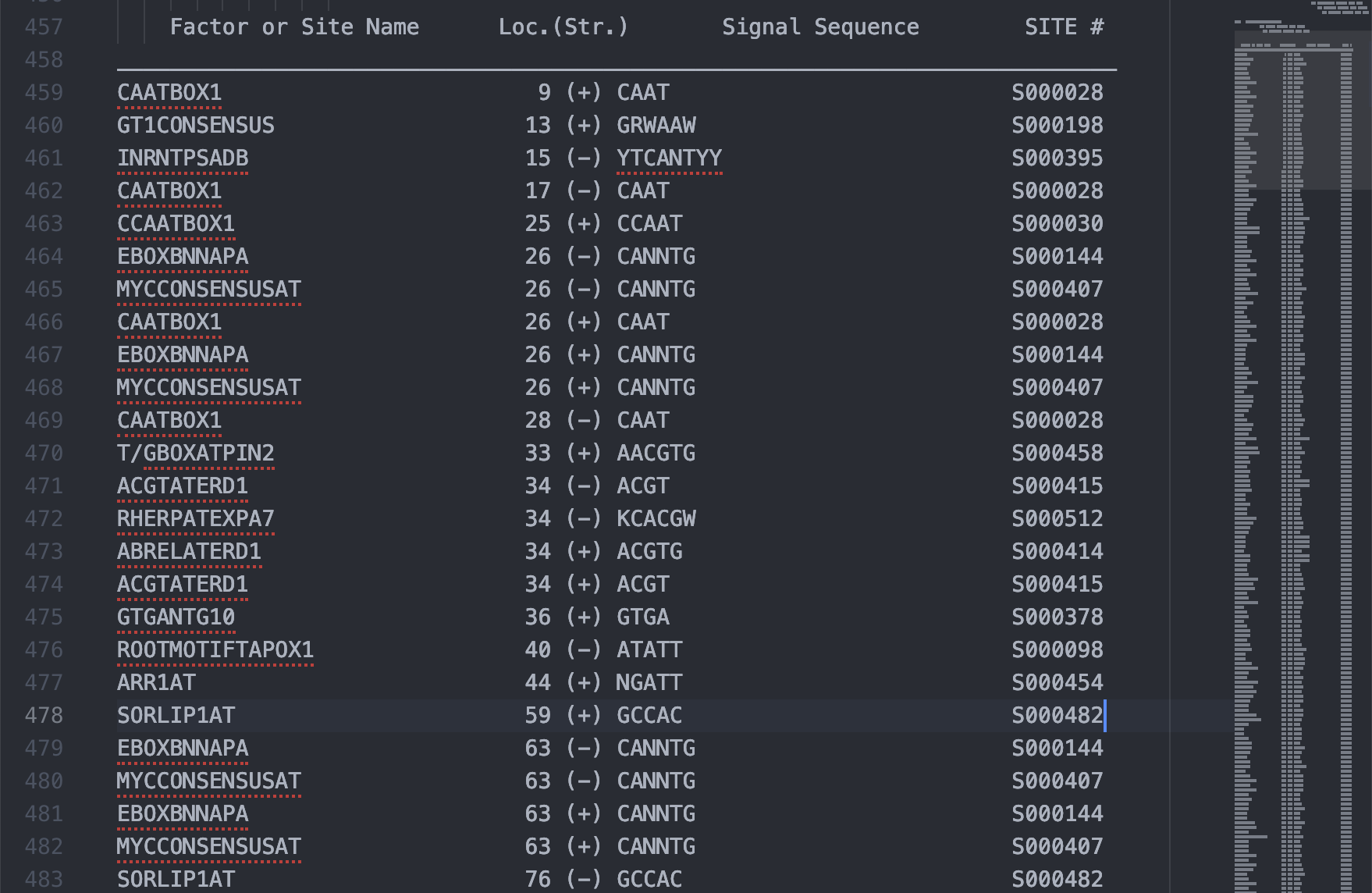
元件列表區,以及轉換成 tsv 格式的結果
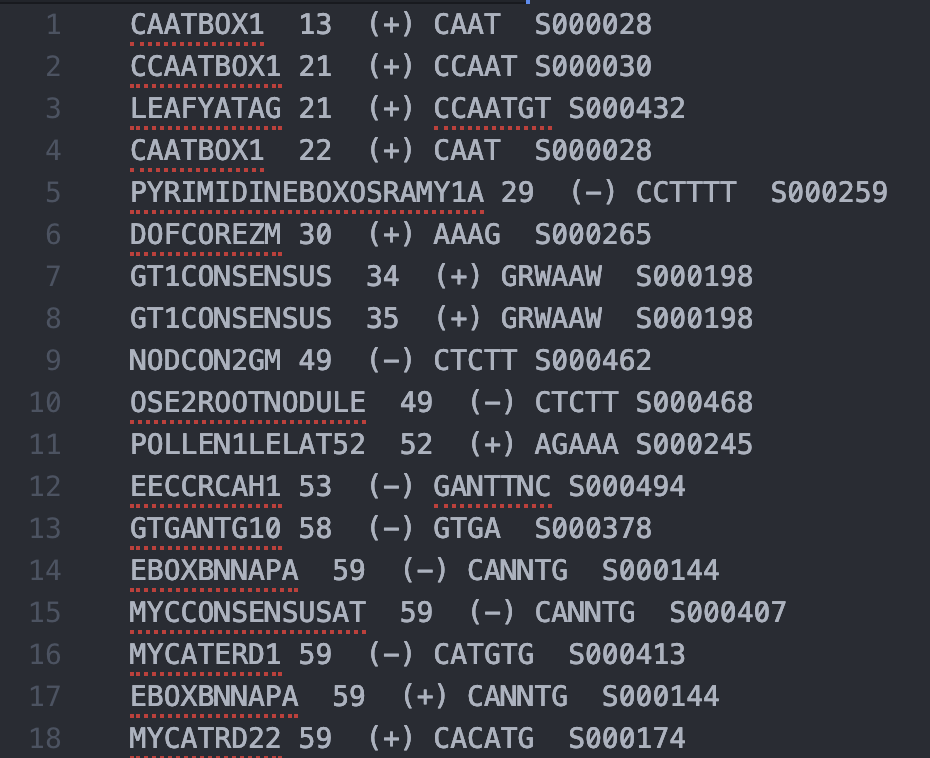
另外因為我們要將不同生理功能的順式作用元件分類,從此處下載了 PLACE 資料庫的參考資料表格,另存成 csv 檔案

引入函式庫以及使用 read_csv 函式輕鬆讀取資料,設定 dataframe 的 index
import csv
import pandas as pd
import numpy as np
import seaborn as sns
import matplotlib.pyplot as plt
import matplotlib
%matplotlib inline
place = pd.read_csv("place_reference.csv", parse_dates=True)
promoter = pd.read_csv("promoter.tsv",sep = "\t", header = None)
promoter = promoter.set_index(0)
PLACE 的參考資料表中,有著該段順式作用元件的代號、DNA 序列內容、發表文獻標題。從發表文獻之標題就可以了解該順式作用元件的功能。因此我準備了一個雙層的 list 來存放我認為夠有辨識度的關鍵字,第一層 list 用來存放不同的屬性之關鍵字,比如同樣屬於植物賀爾蒙的就放在第一個 list 組成中,跟組織部位有關的則放在第二個組成中;第二層則用以存放相同屬性的同義詞,比如同樣跟植物生長素有關的元件之文獻標題可能是 auxin 或 SAUR。這將在實際繪圖階段透過迴圈來逐個存取並辨識歸類。
由於我將順式作用元件分成七大類,每一類中又有可能有不同的成員,因此將一段啟動子分成七條來畫,不同的屬性畫在不同的道上,同一道上不同的次屬性才用不同的顏色來標示。初步設定 seaborn 繪圖要使用到的參數,色盤則是選擇 tab20,每換一種屬性就換一個顏色。
term_list =
[
[
["auxin","Auxin","SAUR"],
["cytokinin","Cytokinin"],
["gibberellin","Gibberellin","GA"],
["ABA","abscisic acid","abscisic acid"],
["ethylene","ETHYLENE", "Ethylene"],
["brassinosteroid"]
],
[
["tissue"]
],
[
["circadian"]
],
[
["cell cycle","Cell cycle","phase-","E2F"]
],
[
["abiotic stress","CBF","heat","cold", "salt","low-temp"],
["biotic stress","infected"]
],
[
["MADS"],
["MYC"],
["MYB"],
["HD-ZIP"],
["WRKY"]
],
[
["light","Light"]
]
]
fig, ax = plt.subplots(len(term_list),1,figsize=(15, len(term_list)*0.3))
cmap = matplotlib.cm.get_cmap('tab20')
xtick = range(0, 1501, 300)
color_ch = 0.04
ih = 1
ax_ch = 0
lw = 6
plt.rcParams.update({'font.size': 29})
我使用 rugplot 來畫圖,總共有七條,每一條都要調整編框顯示狀態。我的迴圈用得比較多層,邏輯上比較直觀但是效能差一點,想法上是依據我安排的關鍵字列表逐步檢索,每拿到一個關鍵字就要去 reference 表格中檢查一遍有哪個 cis-acting element 發表文章標題中有這個關鍵字,有個話才回來看 PLACE 輸出的純文字註解表格中是否有註解到,有的話便取出其註解的數值繪製到畫布上,如此循環每一個關鍵字,每次畫完一個次屬性更換顏色,每次畫完一個大屬性則更換繪製的排數,直到畫完為止。
ax_ch = 0
for term_class in term_list:
ax[ax_ch].set_xlim(0,1500)
ax[ax_ch].set_ylim(0,1)
ax[ax_ch].spines['left'].set_visible(False)
ax[ax_ch].spines['top'].set_visible(False)
ax[ax_ch].spines['right'].set_visible(False)
ax[ax_ch].get_yaxis().set_visible(False)
ax[ax_ch].set_xticklabels([])
ax[ax_ch].set_xticks(xtick)
for term_subclass in term_class:
title_list = []
id_list = []
for term in term_subclass:
for i in range(place.shape[0]):
title = place.loc[i]["title"]
if (term in str(title)):
id = place.loc[i]['identifier']
id_list.append(id)
title_list.append(title)
id_list = list(set(id_list))
id_filter = promoter.index.isin(id_list)
x = promoter[id_filter]
rgba = cmap(color_ch)
vv = list(x[1].values)
vv = list(set(vv))
sns.rugplot(vv, color = rgba, height = ih, ax = ax[ax_ch], linewidth = lw)
color_ch+=0.05
ax_ch +=1
fig
最後的圖片長得像這樣,由於我比較懶,所以沒有再回頭來優化繪圖的邏輯與迴圈使用,剩下的 x label 等等標示也都是後期另外在手稿撰寫的編修軟體中補完~
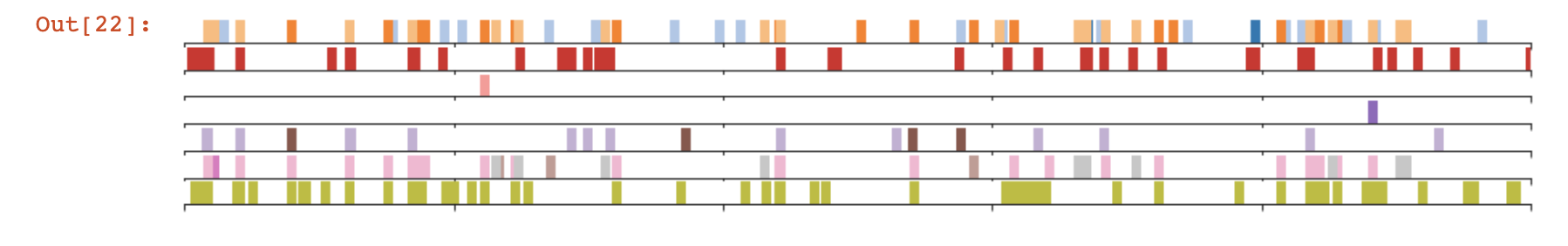
這是我少數自己花了點時間調教的繪圖腳本,還有很多更好的視覺化方式,等著大家來發掘~如果有誤或是想知道更多的話請留言告訴我~
An introduction to seaborn - seaborn 0.9.0 documentation
關於作者
謝晨 (Chen Hsieh),臺大園藝暨景觀學系研究所碩士。讀碩士前的興趣是懷著寫點程式妄圖解決農業問題的夢想參加比賽,拿了幾個黑客松與 Open Data 創新應用競賽的獎,卻都沒有勇氣將項目經營下去;研究所期間的興趣轉換成讀學術期刊的出刊電子報。靠著這些興趣當選 107 學年的臺大優秀青年,畢業後卻成了無業的實驗室居民。現在在農場旁的研究館辦公室寫點東西,希望可以跟世界分享生物資訊與園藝的樂趣!
感謝選擇匿名的朋友協助校閱初稿與提供意見,也敬請各位讀者不吝指教!
