在整個分析流程中,輸入的檔案必須轉換為 QIIME2 artifacts (.qza) 壓縮格式,
這類型檔案之後會在一個一個分析流程中穿梭,
而 .qza 是一種電腦看得懂,人類看不懂的玩意兒,
a 指的就是人為加工過的檔案 (artifacts)。
可以想成將定序資料與註釋包裝的集合檔案。
那資料視覺化呢 ?
部分的 .qza 檔案有提供轉換為 .qzv功能,
v 指的就是視覺化 (visualization) ,
各種美美的圖都會從.qzv跑出來!變成電腦看不懂,人類看了很喜歡的玩意兒
因此接下來 .qza .qzv 兩個靈魂檔案格式會充斥著本系列文!
首先,必須先拎著 [Day 06] 所得到的三個檔案,
將三者轉換為一個 QIIME2 artifacts (.qza),幫助匯入。
先啟動 qiime2-2022.8 環境 :
conda activate qiime2-2022.8
第一步先將上述三個檔案匯入轉換為 .qza :
qiime tools import \
--type 'SampleData[PairedEndSequencesWithQuality]' \
--input-format PairedEndFastqManifestPhred33V2 \
--input-path manifest.tsv \
--output-path demux.qza
qiime tools import : 使用 qiime 軟體 插件為 tools 的 import 函式
--type : 16S 定序主流以 PairedEnd 為主 [詳情]
--input-format : Phred33V2 是一種對序列品質分數表示格式
--input-path : manifest.tsv 的所在路徑
--output-path : 轉換產出的.qza的檔名與路徑
實務上,定序檔案若是來自文獻、廠商,
回到手上時多已拆分 (demultiplex) 完畢,
所以跳過拆分步驟 (Barcode部分我習慣在QC部分處理),
以demux.qza命名,如有需要可以看這裡。
完成後會顯示 :
"Imported manifest.tsv as PairedEndFastqManifestPhred33V2 to demux.qza"
接下來我們將迎來第一個視覺化資料 :
剛剛得到 demux.qza 轉為 .qzv,
輸入 :
qiime demux summarize \
--i-data demux.qza \
--o-visualization demux.qzv
完成後會顯示 :
"Saved Visualization to: demux.qzv"
將demux.qzv 下載後拉到 QIIME VIEW 網站的框框,
就會跑出漂漂的 data ~ QIIME VIEW 可以直接加進我的最愛,之後會超常用到!
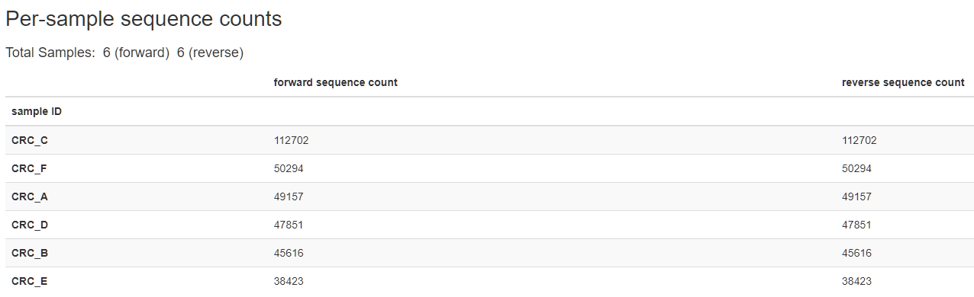
在這裡可以看到所分析的檔案名稱、數量與條數,
因為定序時來回讀取,資料上會有 Forward 與 Reverse,
通常會相同,因為我還沒見過不同的 XD
在這裡要做的是資料的確認,
看有沒有漏上傳或是打錯的名字。
這個頁面非常重要,
會影響到下一步要做的品質管制 (Quality Control),
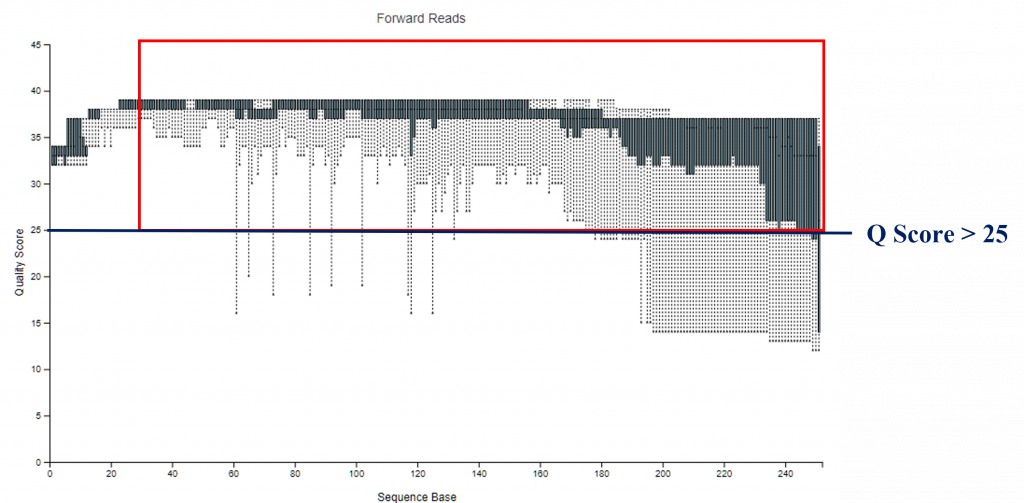
橫軸為每一條序列的長度,
根據 [Day 05] 我們知道,
序列長度取決於要 Primer 夾取哪個片段,
範例中所使用的夾取 V3-V4 片段的 Primer (可以從 Paper得知),
長度大約是 470 bp。
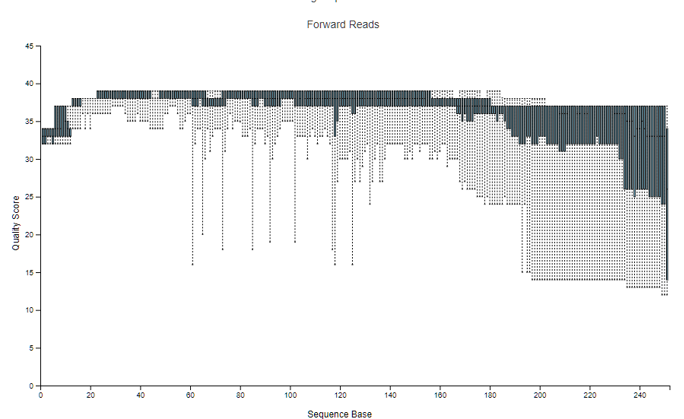
因為含有 adapters 緣故,,
加上兩條必須 overlapping 才能組裝,
所以單條序列長度會達到 250 bp,合起來會超過 470 bp (約 500 bp)。
因此在這圖中就可以看到橫軸長度約為 250 bp,
縱軸則為 Quality Score (Q Score) 品質分數,
代表著機器根據每個 mer 讀取到的螢光,
所給予的品質分數,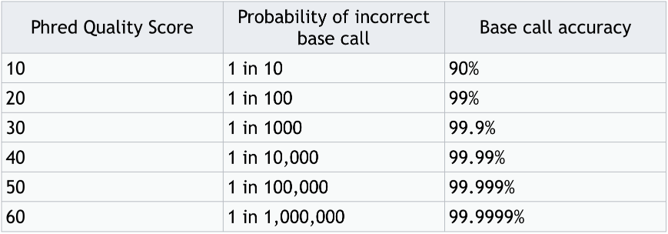
Reference : wikipedia
上面這張圖說明了 Q Score 分數的意涵,
以 Q Score 20 來說,
代表有 99% 肯定這個 mer 是 A/T/C/G,
在次世代定序中 Q Score > 25 就不錯了!
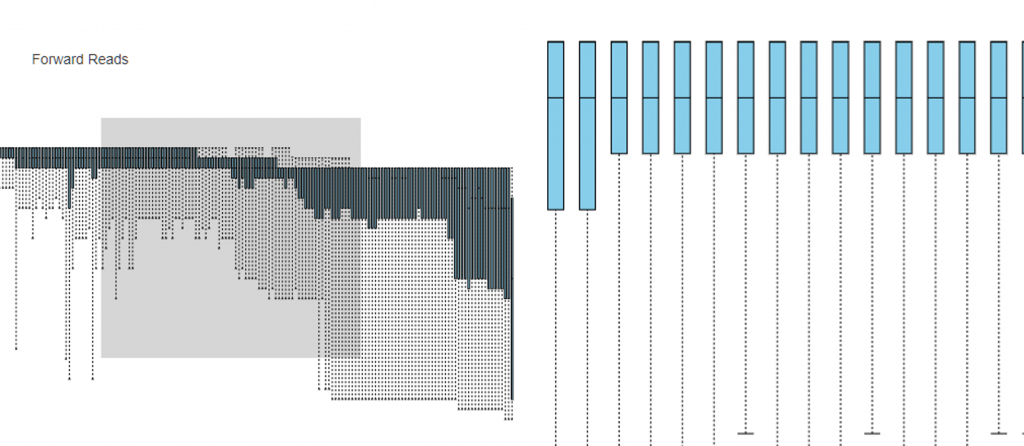
還可以將圖框起來放大特定區域(左),
會發現其實每個 base 都是一根根的四分位距圖(右),
每個 base 都是根據所有序列條數 (sequence counts) 品質所繪製出來的結果 !
如果有些 base 的 Q Score < 25 怎麼辦呢?
這通常會發生在序列的一開始與結束,
這時候就要決定要留多少的nt,進行後續分析。
生科人如果曾經送過 Sanger 定序確認基因重組 / Primer夾取結果,
像是加 His Tag, Plasmid 或是切膠定序確認,
報告中會看到定序剛開始與結束訊號的不穩定,
導致訊號混亂,就會看到這種圖案 :
直到中間序列才會逐漸穩定 :
次世代定序也是如此,頭尾的訊號也會相較中段不穩定,
因此需要進行修剪。
Reference
以範例來說,我習慣選擇 :
本篇使用到的輸入/輸出檔案 :Input : .fastq.gz、manifest.tsv、sample-metadata.tsvOutput: demux.qza、demux.qzv
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
