品質管制 (Quality control) 是序列分析重要的一環,
為了有系統地留下好序列,DADA2 這個套件包被發明了出來,
將 QC 分為修剪 (trimming)、過濾 (filters)、降躁 (denoises)、合併 (merge)、去除重疊 (dereplicates),
而 QIIME2 則收錄了 DADA2 讓一切變得更容易。
還記得在 [Day 07] 中我們獲得了一張便條紙,
上頭寫著每條序列都只留下 10~250 nt 片段,
因此接下來指令中會需要修剪序列。
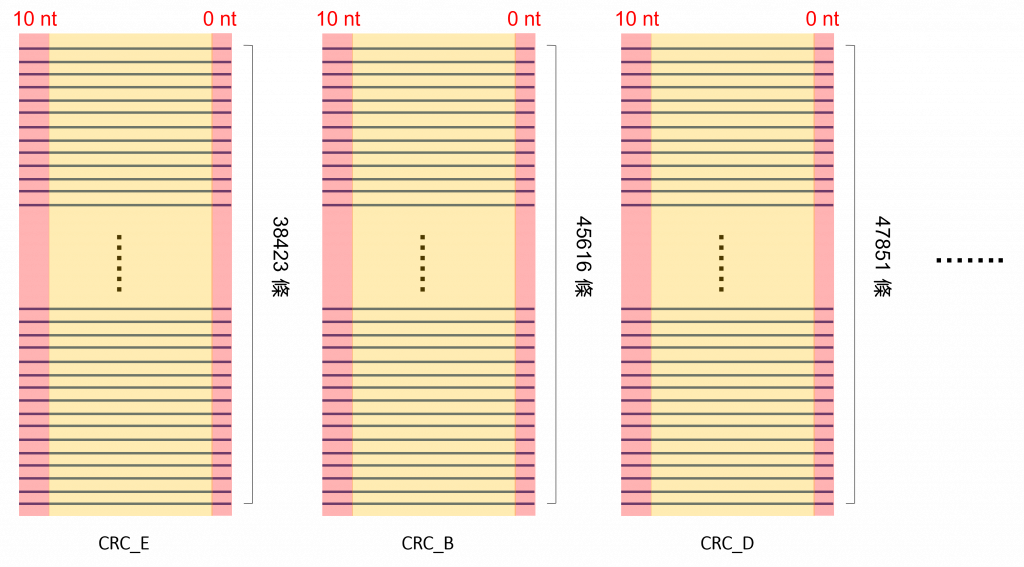
上圖以範例其中三個樣本為例,紅色就是要被修剪的位置,黃色則是留下的序列們。
本次範例因為末端品質還是蠻好的,就沒切了,實務上很多後面都不佳。
然而,每個樣本中都有上萬條序列,
裡頭也可能有品質不佳、為了合併而重疊的序列,
就需要過濾、降躁、合併、去除重疊,
原本的序列們就會像是經過漏斗層層篩選,
最後就會剩下好的序列。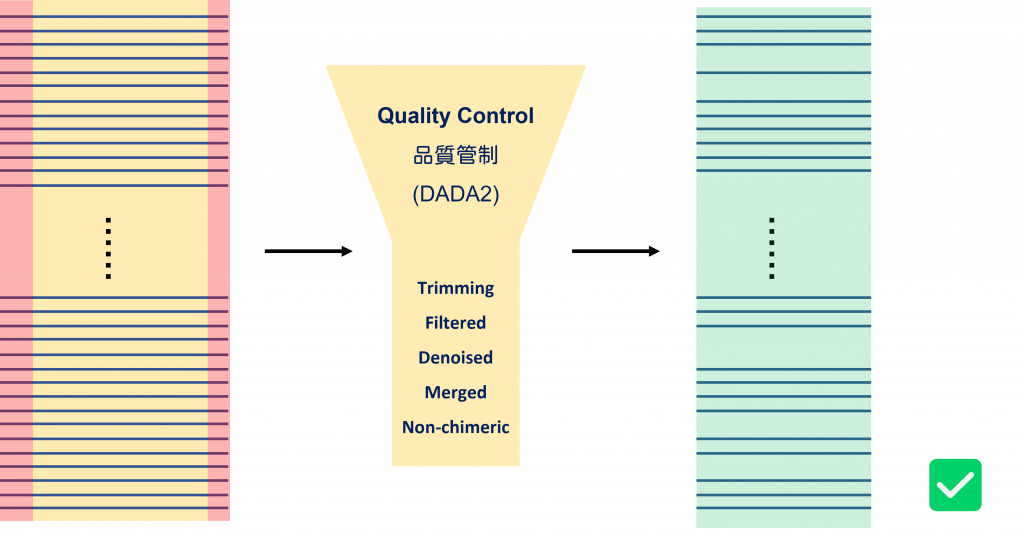
上圖經過 QC 之後,紅色被修剪,並去除品質不好序列,留下品質好的序列。
(注意看藍色一條一條的序列變少了)
記得先啟動 qiime2-2022.8 環境
conda activate qiime2-2022.8
將 [Day 07] 獲得的 demux.qza 拎來處理處理 :
qiime dada2 denoise-paired \
--i-demultiplexed-seqs demux.qza \
--p-trim-left-f 10 \
--p-trim-left-r 10 \
--p-trunc-len-f 240 \
--p-trunc-len-r 240 \
--o-table table-dada2.qza \
--o-representative-sequences rep-seqs-dada2.qza \
--o-denoising-stats stats-dada2.qza \
--p-n-threads 8
qiime dada2 denoise-paired : 使用 qiime 軟體 插件為 dada2 的 denoise-paired 函式
--i-demultiplexed-seqs : 輸入的檔案
--p-trim-left-f / --p-trim-left-r :Forward/Reverse 序列起始端切多少,範例是 10 bp
--p-trunc-len-f / --p-trunc-len-r :Forward/Reverse 序列剪切後的總長度,範例末端切 0 bp,所以 240 ~
--o-table : 輸出的檔案
--o-representative-sequences : 輸出的檔案
--o-denoising-stats : 輸出的檔案
--p-n-threads : 要運算的核心數,因設備而異,不清楚可不加這行。調整為 0 就是全力跑╰(°▽°)╯
Reference
完成後會顯示 :
'
Saved FeatureTable[Frequency] to: table-dada2.qza
Saved FeatureData[Sequence] to: rep-seqs-dada2.qza
Saved SampleData[DADA2Stats] to: stats-dada2.qza
'
輸出產生的檔案除了會做為之後分析的輸入檔案外,
也可以做視覺化輸換 (.qzv)。
輸入 :
qiime feature-table summarize \
--i-table table-dada2.qza \
--o-visualization table-dada2.qzv \
--m-sample-metadata-file sample-metadata.tsv
qiime feature-table tabulate-seqs \
--i-data rep-seqs-dada2.qza \
--o-visualization rep-seqs-dada2.qzv
qiime metadata tabulate \
--m-input-file stats-dada2.qza \
--o-visualization stats-dada2.qzv
完成後會顯示 :
"Saved Visualization to: table-dada2.qzv"
"Saved Visualization to: rep-seqs-dada2.qzv"
"Saved Visualization to: stats-dada2.qzv"
table-dada2 與 rep-seqs-dada2 兩檔案,
會在後續介紹到,
我們先來提提常觀察的 stats-dada2 :
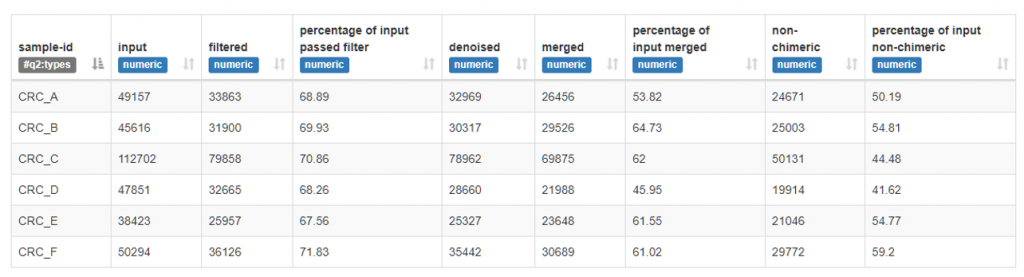
輸入 Input
一開始的尚未進行任何處理的序列總數。
過濾 Filter
目的 : 移除有太多低品質(Quality score) mer 出現的序列 。
The maxEE parameter sets the maximum number of “expected errors” allowed in a read.
可以想成累加錯誤值,會針對每條序列的每個 mer Quality score 換算為分數進行累加,
Quality score 越低,分數則越高,
舉例來說(分數僅示意),
若 Quality score 分別為 10, 20, 10, 20, 30...
分數則為 0.1 + 0.01 + 0.1 + 0.01 + 0.001 + ... + ,
整條序列若累積的錯誤分數達到閾值(預設為2),則該條序列就會被拋棄。
降噪 Denoised
目的 : 移除PCR放大與定序過程中出錯的序列
像是聚合酶(Polymerase)偷懶跳過、引子(Primer)亂亂黏,
或是溫度條件不穩造成的循環(Cycle)未完成即到下一個循環等,
都會導致錯誤的序列被製造與放大,會在這個步驟透過演算法篩選。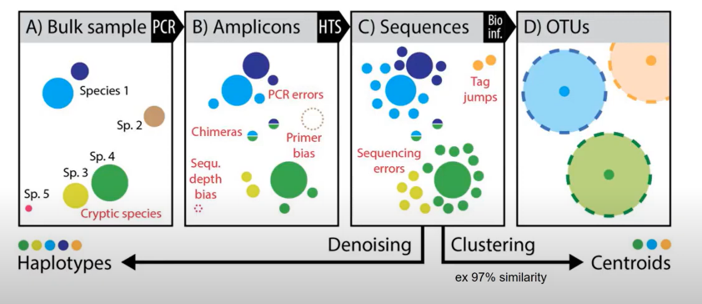
Reference : QIIME2 Youtube
可以發現在擴增(Amplicons)、定序(Sequences)過程 (A~C),
會產生各類的錯誤,導致序列與現實有所差距,
透過 Denoising 演算法還原真實情況。
合併 Merged
目的 : 移除 Forward、Reverse 合併時組不起來的序列
[Day 05] 中提及雙尾 (pair-end)是藉由兩段序列組裝成帶有 V3-V4 區的序列,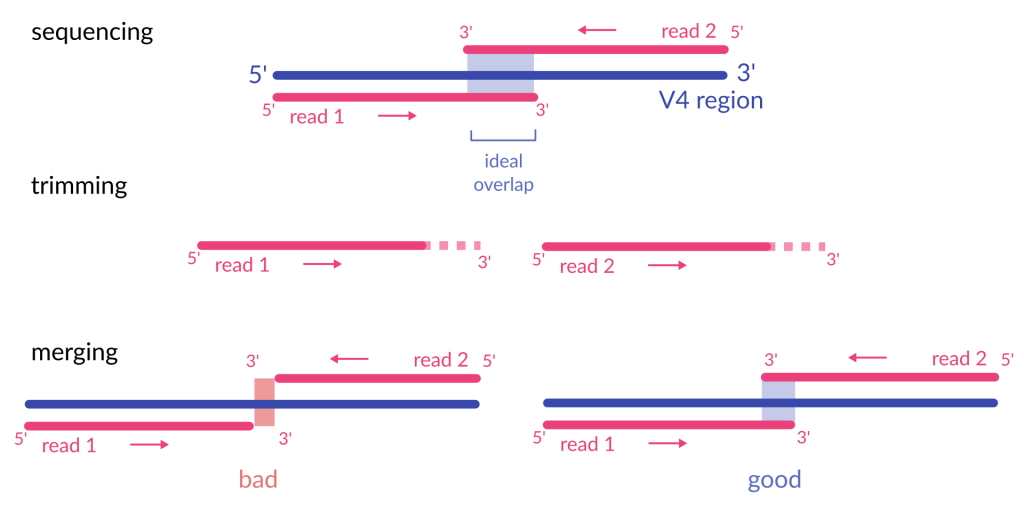
Reference : ISB Microbiome Course 2020
若兩段序列的重疊區域因為各種原因,如品質過低、剪切過多(如圖示 trimming),
都會造成序列合併時找不到另一半,找不到另一半的序列就會被淘汰 (bad) QQ
非嵌合體 Non-Chimeric
目的 : 移除因為PCR 溫度條件出現的嵌合體序列
Reference : ISB Microbiome Course 2020
在 PCR 擴增 (Amplicons) 過程中,每一個循環的溫度相當重要,
如果可愛的聚合酶在複製的過程中,
PCR 機溫度或是其微環境頑皮了,導致聚合酶來不及複製完 (圖示 B),
就會出現大大大大大 Primer (圖示 C),
並在下一個循環中製造出嵌合體(chimera) (圖示 D)。
經歷層層篩選後的序列,
就會成為後續分析的主力,
剩下被移除的序列就會消失在這地球上。
上述每一步是經由一系列演算法才得以達成,有興趣可以閱讀這篇 Medium,
也歡迎拜讀開發者 (Callahan, Benjamin J., et al., 2016),這邊以好讀版為主。
本篇使用到的輸入/輸出檔案 :Input : demux.qzaOutput: table-dada2.qza、rep-seqs-dada2.qza、stats-dada2.qza、table-dada2.qzv、rep-seqs-dada2.qzv、stats-dada2.qzv
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。

第一個指令中的
“--p-trim-left-f / --p-trim-left-r : Forward/Reverse 序列起始端切多少,範例是 30 bp”--> 示範指令應該是 10 bp?
是10 bp,已更正,感謝~~