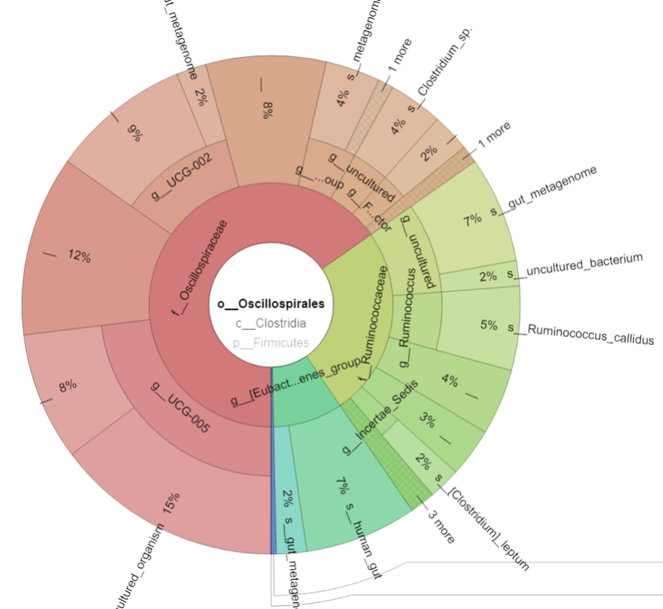
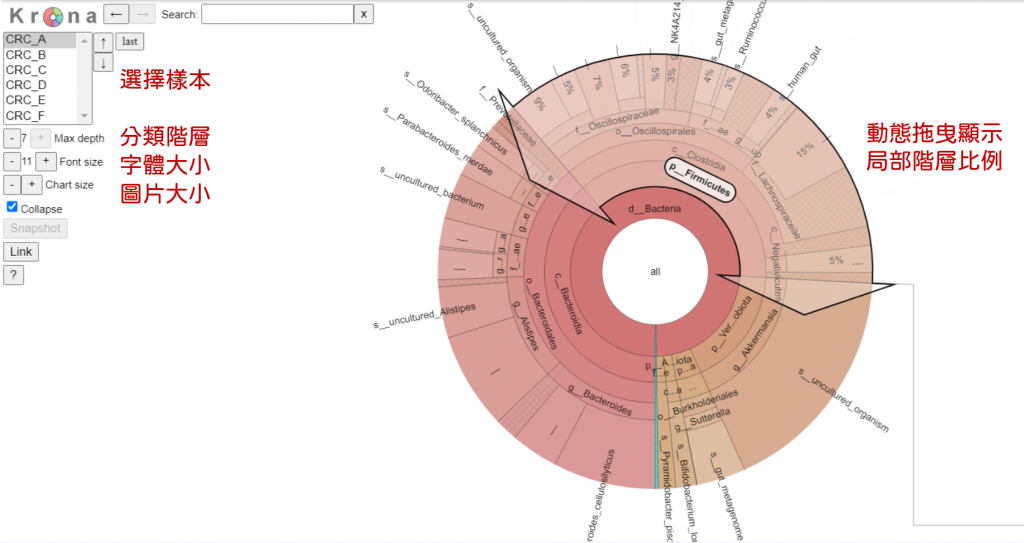
Krona 互動式多層圓餅圖
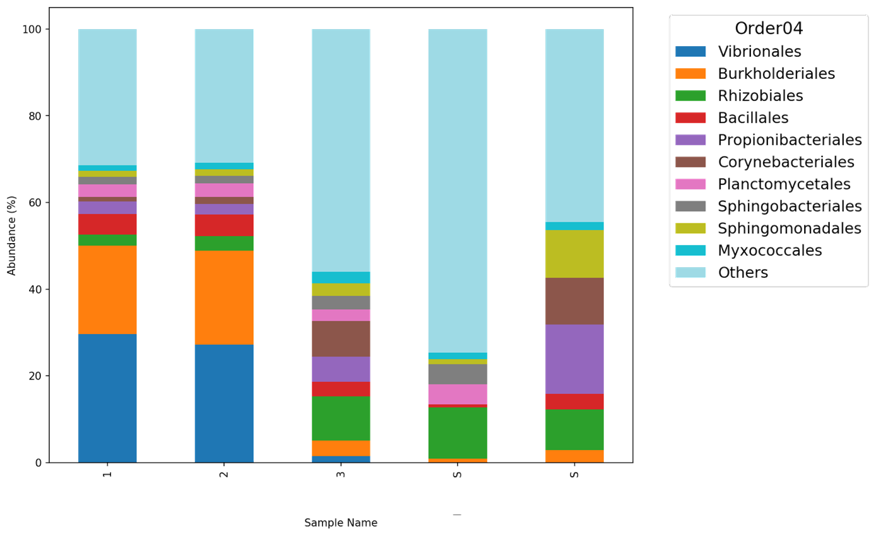
Top N分類柱狀圖
| 視覺化類型 | 環境條件 | 適合 | 優點 | 缺點 |
|---|
分類柱狀圖 (Taxonomy Bar Plot) [Day 11] 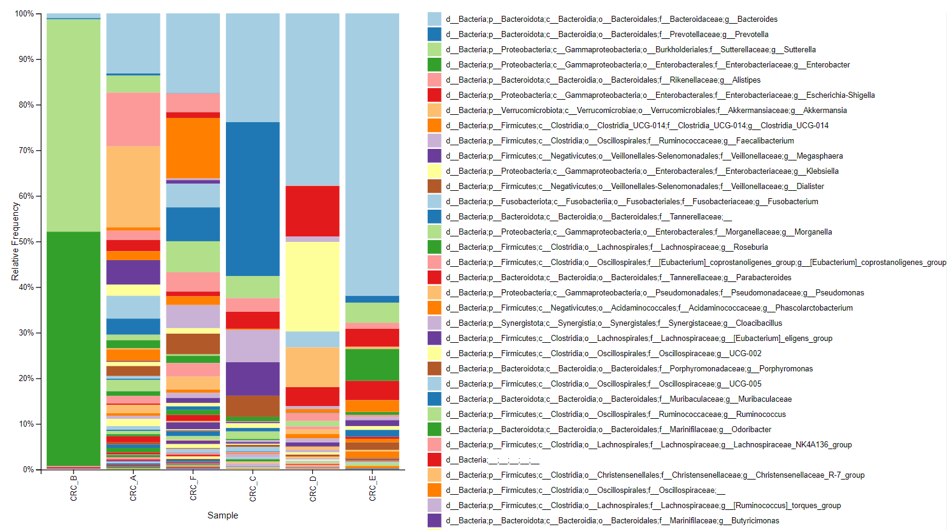 | QIIME2 內建 | 全部顯示、快速分析瀏覽分類器結果 | 篩選 (Screening) 共同菌種方便、檢視分類效果(Unassign 與 Uncultured 易觀察) | 無法將其他菌種歸為 Other,僅顯示前幾菌種、無法輕易更改樣本名稱
| QIIME2 內建 | 全部顯示、快速分析瀏覽分類器結果 | 篩選 (Screening) 共同菌種方便、檢視分類效果(Unassign 與 Uncultured 易觀察) | 無法將其他菌種歸為 Other,僅顯示前幾菌種、無法輕易更改樣本名稱
Krona 互動式多層圓餅圖  | QIIME2 插件 (需要多幾個指令安裝擴充) | 檢視各分類階層、特定菌階層下物種比例、 | 可調整階層、字體、圖片大小、比例可依階層繪製、超級漂亮炫炮 | 菌名都很長,動畫很多,不小心容易迷路
| QIIME2 插件 (需要多幾個指令安裝擴充) | 檢視各分類階層、特定菌階層下物種比例、 | 可調整階層、字體、圖片大小、比例可依階層繪製、超級漂亮炫炮 | 菌名都很長,動畫很多,不小心容易迷路
Top N 分類柱狀圖  | Python IDE (Spyder or Jupyter) | 檢視主流(Dominant)菌 | 顯示前2~20名菌種、解決分類柱狀圖缺點、顏色可調整、菌名短(僅顯示該階層名稱) | 需要基礎 Python 知識、分組情形目前無納入
| Python IDE (Spyder or Jupyter) | 檢視主流(Dominant)菌 | 顯示前2~20名菌種、解決分類柱狀圖缺點、顏色可調整、菌名短(僅顯示該階層名稱) | 需要基礎 Python 知識、分組情形目前無納入
先進入 qiime2-2022.8 環境,
安裝依賴項(dependencies),按Y :
conda install -c bioconda krona
完成後會顯示 :
'
Preparing transaction: done
Verifying transaction: done
Executing transaction: /
Krona installed. You still need to manually update the taxonomy
databases before Krona can generate taxonomic reports. The update
script is ktUpdateTaxonomy.sh. The default location for storing
taxonomic databases is /home/XXXXX/envs/qiime2-2022.8/opt/krona/taxonomy
'
安裝插件 : Reference : kaanb93
pip install git+https://github.com/kaanb93/q2-krona.git
完成後會顯示 :
'Successfully' built q2-krona
Installing collected packages: q2-krona
'Successfully' installed q2-krona-1.0.2
以上只有第一次安裝需要,之後就可以從以下執行 Krona 作圖 :
所需要的檔案包含 table-dada2-240.qza、taxonomy.qza [Day 11],
qiime krona collapse-and-plot \
--i-table table-dada2-240.qza \
--i-taxonomy taxonomy.qza \
--o-krona-plot krona.qzv
完成後會顯示 :
'Saved Visualization to: krona.qzv'
同樣地,下載krona.qzv後拖曳到 QIIME2 VIEW :
前面表格有提到,
由於 QIIME2 分類柱狀圖與 Krona 無法將其他菌種歸為 Other,
僅顯示前幾菌種(如Top10/Top20),
許久以前我寫了 Python 小程式試圖解決這個問題,
Windows / Mac 需安裝 Git, Python IDE (Spyder or Jupyter) 方邊操作 。
網路上很多上述安裝教學。
準備輸入檔案 level-7.csv
將先前的taxa-bar-plots.qzv 拖曳入 QIIME2 VIEW,
Taxonomic Level 欄位選擇 Level 7,並點擊左側的 Download csv 檔案 :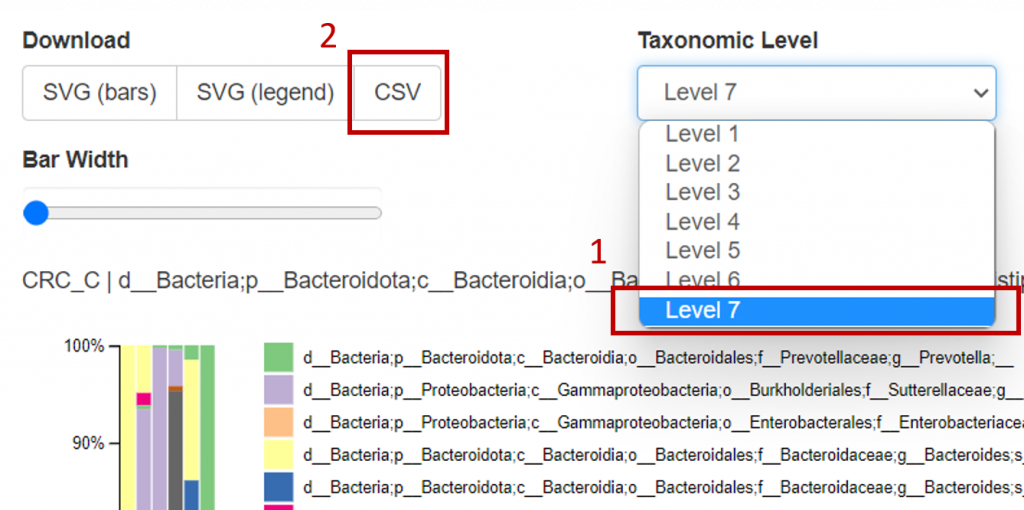
會獲得 level-7.csv 。
下載 NTGS 分類柱狀圖程式在 Windows / Mac cmd 執行
git clone https://github.com/ba2c7yoyo/NTGS
以 Spyder IDE 為例
開啟資料夾內的TopN_abundance_NGS.py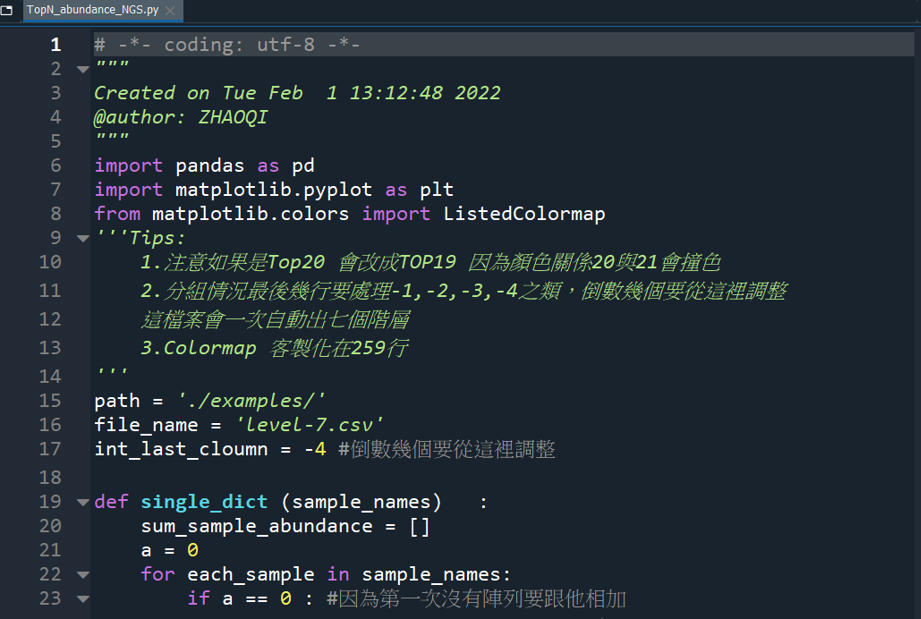
若無安裝過 pandas matplotlib 則安裝 :
pip install pandas matplotlib
TopN_abundance_NGS.py有三個參數需要調整,Line 15~17 :
path = './examples/'
#剛下載 level-7.csv 的路徑位置
file_name = 'level-7.csv'
#剛下載 level-7.csv 的檔名
int_last_cloumn = -2
#sample-metadata.tsv 內的分組數,若依照本篇文範例調整為 -2 (Index 與 Sex)
執行程式後,會詢問要繪製哪些樣本、檔名、前幾名,
Top 建議在2~20之間,因為預設顏色最多二十種(含 Other ) :
(File name 預設無作用,可參閱文件儲存檔案)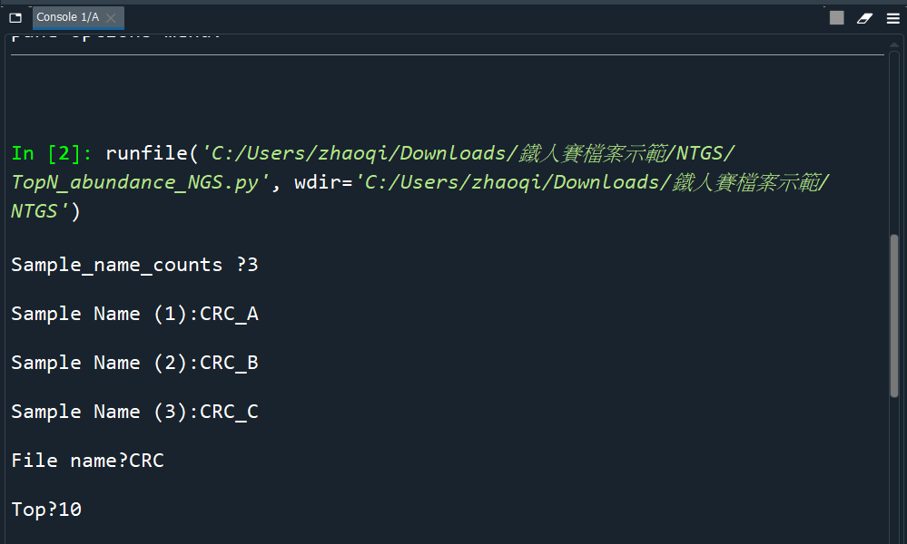
在程式內會產出各階層 Top N結果圖 :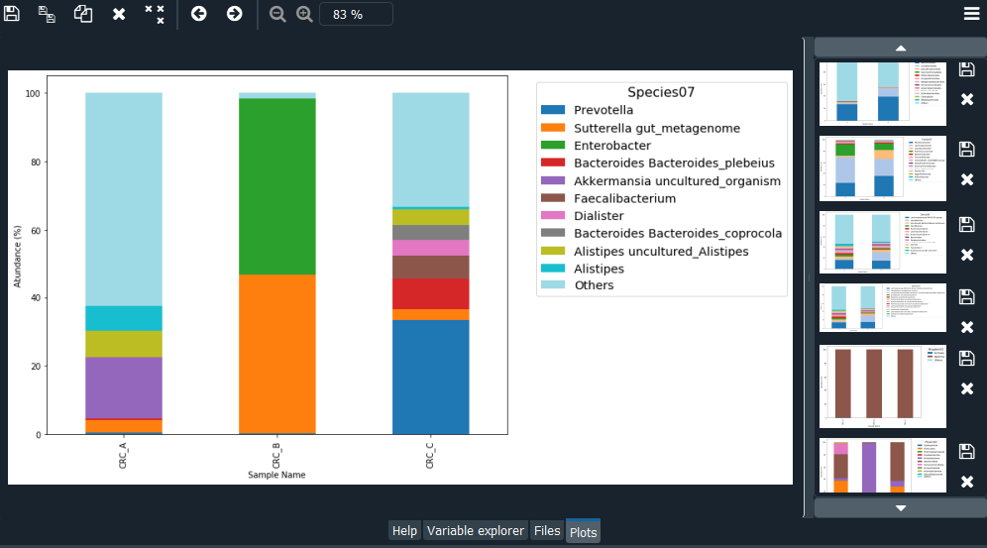
這個程式是我剛學 Python 寫的,變數命名其實蠻混亂,
在設計邏輯上用了一堆字典形式當作練習 XD,
稱不上是一個好讀的程式,但因為好用就一直用到現在,
開源的程式碼,歡迎各位前輩 Fork 改良,
其實還有一些進階功能,比如改變顏色、儲存檔案,
詳見 ba2c7yoyo on GitHub (作者是自己w)
各種漂漂圖就這樣出來啦~
Top 20 Species level :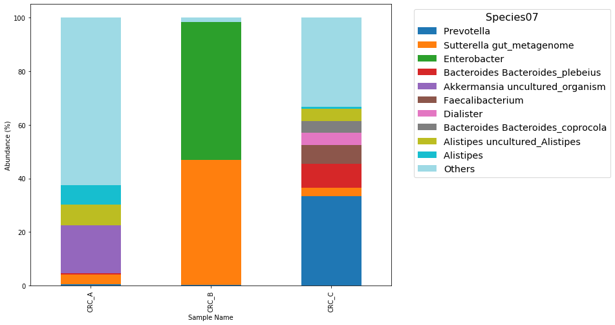
Top 15 Class level (從這裡可以發現 CRC_A 及 CRC_C 都有 Clostridia 菌) :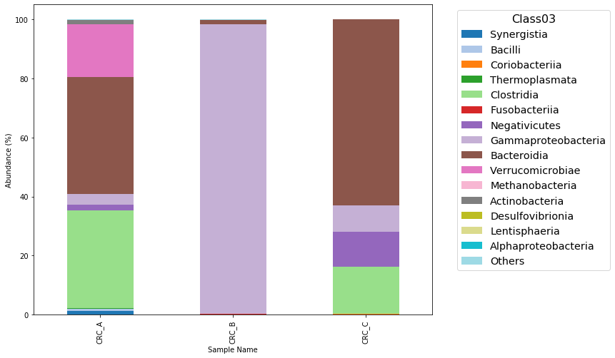
本篇使用到的輸入/輸出檔案 :Input : table-dada2-240.qza、taxonomy taxonomy.qza、taxa-bar-plots.qzvOutput: krona.qzv、level-7.csv
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
