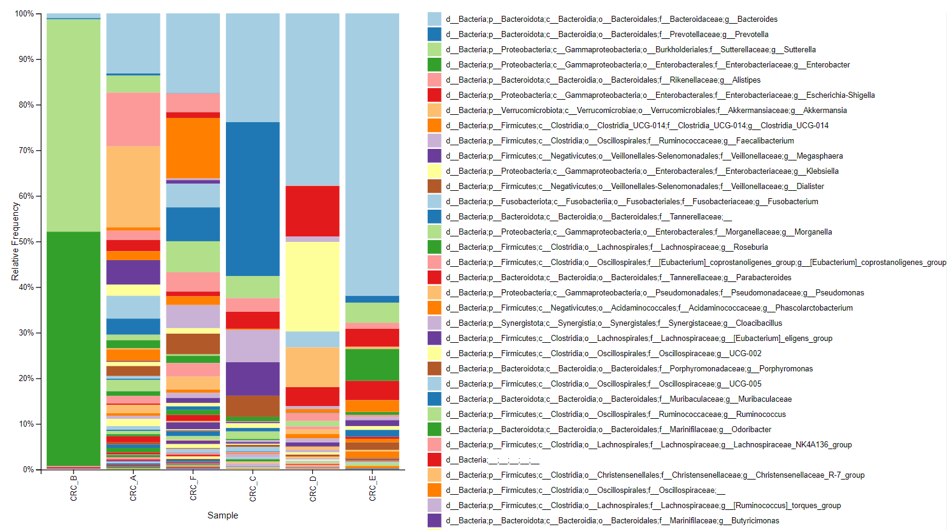
分類柱狀圖(Taxonomy bar plot)可以幫助研究者一窺樣本的物種分布,
QIIME2 VIEW 提供的互動式介面可以更改顏色、排序、尺存以及強調標示特定物種。
如 [Day 10] 所說,
先將已訓練好的 Silva 138 V3-V4 分類器silva138_AB_V3-V4_classifier.qza放至 Linux 目錄中。
Reference : anweshmaile
使用以下指令,讓分類器註釋我們手上的資料 :
qiime feature-classifier classify-sklearn \
--i-classifier silva138_AB_V3-V4_classifier.qza \
--i-reads rep-seqs-dada2-240.qza \
--o-classification taxonomy.qza
sklearn 是機器學習廣泛使用的套件,
有興趣可以參考 queenawu 大大的鐵人賽作品。
完成後會顯示 :
'
Saved FeatureData[Taxonomy] to: taxonomy.qza
'
qiime metadata tabulate \
--m-input-file taxonomy.qza \
--o-visualization taxonomy.qzv
完成後會顯示 :
'
Saved Visualization to: taxonomy.qzv
'
taxonomy.qzv扔到 QIIME2 VIEW 檢視 :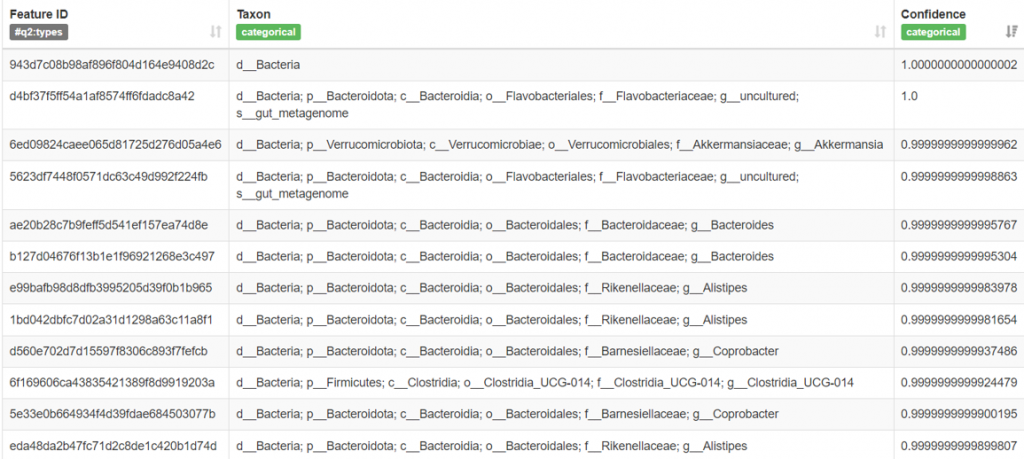
可以在這裡檢視分類器的效果如何,
就想像是分類器小朋友繳交的答案卷,
Taxon 呈現各階層的結果 (界d 門p 綱c 目o 科f 屬g 種s)
(在Greengene 資料庫則以 D_0, D_1, D_2, D_3 .......數字呈現階層)。
若是出現許多顯示 Unassigned 或多數無法到 G (Genus) / S (Species),
就要考慮分類器是否合適。
仔細觀察開頭的柱狀圖,
發現可能需要樣本名稱sample-metadataa.tsv、剛剛的分類結果等資料,
如果是照著做下來,這些都之前都已經有了~
qiime taxa barplot \
--i-table table-dada2-240.qza \
--i-taxonomy taxonomy.qza \
--m-metadata-file sample-metadata.tsv \
--o-visualization taxa-bar-plots.qzv
完成後會顯示 :
'
Saved Visualization to: taxa-bar-plots.qzv
'
taxa-bar-plots.qzv扔到 QIIME2 VIEW 檢視 :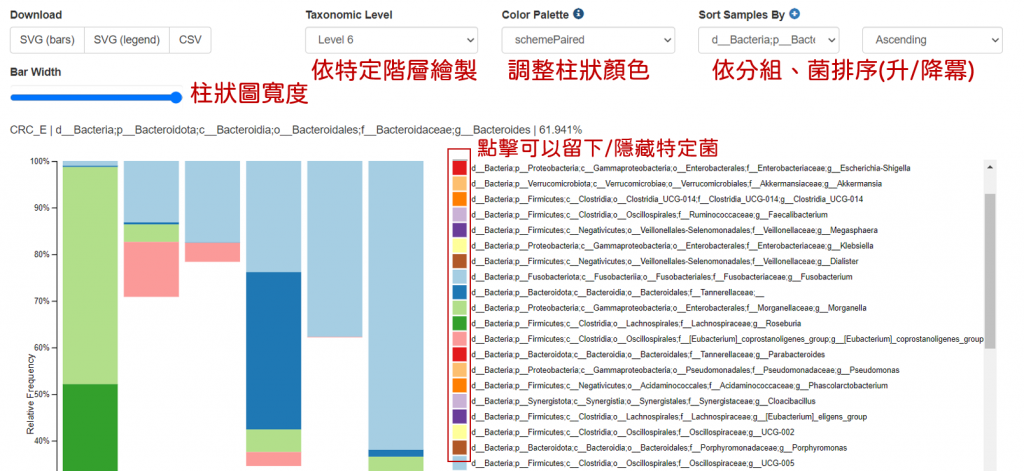
習慣上會將 Bar width 調到最寬,階層會先看5~7,
顏色上有個小缺點 : 每十個菌顏色就會開始重複,
如果想要自行繪製可以在左上角下載表格。
調整好後就回得到一張漂亮的圖啦~
本篇使用到的輸入/輸出檔案 :Input : silva138_AB_V3-V4_classifier.qza、rep-seqs-dada2-240、table-dada2-240.qza、sample-metadata.tsv、taxonomy.qzaOutput: taxonomy.qza、taxonomy.qzv、taxa-bar-plots.qzv
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
