還記得 [Day 01] 放了一張酷酷的親緣關係樹嗎?
今天要來繪製它!
Rooted Phylogenetic Tree
做生物資訊成就感之一就是看著一張張美美的圖產出。
親緣關係樹是藉由序列與序列之間的比對 (alignment) 得出,
利用分子生物學角度繪製物種與物種之間的「距離」,
蠻推薦從未手繪一棵「樹」的同學,
可以修習關於序列比對的課程(通常出沒在生物資訊學),
在檢視結果時會更有概念。
兩者使用時機依想解決的研究問題而定,
有根樹因為所有序列的上游皆源自於一點,
具有「追本溯源」的特性,帶有演化的方向性,
適合應用在觀察演化的過程與關係。
Rooted Phylogenetic Tree,原來是迷宮阿,還以為是演化樹。
無根樹則無定義出演化的方向與路徑,
著重在分類群之間的關係(relationships among the taxa),
適合樣本內菌相差異極大(multispecies coalescent phylogenetic framework)的觀察。
(Kinene, Tonny, et al., 2016)

Unrooted Phylogenetic Tree
進入 qiime2-2022.8 環境後輸入 :
qiime phylogeny align-to-tree-mafft-fasttree \
--i-sequences rep-seqs-dada2-240.qza \
--o-alignment aligned-rep-seqs-240.qza \
--o-masked-alignment masked-aligned-rep-seqs.qza \
--o-tree unrooted-tree.qza \
--o-rooted-tree rooted-tree.qza
--o-masked-alignment 是在產出樹的過程中,需遮罩(mask)一些高度變異的序列避免造成過多噪點。
完成後會顯示 :
'
Saved FeatureData[AlignedSequence] to: aligned-rep-seqs-240.qza
Saved FeatureData[AlignedSequence] to: masked-aligned-rep-seqs.qza
Saved Phylogeny[Unrooted] to: unrooted-tree.qza
Saved Phylogeny[Rooted] to: rooted-tree.qza
'
完成後主要會使用到aligned-rep-seqs-240.qza、unrooted-tree.qza 及 unrooted-tree.qza
QIIME2 目前尚未提供樹的視覺化於 VIEW,
於是這時候 iTOL 視覺化樹樹網就出場了! (?
因為他的功能過於強大,(這裡可以觀賞世界各地強者的美化樹樹結果),
這邊僅介紹常見功能~
taxonomy.qza : [Day 11] 分類器分析後的結果檔table-dada2-240.qza : [Day 08] 品質管制後的結果檔 (終於要用上了)aligned-rep-seqs-240.qza : [Day 13] 剛剛熱騰騰產出來的unrooted-tree.qza : [Day 13] 剛剛熱騰騰產出來的rooted-tree.qza : [Day 13] 剛剛熱騰騰產出來的愉快前往 iTOL 上傳檔案區,
上傳 rooted-tree.qza 或 unrooted-tree.qza 檔案 :
(這邊以 rooted-tree.qza 為例)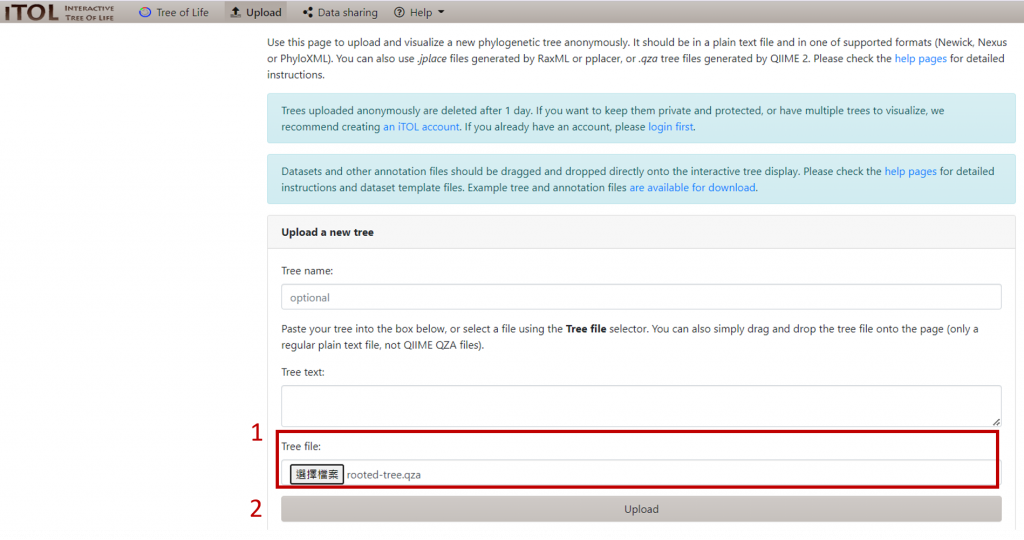
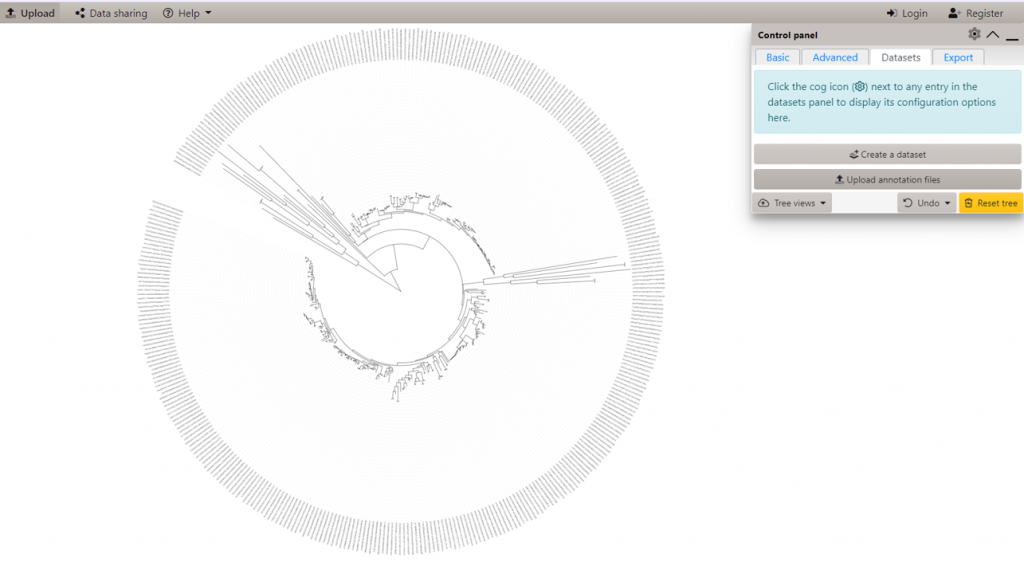
一棵圓形的樹就這麼長出來了,如此突然,
仔細看會發現都是以特徵ID(Feature ID)註釋
(差不多長這樣 311387e184d203ce7d2b0),
但人類根本看不懂,於是我們補上傳一些註釋資料(Annoation),
在右側的 Datasets 上傳註釋資料 taxonomy.qza: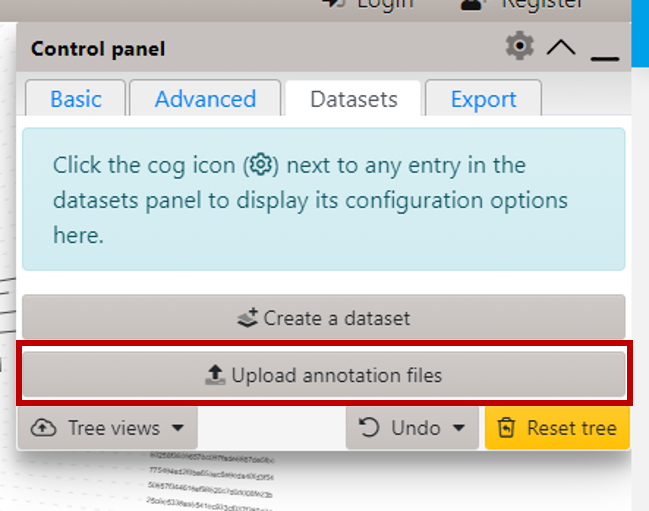
再上傳其他註釋資料 (上傳按鈕躲到最下面)aligned-rep-seqs-240.qza、table-dada2-240.qza :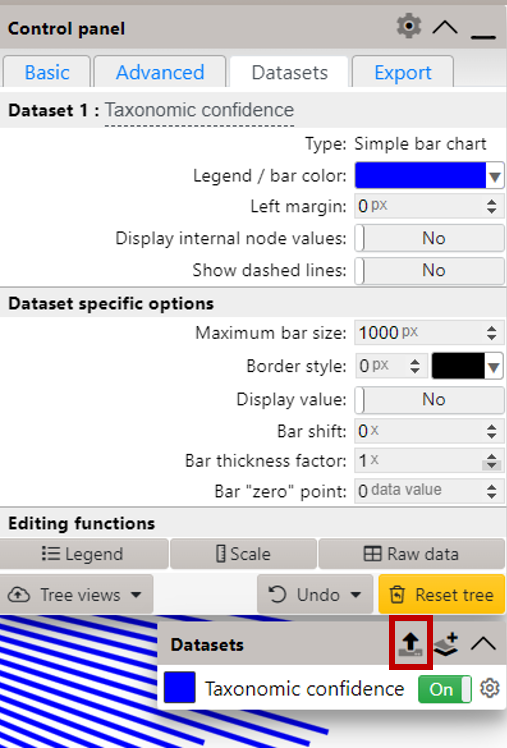
好了,這下已經非常繽紛了 :
taxonomy.qza : 將特徵ID(Feature ID) 置換為物種名稱,aligned-rep-seqs-240.qza : 顯示比對 (alignment) 中 mer 的差異table-dada2-240.qza : 顯示不同樣本的對該序列的條數,以長條圖表示iTOL 還提供三種呈現方式 Circular、Rectangular、Unrooted ,在右側控制板都能切換,
選項功能很多,試著切換來切換去很好玩,而登入後可以保存之前上傳的資料,
就能跑出以下漂漂的圖啦~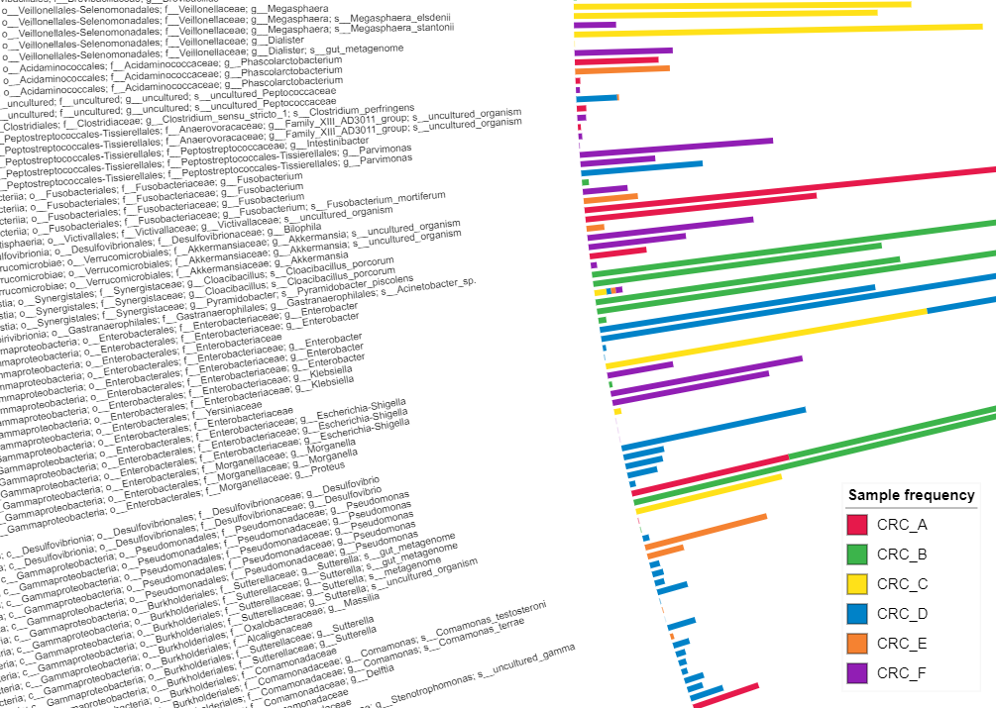
Circular / taxonomy.qza OFF / table-dada2-240.qza ON / Invert tree : Yes

Rectangular / taxonomy.qza : OFF / table-dada2-240.qza : ON / Invert tree : Yes
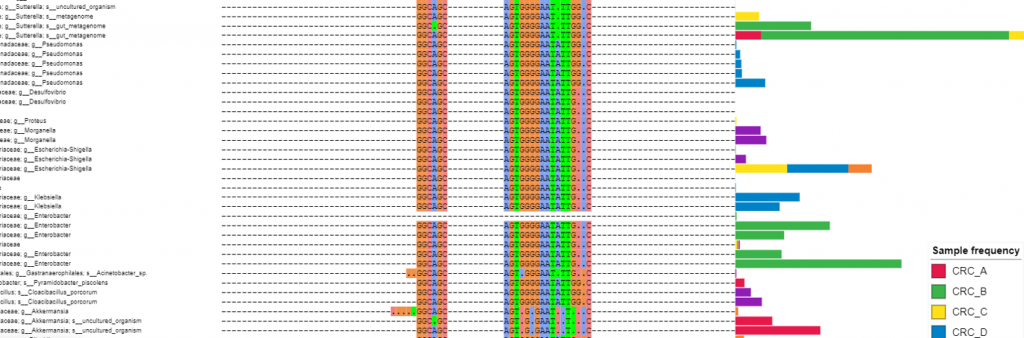
Rectangular / taxonomy.qza : ON / aligned-rep-seqs-240.qza : ON / Invert tree : NO
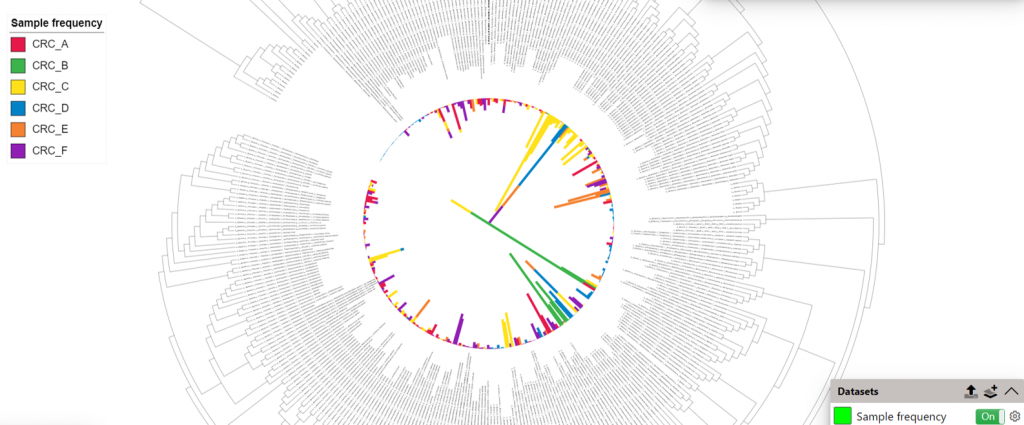
Circular / taxonomy.qza : OFF / table-dada2-240.qza : ON / Invert tree : Yes
實務上,我會拿來觀察極為相似序列的比對關係(aligned-rep-seqs-240.qza)、
未知物種的差異(taxonomy.qza),
還有豐富度的比較(table-dada2-240.qza)。
本篇使用到的檔案 :
rep-seqs-dada2-240.qza、taxonomy.qza、table-dada2-240.qza、aligned-rep-seqs-240.qza、masked-aligned-rep-seqs.qza、unrooted-tree.qza、rooted-tree.qza
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
