上一篇中已經簡單地說明基因本體論富集分析的概念,這次就來實際操作吧!

(截圖來自 Caterpillarplasty)
在開始之前補充一下,從基因列表提取出人類可以理解的詞彙的富集分析,整體流程包含前置作業與實際呈現其實分成三大步驟:

今天要做的就是評分的過程,註解的方法可以參考 Trinotate,而視覺化工具將留在下一篇介紹。

(截圖來自 Caterpillarplasty)
評分使用 Panther 網頁所提供的服務,Panther 有自己的一套註解系統,但是也可以支援 GO 系統的分析。
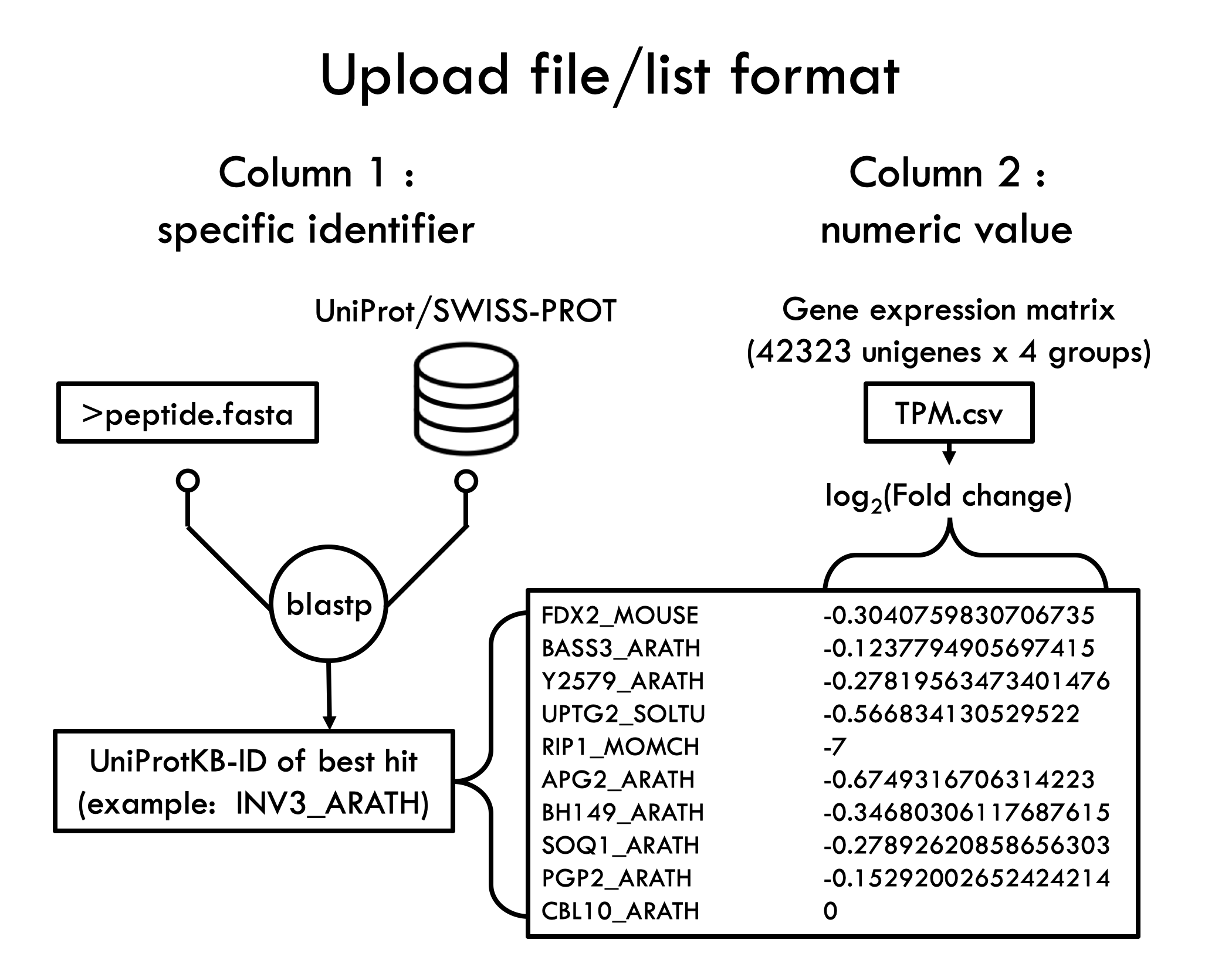
要上傳的檔案有既定的格式,如果手邊只有序列檔案的話,請參考 Trinotate 的流程來完成註解,也就是透過 blastp 取得每個序列最相似的 UniProt 之 ID。之後分成兩種分析,一種是 Overrepresentation test,另一種則是 Enrichment test。前者字面上的意思就是看看這個基因列表中誰存在感比較重,所以輸入只需要基因列表;後者 enrichment 不只是看存在感,還會看該基因差異表現的程度,因此需要輸入兩個 column 的 tsv 檔案,第二個 column 要提供一個我們所在意的關於該基因的數值,官方建議最簡單的方式就是採用 log2(Fold Change)。

輸入檔案,頁面下方有些許參數需要設定。記得勾選紅框中的指定分析項目,物種的部分請選擇一個最適合的,如果是非模式植物的話,可能想選的物種不會出現在其中,使用者可以選擇一個最接近的,但是記得這樣的話,所有不符合該物種的 UniProt ID 都會被捨棄掉,也就是結果僅供參考。

來到結果頁面,上方有一個選項可以選擇所要分析的註解系統,此處就可以選擇是官方的 GO 系統亦或是 Panther GO-Slim 系統,所謂的 Slim 指的是略為刪減過的系統,畢竟一個完整的本體論系統一定會有很多為了完善而必須加入的詞。除了 GO 以外也有 Panther Pathway 和 Reactome pathways 等等不同的系統可以選擇分析。

關於結果的解讀,由左至右分別是我們想要知道的富集的條目、我們選擇的提供背景值之物種該條目被註解了的基因數量、我們提供的基因列表中被註解為該條目的基因數量、期望應該要有的基因數量、富集的倍數、是 over or under-representation、p 值、和 False Discovery Rate。

(截圖來自 The Absence of Eddy Table)
明天我們將會示範將這一個輸出的表格視覺化的工具,請不吝指教與留言討論~
關於作者
謝晨 (Chen Hsieh),臺大園藝暨景觀學系研究所碩士。讀碩士前的興趣是懷著寫點程式妄圖解決農業問題的夢想參加比賽,拿了幾個黑客松與 Open Data 創新應用競賽的獎,卻都沒有勇氣將項目經營下去;研究所期間的興趣轉換成讀學術期刊的出刊電子報。靠著這些興趣當選 107 學年的臺大優秀青年,畢業後卻成了無業的實驗室居民。現在在農場旁的研究館辦公室寫點東西,希望可以跟世界分享生物資訊與園藝的樂趣!
感謝選擇匿名的朋友協助校閱初稿與提供意見,也敬請各位讀者不吝指教!
