基因本體論富集分析,由基因名稱列表濃縮成人類可以理解的詞彙,可以說是一種轉化的過程。
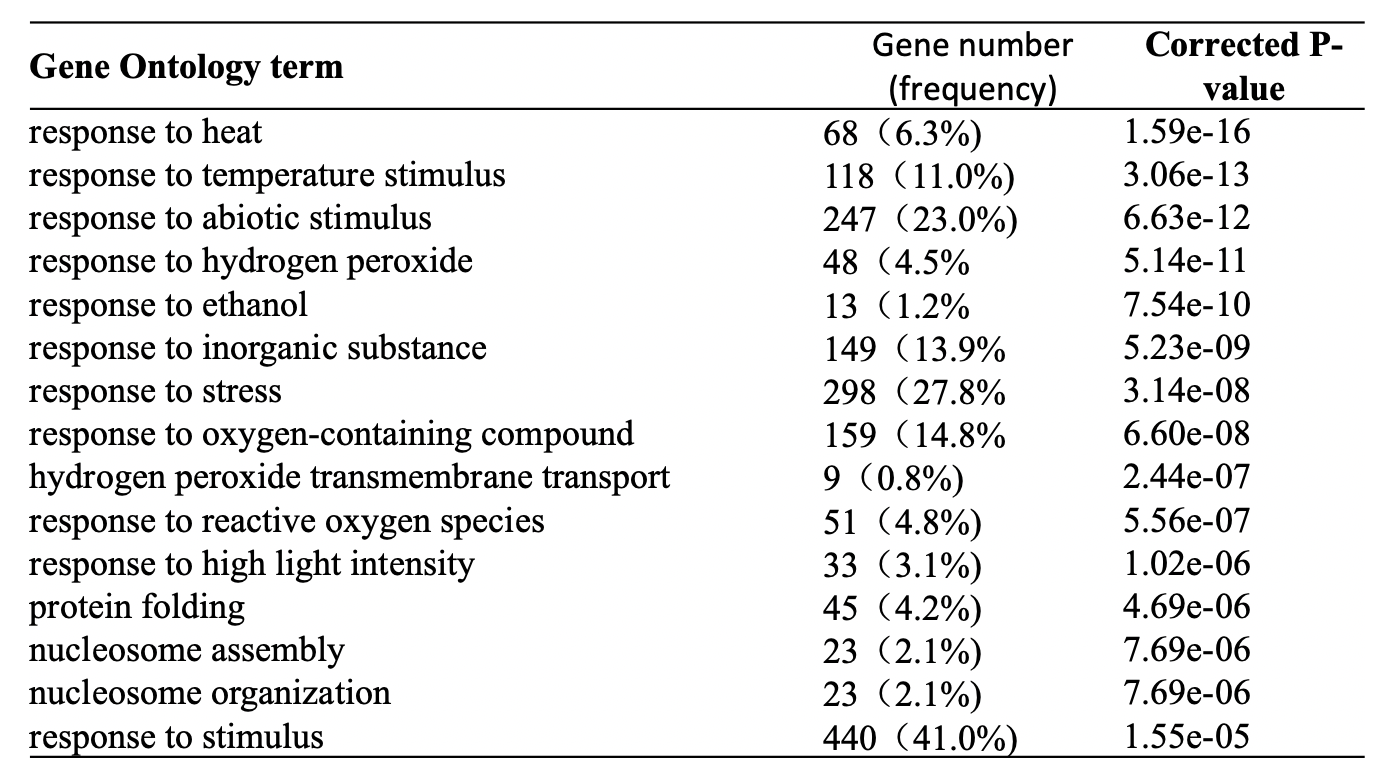
但是這個結果還可以進一步濃縮,由詞彙與輸出數值的表格再近一步轉化成為視覺化的結果。期刊論文上常見的視覺化方向可以分成下列幾種風格:著重詞彙所對應的數值,詞彙展開排排站,長條圖表示他們對應的數值,比如 p-value 或基因數量
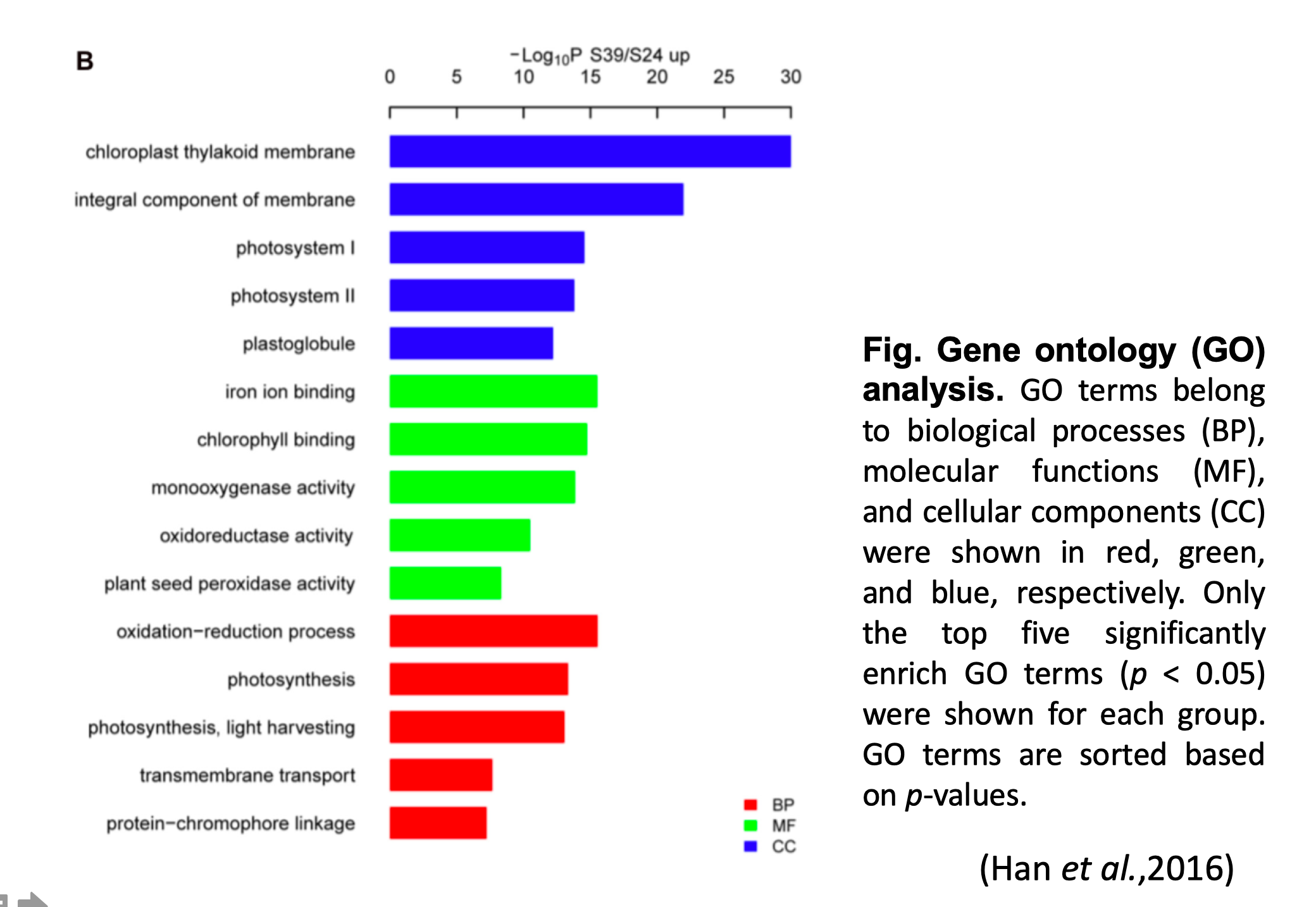
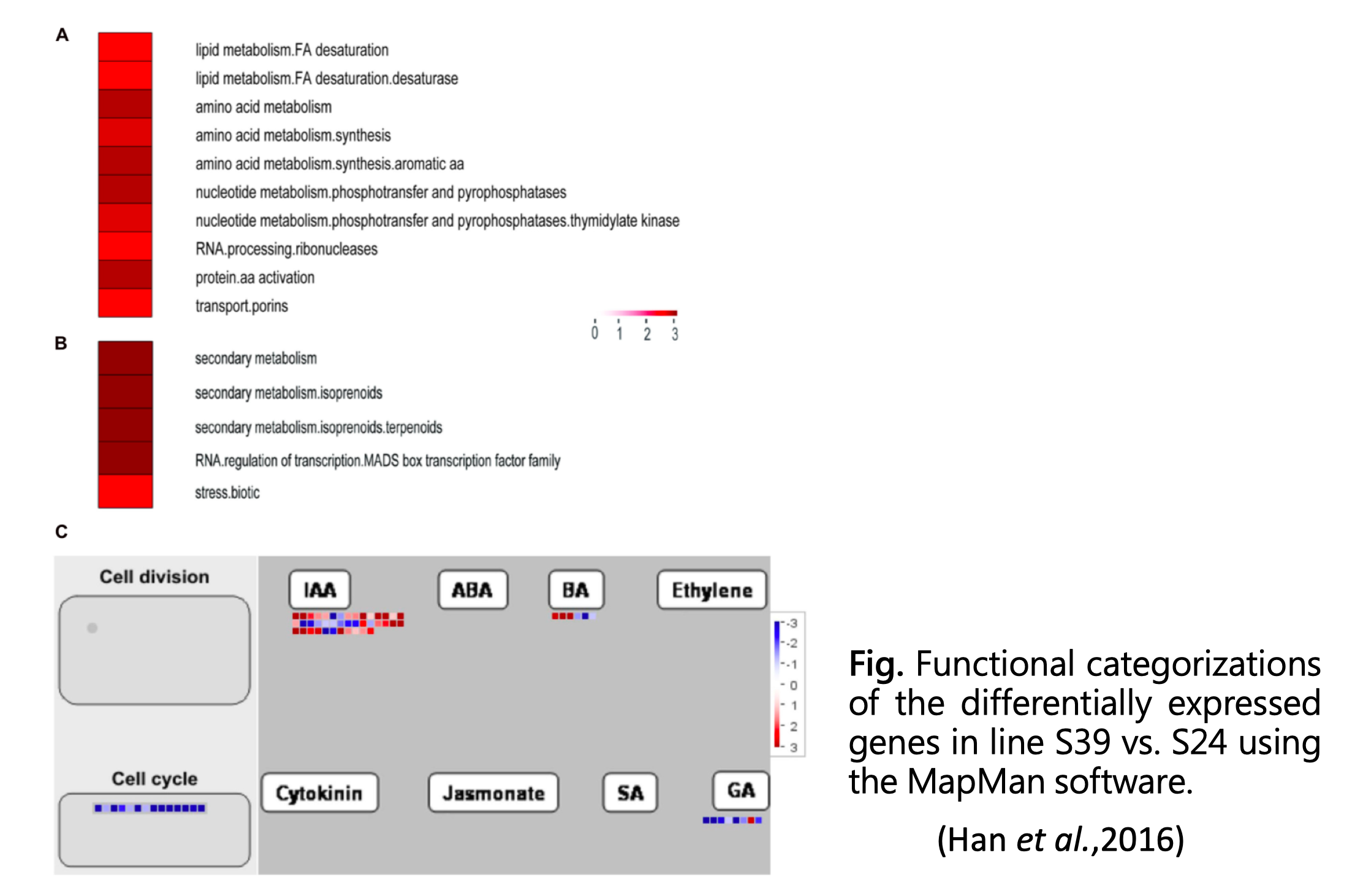
詞彙依然展開排排站,對應的資訊則改用顏色呈現數值的熱度圖表示
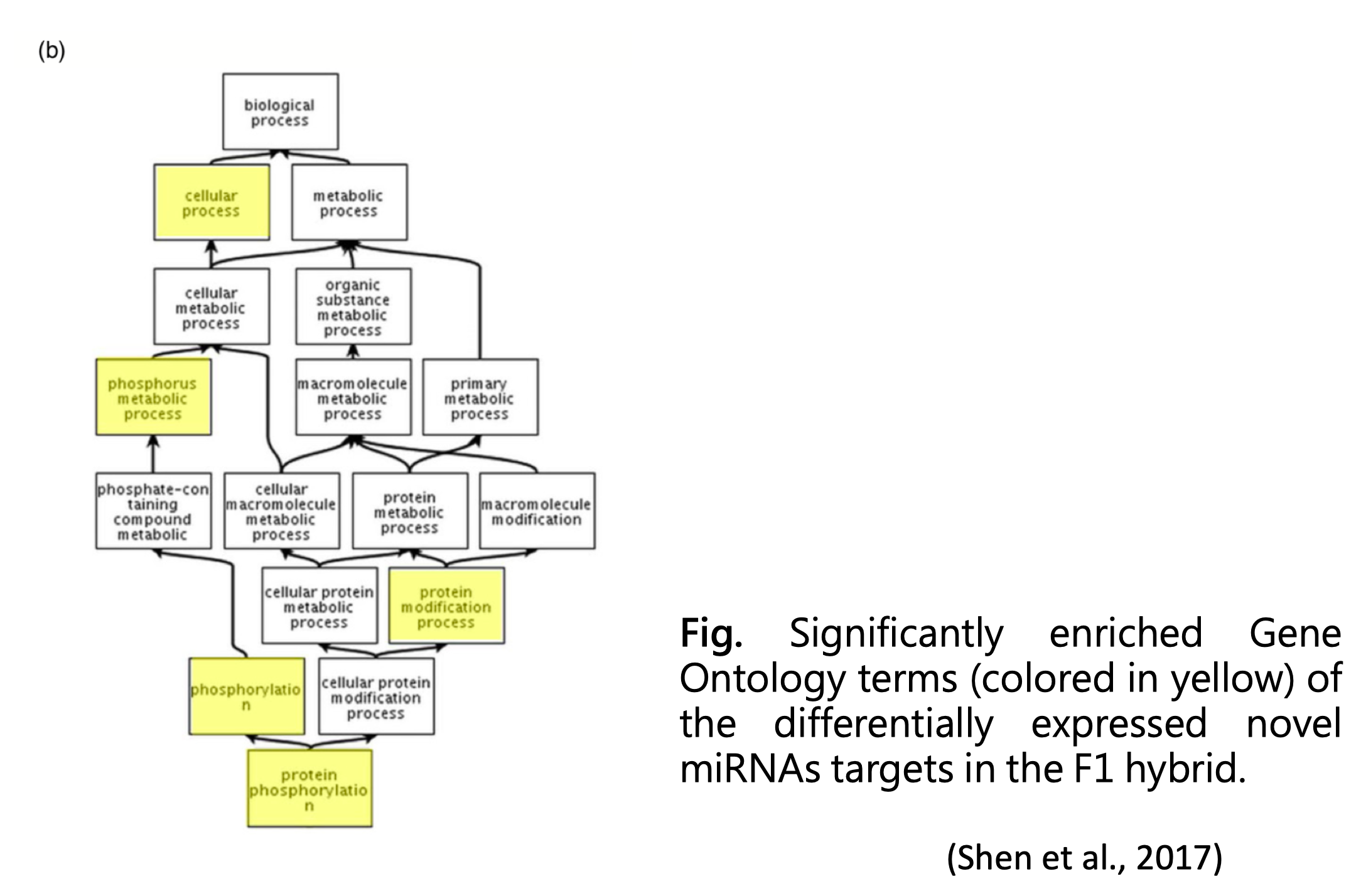
著重於詞彙之間的關係,以 GO 詞彙本身的關係圖為基底,標記出有富集的詞目
詞彙關係與數值資訊並重者,也就是下文會提到的 REViGO。
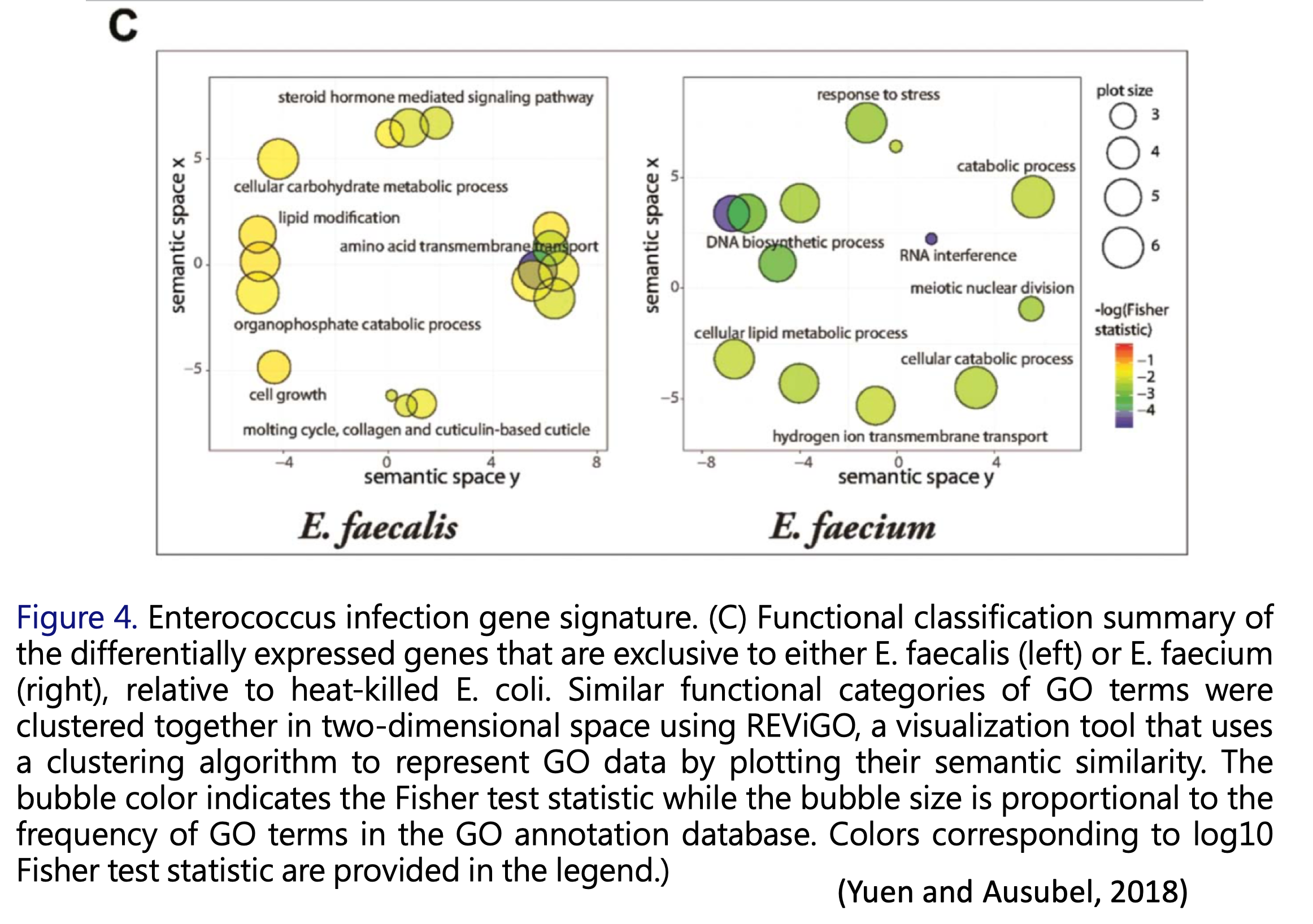
視覺化的工具很多,以下表格簡單地提幾個Google 關鍵字一丟就可以搜尋到的工具以及他們的簡單資訊:
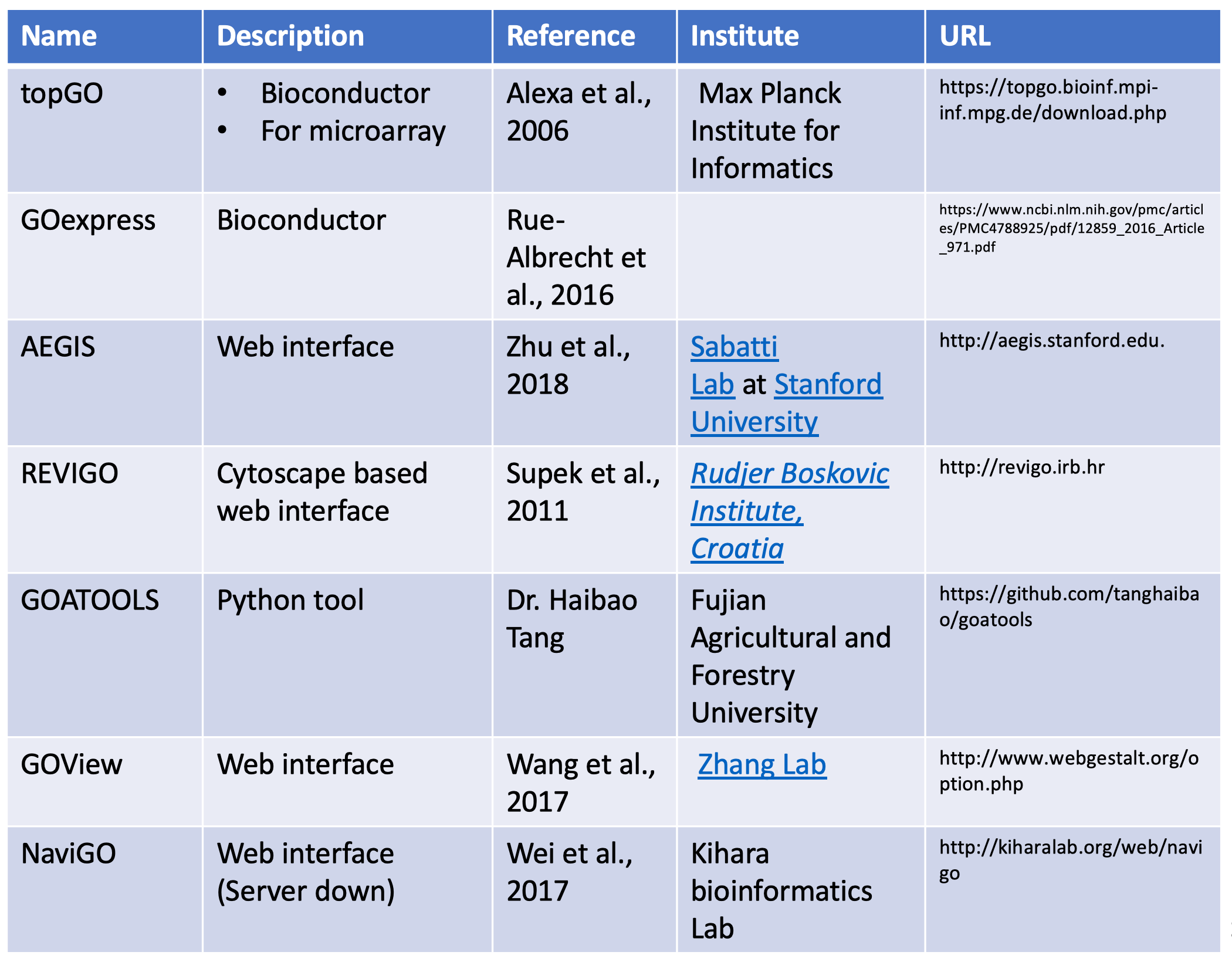
其中 AEGIS, REVIGO, GOView 是乍看不用寫程式就可以完成視覺化的工具,經過本人簡單評測之後心得如下:
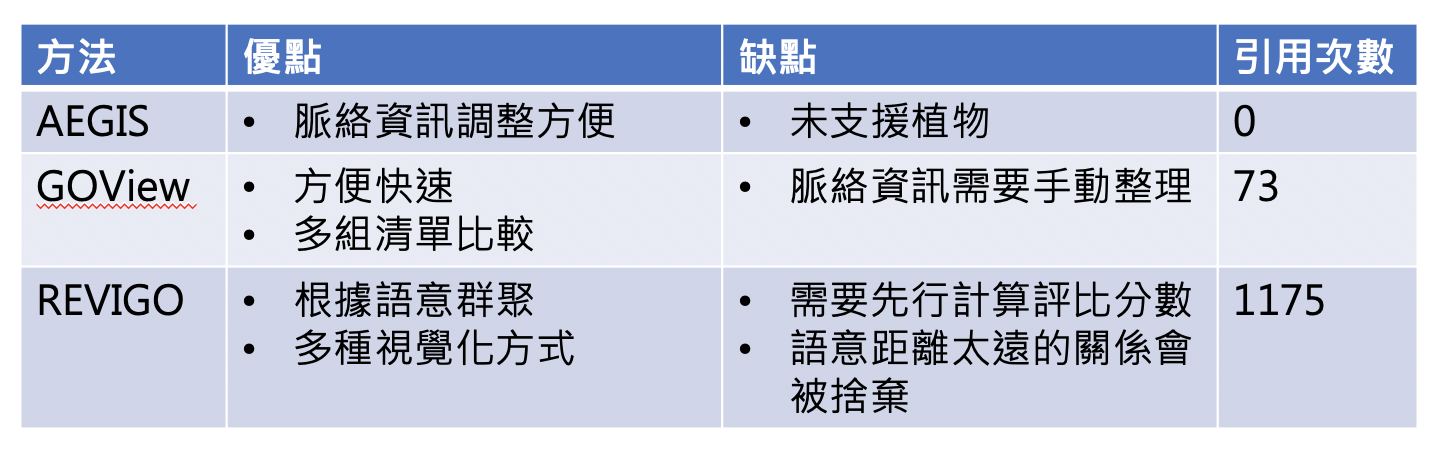
引用次數於 2018.10.26 調查,其實比較新的工具引用次數自然比較少。
實際操作很簡單,首先來到 REViGO 的網頁,輸入格式很直觀,一排詞彙一排數值,用 tab 分隔。下方參數簡單勾選,結果不滿意的話就回來重新調整。
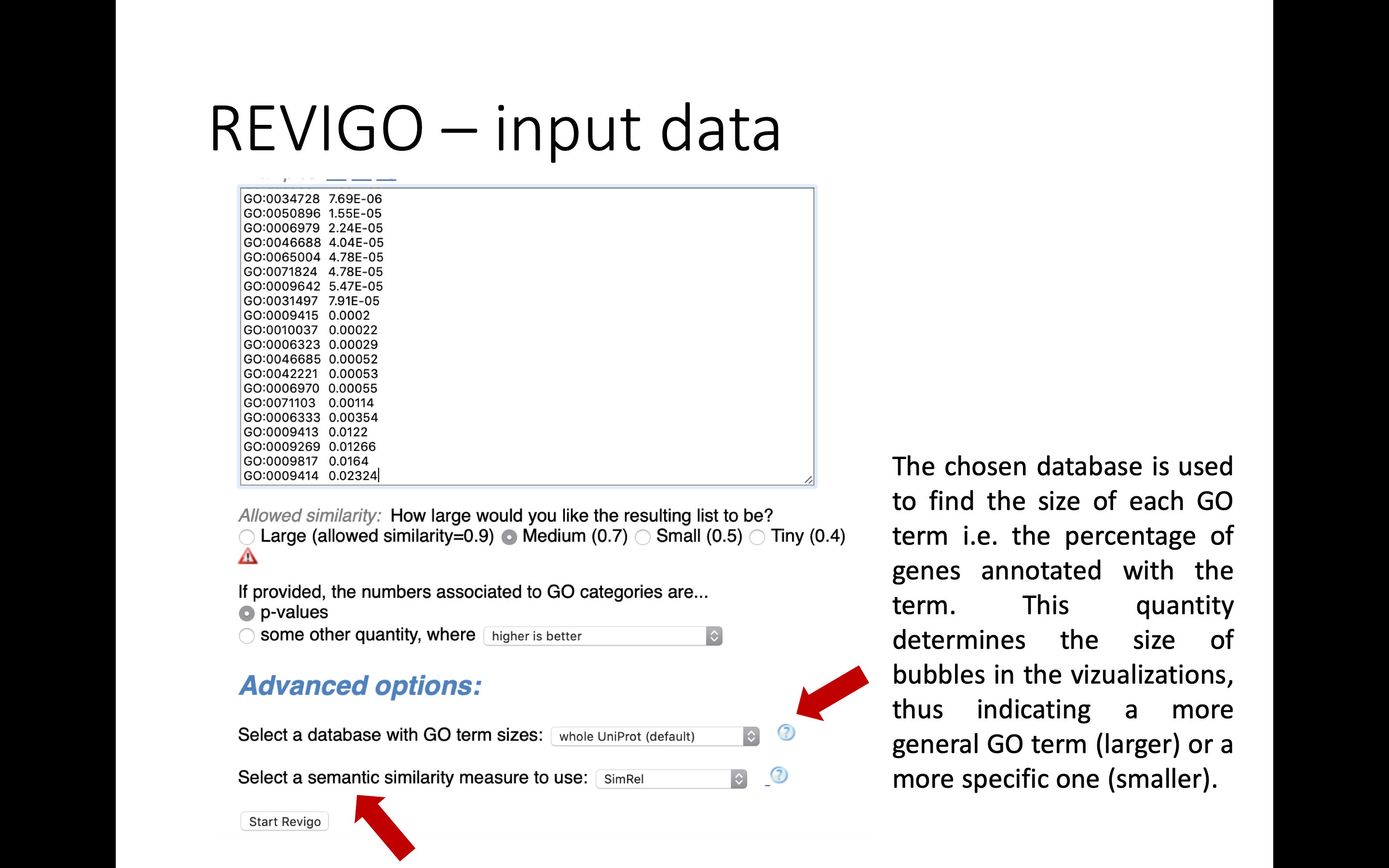
其中比較難以理解的大概是所謂的語意相似度 (semantic similarity),但其實影響不大。

有幾種不同的輸出結果,第一種是 scatter plot,可以下載下來用自己電腦上的 R 語言修改繪製細節。網頁介面下也可以自己勾選來調整要顯示的詞彙名字,詞彙標籤也可以拖曳來調整。
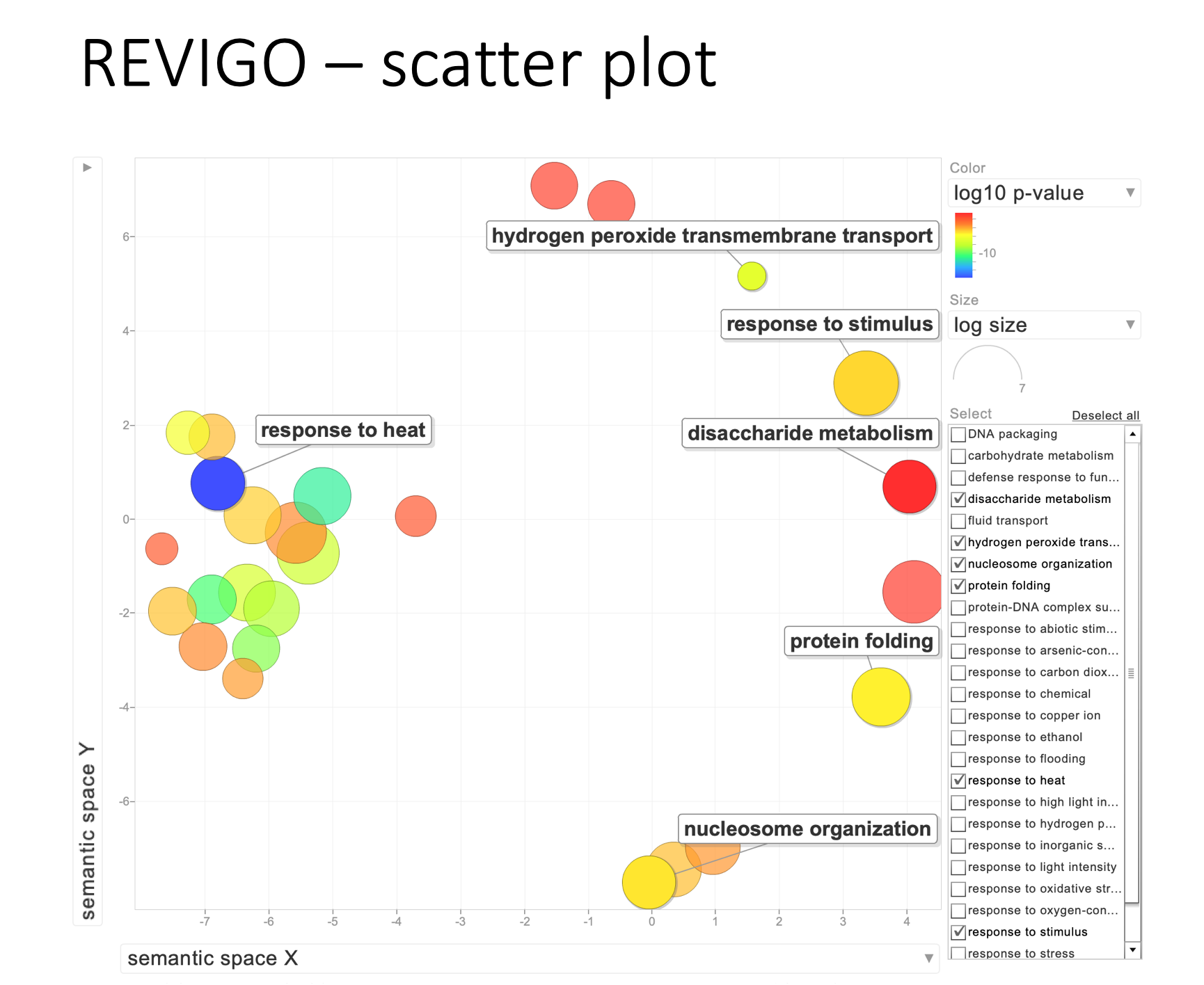
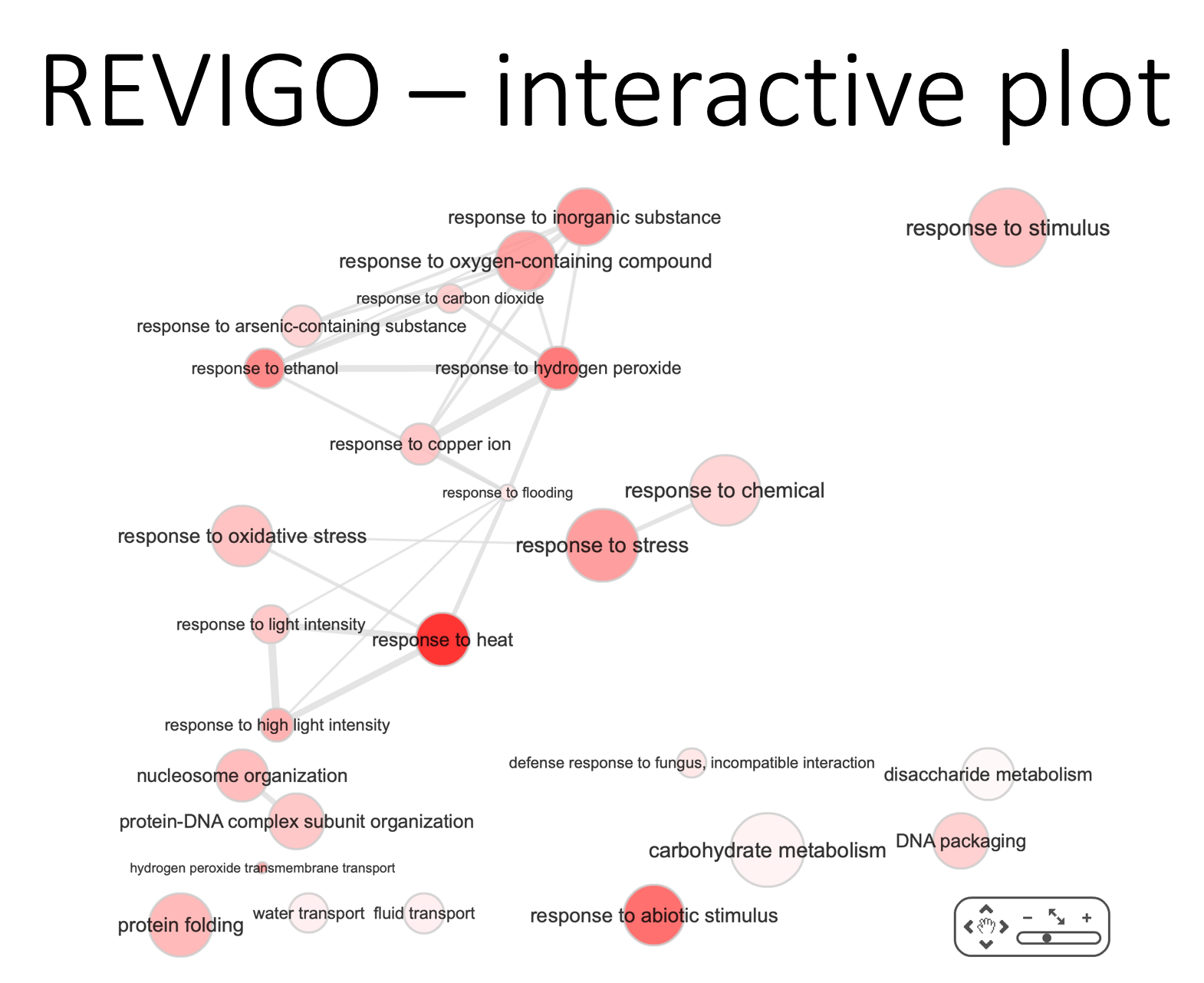
第二種是 interactive plot,相似關係用線條表示,每個點點都可以拖曳。可以下載到 cytoscape 中近一步優化畫面。以上兩種方式都是直接用空間關係表示語意的相似程度,但是捨棄了 GO 原有的階層式關係。
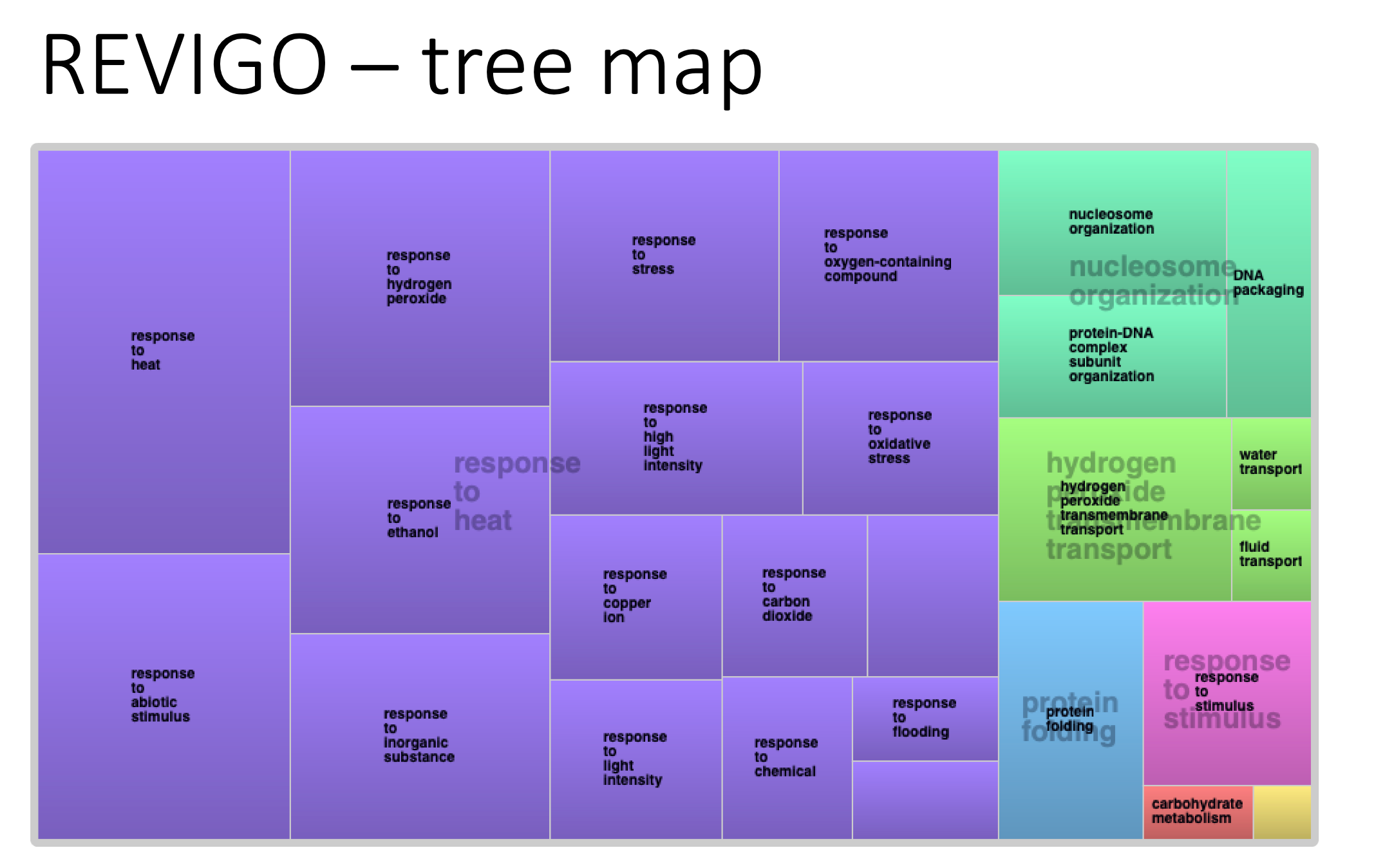
第三種是 Tree map,這就比較抽象了,也比較少看到。這個的優勢在於保留了詞彙之間的階層關係。
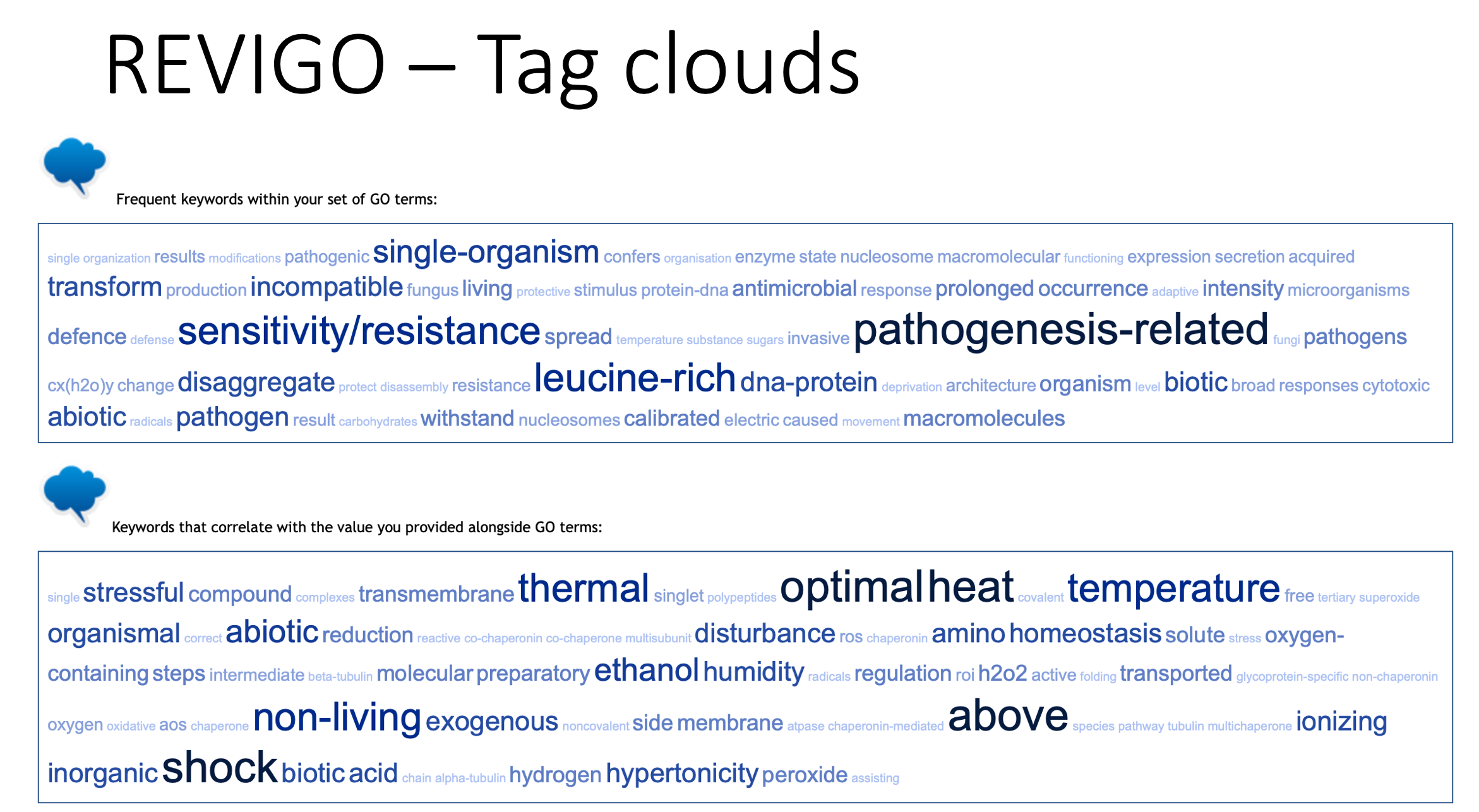
第四種則是標籤雲,更少看到期刊論文使用,但是用來快速了解影響方向還是很方便地。
最後來看看 REViGO 被引用的文章不乏刊載於高點數期刊的研究,因此可以安心使用。
以上就是簡單的基因本體論富集分析的三篇介紹,從基本概念、富集分析、到視覺化,如果還有交代不清楚的地方請留言告訴我~
REVIGO Summarizes and Visualizes Long Lists of Gene Ontology Terms
REVIGO summarizes and visualizes long lists of Gene Ontology terms
關於作者
謝晨 (Chen Hsieh),臺大園藝暨景觀學系研究所碩士。讀碩士前的興趣是懷著寫點程式妄圖解決農業問題的夢想參加比賽,拿了幾個黑客松與 Open Data 創新應用競賽的獎,卻都沒有勇氣將項目經營下去;研究所期間的興趣轉換成讀學術期刊的出刊電子報。靠著這些興趣當選 107 學年的臺大優秀青年,畢業後卻成了無業的實驗室居民。現在在農場旁的研究館辦公室寫點東西,希望可以跟世界分享生物資訊與園藝的樂趣!
感謝選擇匿名的朋友協助校閱初稿與提供意見,也敬請各位讀者不吝指教!
