昨天在研究MDS的時候順便把PCA也複習了一下,所以今天來把它相關的原理補上(非常推薦觀看reference的影片呦,大概是我看過PCA講解得最清楚的!),PCA是另一種把高維度資料轉化成低維度資料的方法,讓我們將本多維度的資料視覺化。
主要利用“線性”辦法降維,也就是降維生成的每一個PC都是原本變數(x1,x2,x3...)的線性組合,因為是線性組合,我們也可以透過beta對應到哪一些變數是比較有用的,比如 PC1=0.8x1+0.25x2,由於0.8>0.25,可以知道x1為主要成份並有較大的影響。
那PC是如何生成的呢?
假設我們現在有一個實驗室資料集,我們想知道老鼠間基因的相似程度(gene1,gene2),我們先把資料畫出。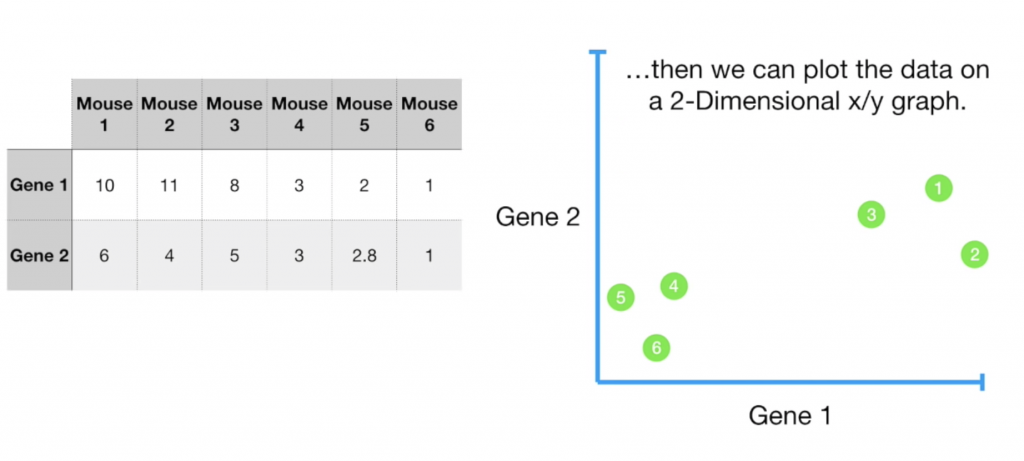
接著移動資料,將中心點調整至(0,0),並找出一條通過原點的線,讓每個點到這條線的平方距離最小,這條線就會是我們的PC1,而PC2就是垂直於PC1的線,若有PC3,則為一條垂直於PC1與PC2的線,依此類推。
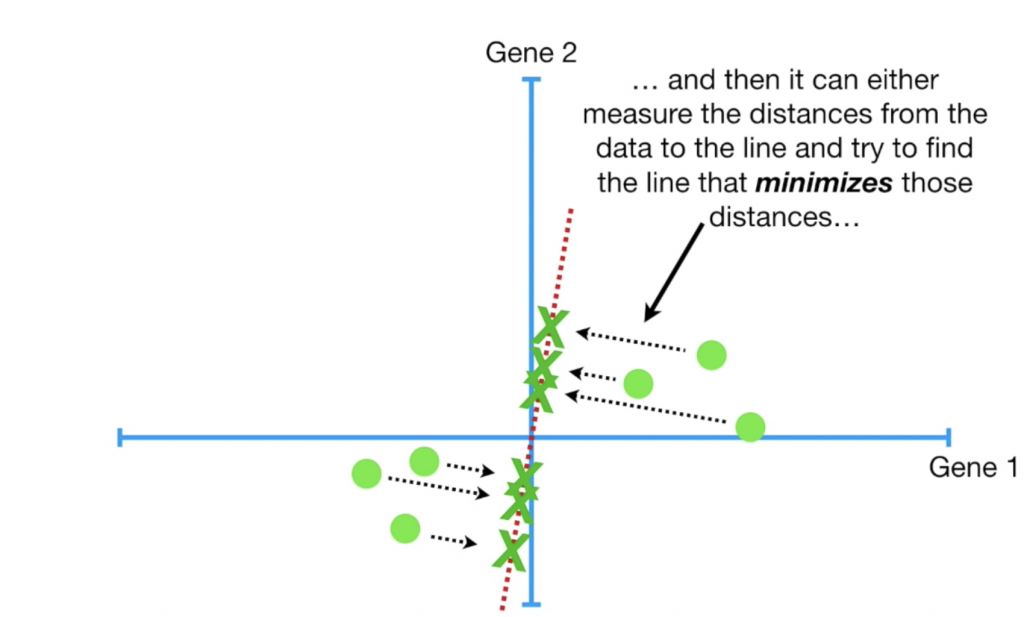
在實際計算中,其實pc計算距離時並不是計算min(點到線的距離平方和),而是計算max(點投影在線上之後到原點的距離平方和)(eigenvalue),我們可以用簡單的畢氏定理知道這是同一件事:當a固定,想求出最小的b等同於球出最大的c。
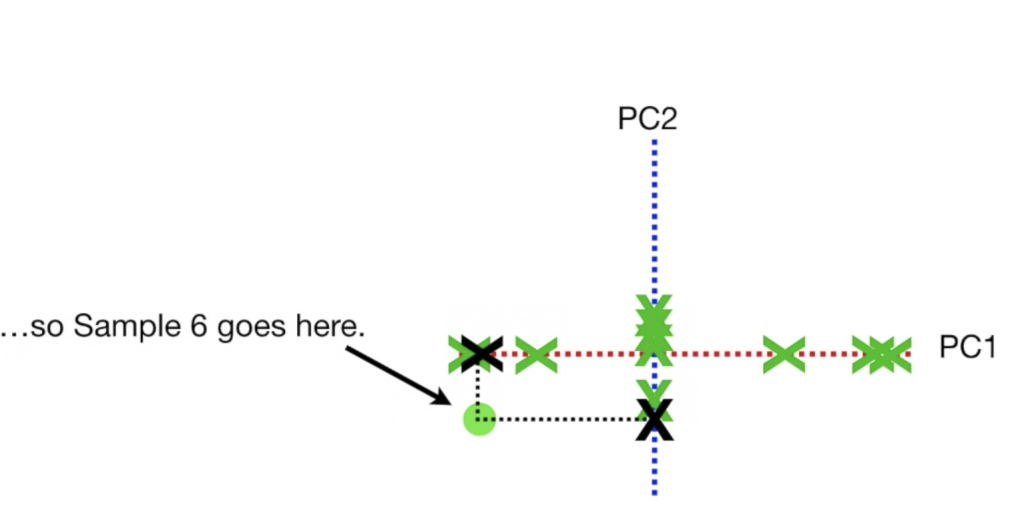
最後呈現的圖,就是把圖旋轉,讓PC1為x軸,PC2為y軸,再把資料點根據投影在兩條線上的位置畫出對應位置: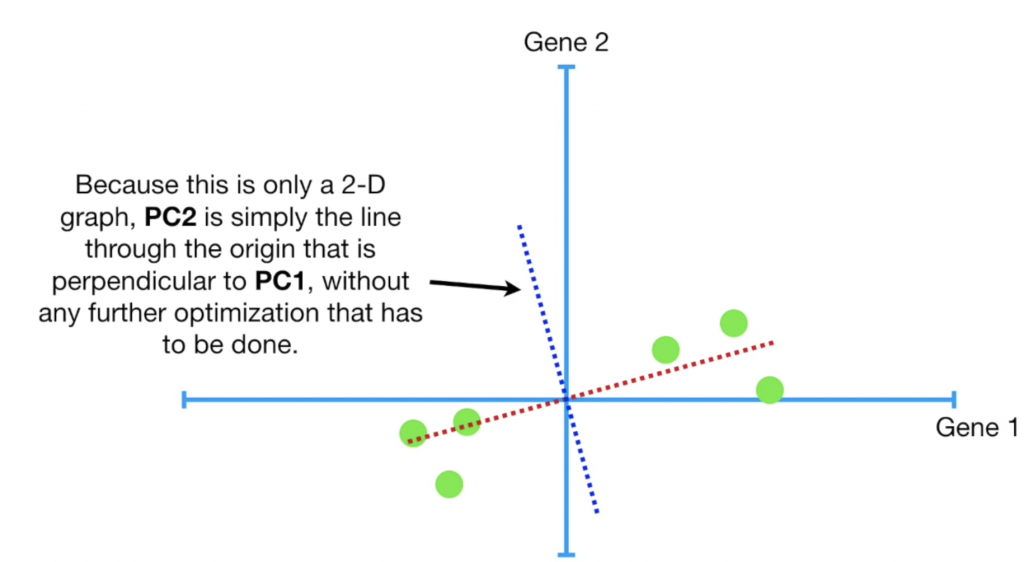
另一個常見的問題是要將資料將成幾個維度,就像是k-mean cluster要定義的k一般,這時候我們可以看看每個pc解釋了多少資料離散程度,variance公式為 eigenvalue/(n-1),加總所有PC的variance並算出每個PC解釋的比例,解釋比例越高代表PC越有效(通常variance解釋比例會逐漸下降),我們可以由此找出想要的PC數量,比如總共有5個PC,但PC1,PC2已經解釋95%的離散,那選擇2個PC就可以很好的表達資料並不需要再增加。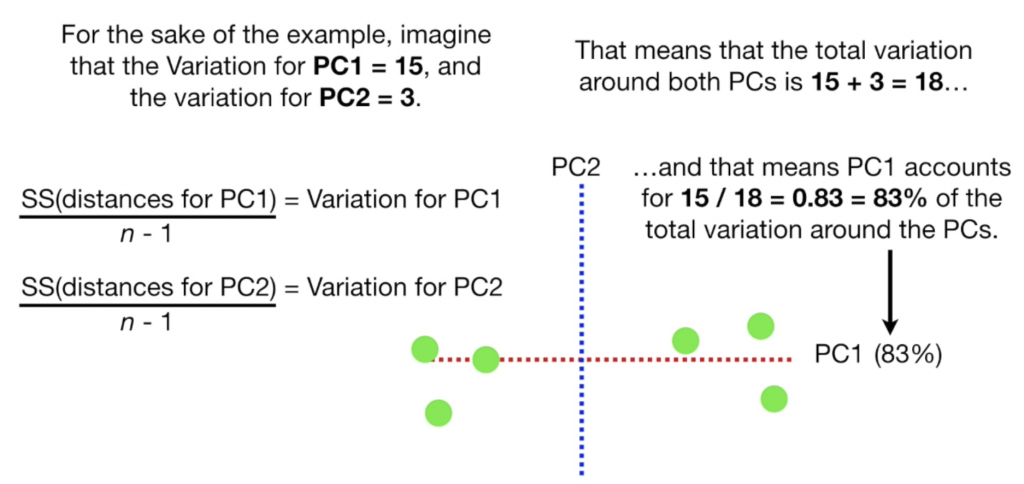
我們一樣用昨天iris dataset來舉例:
import numpy as np
import pandas as pd
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
from sklearn.manifold import MDS
from sklearn.preprocessing import MinMaxScaler
data = load_iris()
X = data.data
scaler = MinMaxScaler()
X_scaled = scaler.fit_transform(X)
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
pca = pca.fit_transform(X_scaled)
pca_df = pd.DataFrame(data = pca,columns = ['pc1', 'pc2'])
pca_df['target']=data.target
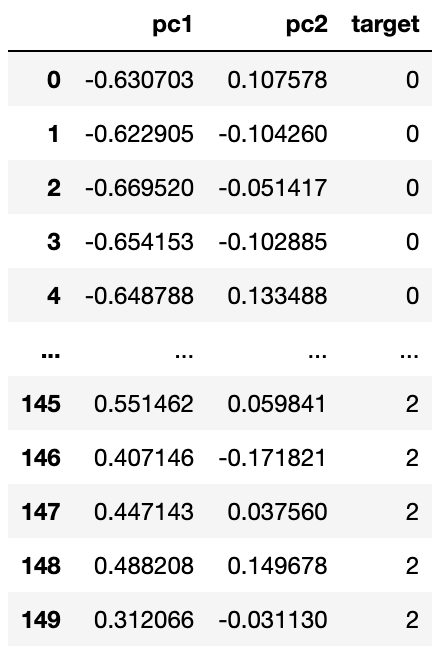
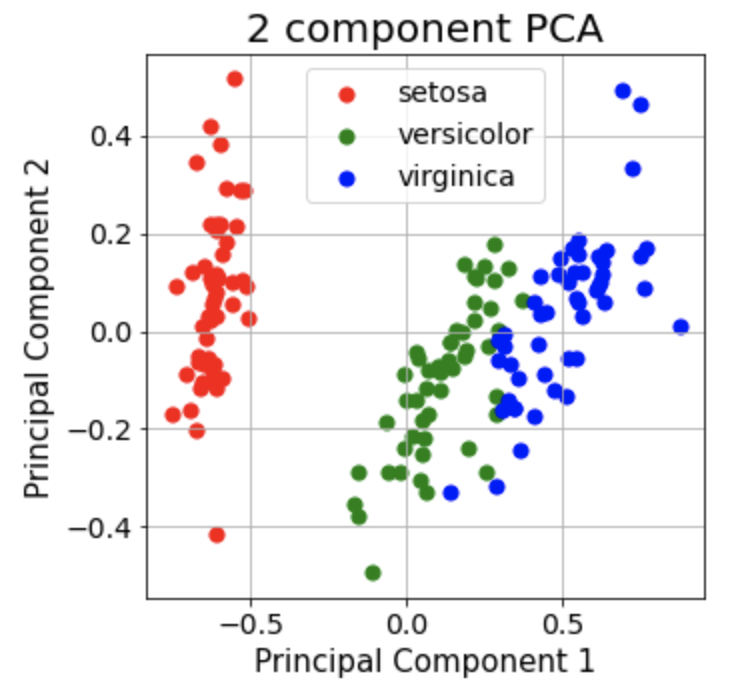
fig = plt.figure(figsize = (5,5))
ax = fig.add_subplot(1,1,1)
targets = [0, 1, 2]
colors = ['r', 'g', 'b']
for target, color in zip(targets,colors):
indices = pca_df['target'] == target
ax.scatter(pca_df.loc[indices, 'pc1']
, pca_df.loc[indices, 'pc2']
, c = color
,label=data.target_names[target])
ax.legend()
ax.set_xlabel('Principal Component 1', fontsize = 15)
ax.set_ylabel('Principal Component 2', fontsize = 15)
ax.set_title('2 component PCA', fontsize = 20)
ax.grid()
pca.explained_variance_ratio_
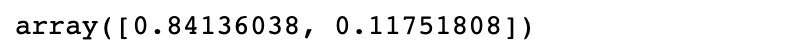
references:
https://scikit-learn.org/stable/modules/generated/sklearn.decomposition.PCA.html
https://www.youtube.com/watch?v=FgakZw6K1QQ
