今天想來談談一個把高維度資料可視化的應用:MDS,MDS是一種unsupervised machine learning approach 用來降低資料的維度,也有另一個說法叫做Principal Coordinate Analysis(PCoA)。
當MDS要把高維的資料降低成低維度時,會確保樣本間的距離保持不變,而如果今天我們把維度降為2,那我們就可以把降維樣本畫出2D圖像來看分佈情形。可以應用在偵測資料有沒有outliers,或是在做cluster之前也可以先畫MDS看看資料分布。
MDS主要的原則是保持“距離”不變,但這裡的距離是可以變換的,我們可以使用最簡單的euclidean distance,也可以使用第一天提到的DTW,要注意的是如果使用euclidean distance,跑出的分佈結果會和PCA相同。另外一個要注意的地方是因為牽涉到距離,所以要執行MDS的資料必須要先標準化(normalization)。
來用有名的iris dataset舉例:
import numpy as np
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
from sklearn.manifold import MDS
from sklearn.preprocessing import MinMaxScaler
data = load_iris()
X = data.data
scaler = MinMaxScaler()
X_scaled = scaler.fit_transform(X)
MDS的重要參數有:
mds = MDS(n_components=2,random_state=0)
X_2d = mds.fit_transform(X_scaled)
colors = ['red','green','blue']
for i in np.unique(data.target):
subset = X_2d[data.target == i]
x = [row[0] for row in subset]
y = [row[1] for row in subset]
plt.scatter(x,y,c=colors[i],label=data.target_names[i])
plt.legend()
plt.show()
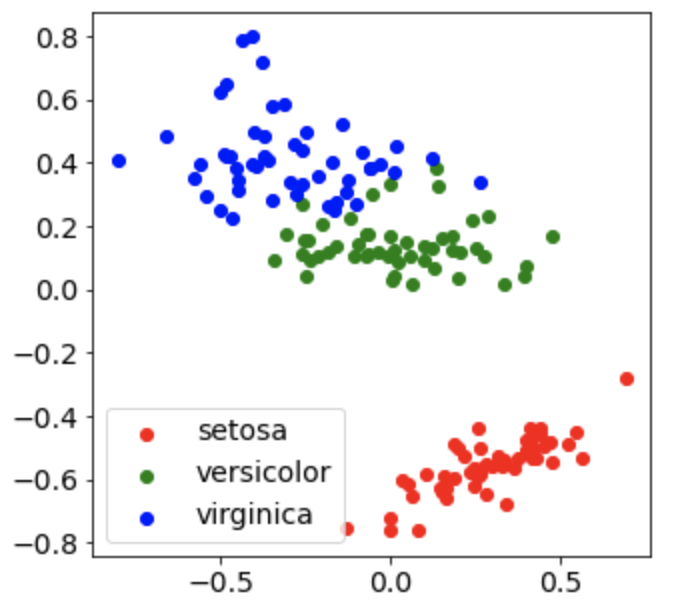
最後要注意的是,上圖中的x與y軸能代表每個資料轉換後的座標,並沒有實質上的解讀意義呦!
references:
https://www.youtube.com/watch?v=GEn-_dAyYME
https://towardsdatascience.com/visualize-multidimensional-datasets-with-mds-64d7b4c16eaa
https://scikit-learn.org/stable/modules/generated/sklearn.manifold.MDS.html
