上一篇我們的基因體時代-AI, Data和生物資訊 Day28-COVID大數據:資料哪裡來
開始進入另一個主題,關於COVID時期,其衍生的資料量其實是相關可觀的,分享目前有哪些關於疫情的公開資料是可以由API、官方網站或是相關應用而來的。
這次COVID疫情,造成全世界的動盪不安,但其實對於生醫領域的人則是看到完全不一樣的一面,就是人類生醫技術的進步,怎麼說呢?以前對於新興傳染病的認知和診斷,從來沒有像現在那麼的快速,甚至過去歷史沒有在這麼短的時間內就開發了藥物以及檢驗試劑,這其實就歸功於基因定序技術的進步以及檢測大數據輔助開發的關係。過去,對於新興病毒傳染病的診斷,都是依據病人的症狀以及相關的旅遊史來做推測,甚至如過去的SARS,可能都快過一年以上,才把相關的基因定序完成,可以看看下面這個時間軸,在2019年12月31號開始有相關新聞發布。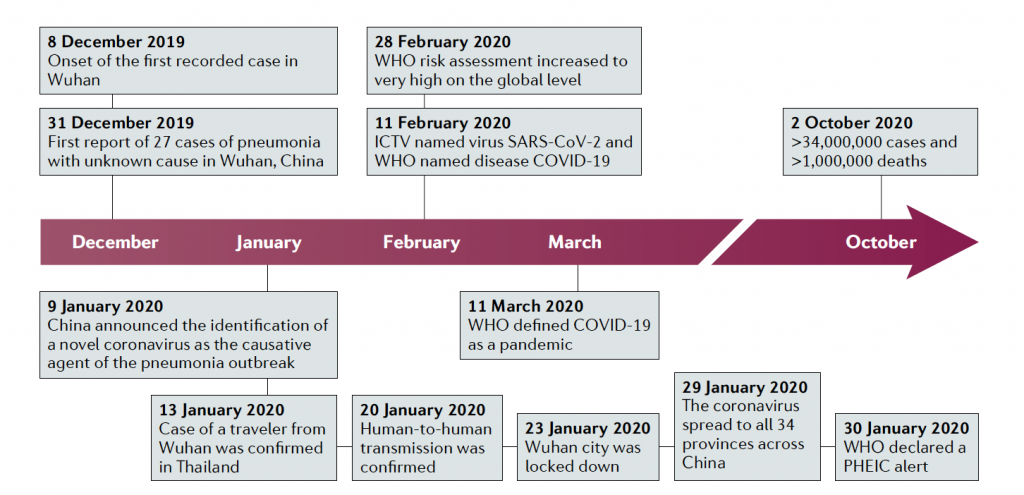
而在2020年1月11日左右我們就有這隻新興病毒的定序資料,而且這樣跨國合作的資訊就在Twitter平台上公開,相關資料也能很輕易地取得,這個算是前所未見的事情。
也許這樣過度簡化病毒了,但本質上其實可以這樣理解,細菌之於質體,有如人類之於病毒的關係,而這段基因其實決定了整個病毒的一切個性,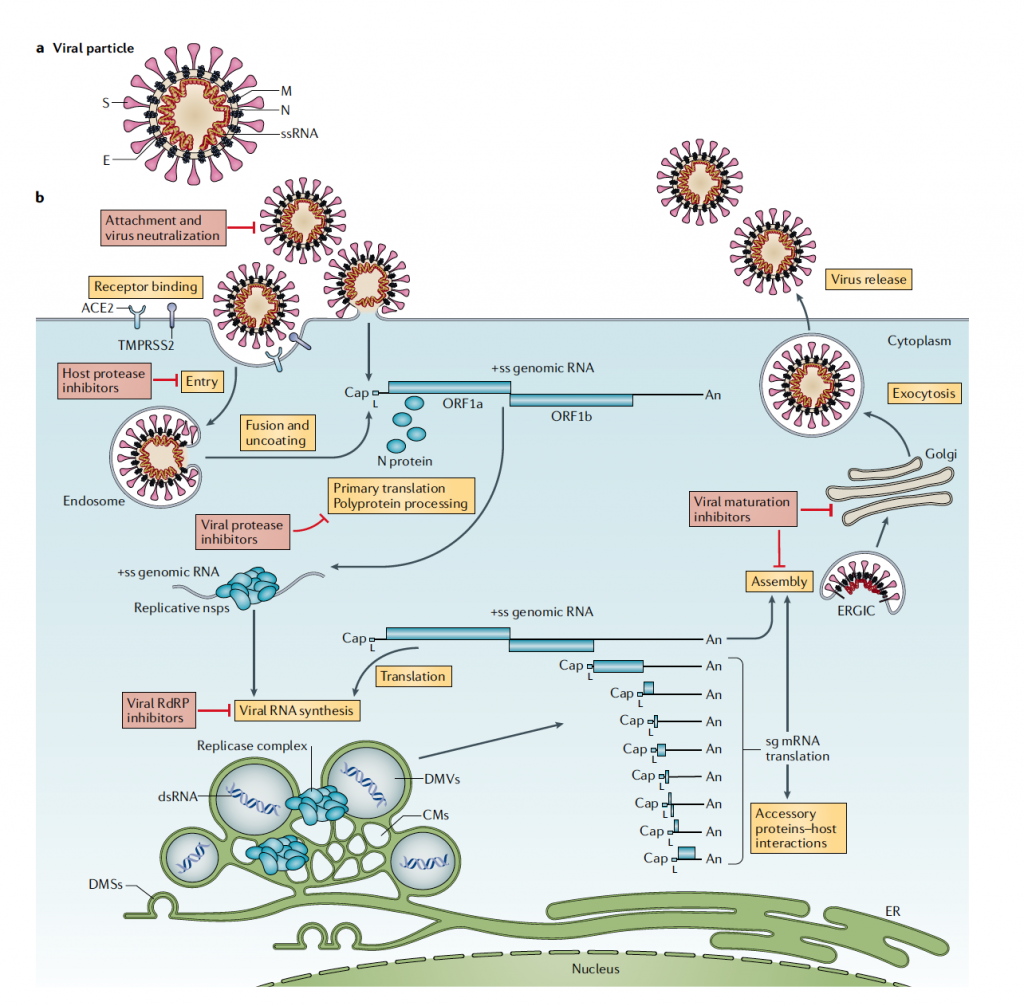
從上面這個figure,其來自於Nature Reivews Microbiology在2021年三月發表的文章:Coronavirus biology and replication:implications for SARS-CoV-2,可以看到整個病毒的序列基本決定了這個病毒的行為和組成。而且這個序列過了一年,我們還持續理解更多事情。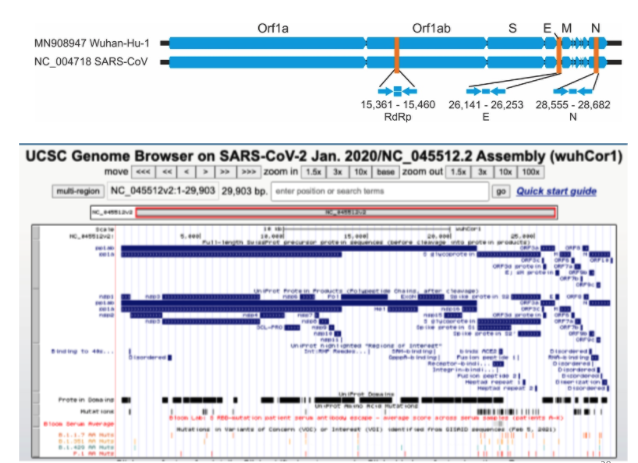
上面這張圖的上面示意圖是我們2020年初對於COVID病毒基因區塊的理解,下面則是過了一年各式各樣的定序資料取得後我們對其更深入的理解。
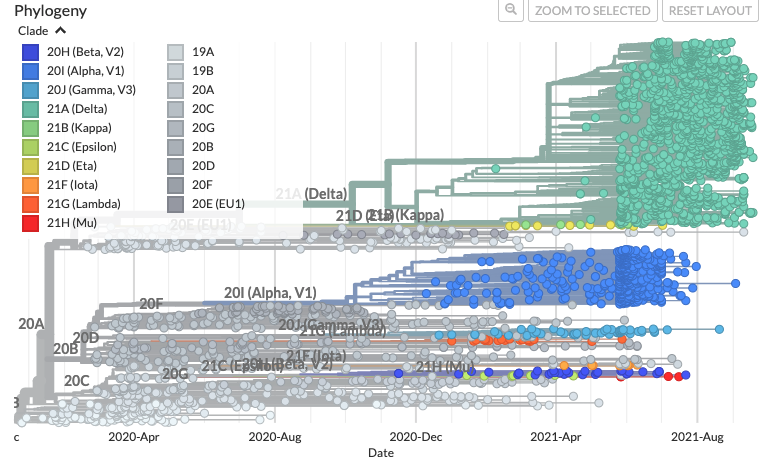
這個來自Nextstrain網站的截圖,這邊視覺化清清楚楚每個COVID病毒定序資料就是一個點,每組資料的相近程度,和演進前後可以由距離和相對關係所組成的,這樣的分析其實相當複雜,方法也很多,其中蠻常見的是使用[Bayesian inference in phylogeny](Bayesian inference in phylogeny)的方法
https://en.wikipedia.org/wiki/Maximum_clade_credibility_tree
Su, S., Du, L. & Jiang, S. Learning from the past: development of safe and effective COVID-19 vaccines. Nat Rev Microbiol 19, 211–219 (2021). https://doi.org/10.1038/s41579-020-00462-y
V’kovski, P., Kratzel, A., Steiner, S. et al. Coronavirus biology and replication: implications for SARS-CoV-2. Nat Rev Microbiol 19, 155–170 (2021). https://doi.org/10.1038/s41579-020-00468-6
Hu, B., Guo, H., Zhou, P. et al. Characteristics of SARS-CoV-2 and COVID-19. Nat Rev Microbiol 19, 141–154 (2021). https://doi.org/10.1038/s41579-020-00459-7
Fernandes, J.D., Hinrichs, A.S., Clawson, H. et al. The UCSC SARS-CoV-2 Genome Browser. Nat Genet 52, 991–998 (2020). https://doi.org/10.1038/s41588-020-0700-8
這個月的規劃貼在這篇文章中我們的基因體時代-AI, Data和生物資訊 Overview,也會持續調整!我們的基因體時代是我經營的部落格,如有對於生物資訊、檢驗醫學、資料視覺化、R語言有興趣的話,可以來交流交流!
