上一篇[我們的基因體時代-AI, Data和生物資訊 Day25- 再深一點:AnnotationHub,從註釋到序列]https://ithelp.ithome.com.tw/articles/10271911/)持續往下分享怎麼用Bioconductor系統的library來調用基因相關的資料,來做近一步的分析使用。
library(AnnotationHub)
ah <- AnnotationHub()
上一篇我們從AnnotationHub的使用,可以調用世界知名機構所產出的資料如美國國家生物資訊中心、博德研究中心、英國生物資訊中心等等,我們也在上一篇中看到其中所含的各式各樣資料類型:
這邊我們主要來分享一下TwoBit這個檔案格式,之前有介紹過傳統的基因序列資訊會使用fasta這種格式來儲存,但為了檔案大小和資料分析方便,而有了壓縮過的基因序列格式,即為twobit,否則動輒好幾Gb的檔案,很容易造成資料傳輸和下載的時候一些問題,這個格式也是由UCSC Genome Broswer裡面所提出的。
我們使用昨天教學的方式來下載一個twobit的檔案,看如何使用
來看一下呼叫這個檔案,他會顯示什麼:
> ahs.GRCh38.cdna.all
TwoBitFile object
resource: /Users/weitinglin66air/Library/Caches/AnnotationHub/fa9efe25aaa_56452
>
他會明顯地顯示這是一個TwoBit檔案物件,其實際上是儲存在這部電腦的哪個資料夾之中。
> class(ahs.GRCh38.cdna.all)
[1] "TwoBitFile"
attr(,"package")
[1] "rtracklayer"
> seqinfo(ahs.GRCh38.cdna.all)
Seqinfo object with 175372 sequences from an unspecified genome:
seqnames seqlengths isCircular genome
ENST00000415118 8 <NA> <NA>
ENST00000448914 13 <NA> <NA>
ENST00000631435 12 <NA> <NA>
ENST00000434970 9 <NA> <NA>
ENST00000632684 12 <NA> <NA>
... ... ... ...
ENST00000627692 927 <NA> <NA>
ENST00000631343 1490 <NA> <NA>
ENST00000633652 508 <NA> <NA>
ENST00000634119 1226 <NA> <NA>
ENST00000631874 147 <NA> <NA>
接者,我們可以看一下怎麼來manipulation這個物件,因為這個是一個S4系統的物件,所以行為會跟傳統R系統的物件不同,開發者可以定義很多不同的使用模式,所以可以先用class函數看一下這個物件是以什麼函數包為主,再去看那個包的介紹。這邊發現他是一個由rtracklayer包所定義的物件,rtracklayer包即為UCSC Genome Browser團隊開發用來跟他們track系統所做的一層應用層。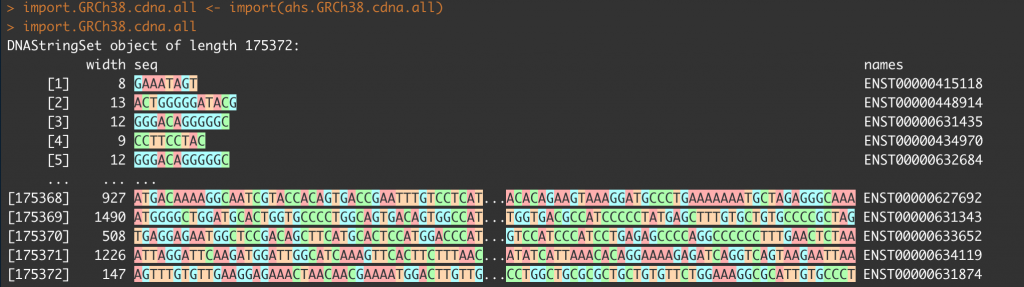
這邊我們直接使用rtracklayer包中的import函數來將twobit物件轉換成序列資訊,這邊也可以用GRanges來指定想要import的序列,這樣也比較不會佔用Ram空間。
How to forge a BSgenome data packag.2021
Efficient genome searching with Biostrings and the BSgenome data
packages.2022
這個月的規劃貼在這篇文章中我們的基因體時代-AI, Data和生物資訊 Overview,也會持續調整!我們的基因體時代是我經營的部落格,如有對於生物資訊、檢驗醫學、資料視覺化、R語言有興趣的話,可以來交流交流!
