「定序」就是將一段序列判讀出ATCG排列的順序,
「迭代」則是為了接近最終目標而反覆改良的過程,
為了能精準有效率知道生物的基因序列,
DNA定序技術一代一代的不斷精進改良。
定序的歷史其實很有趣,
從利用嗜菌體定序、2D電泳,
人類基因組計畫之民間與政府的技術競賽,
故事於網路上資源相當多就不再贅述。
這邊則以1977年Sanger大師發明Chain-termination為界線,
往後介紹人類解開生物程式碼的逆向工程。
又名 Sequencing-Based Typing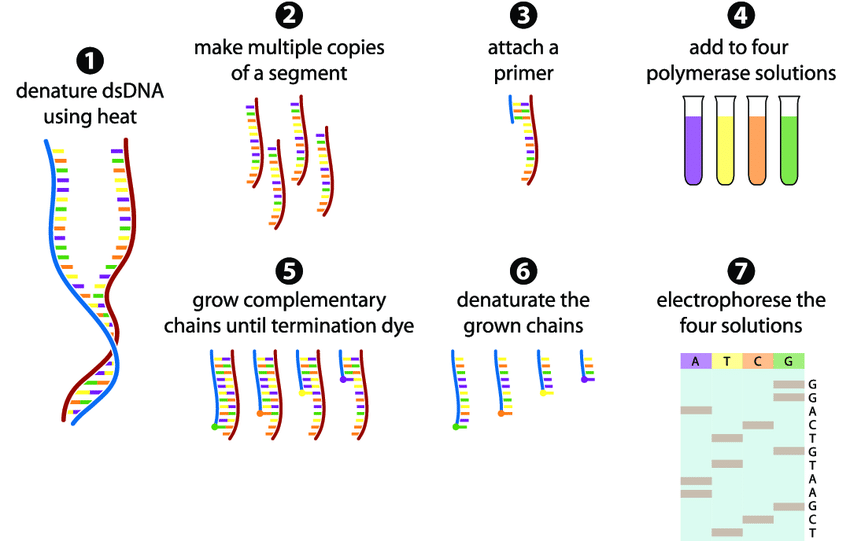 (Gauthier, M. G., 2008)
(Gauthier, M. G., 2008)
其採用的原理是 Chain-termination,也就是藉由合成中終止的方式,
獲得長度不一片段,用跑膠判斷序列
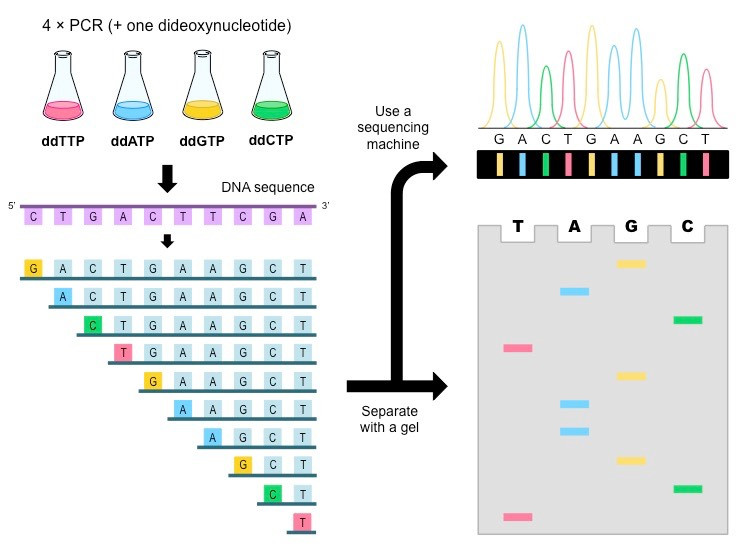
現今已經使用毛細管電泳及機器偵測(右上),因為第一代定序成本較低,時至今日,PCR 或切膠產物的低通量定序還是使用 Sanger 為主。Reference : Microbe Notes
為了解決長片段、大量定序需求以及效率低落等等心累的問題,
經歷多家廠商的競爭,Illumina 公司開發的新技術成功打贏市場,
目前 RNA seq、Single cell sequencing、
Whole genome sequencing、Metagenomic(16S, 18S等等)
主流都採用 Illumina 的 NGS,他們的原理相同,
差別在於樣本的前處理(e.g 轉 cDNA or 夾16S)以及後續分析軟體的流程。
而Illumina 採用的原理是 Bridge amplification + Sequencing by Synthesis (SBS),

他們設計一款 Flow cell 並在像是跑道的地方舖上一層草皮(lawn),
草皮長了兩種 oligos 序列(像是長了兩種小草,分別為紫色跟藍色)。
oligos 指的是短片段單股的 DNA 或 RNA ,又名 Oligonucleotides。
接下來,了解下列步驟非常重要,因為後續分析會用到步驟中很多的觀念 :
將DNA樣本打碎 (約80bp) (第一次看到先打碎序列其實蠻反邏輯的XD
加入Adapters,並使用連接酶加在序列片段頭尾
(可以想像改良過的Primer,差別是頭尾都有,後續可以幫助放大片段)

- Adapters =
目標序列結合位(sequencing binding site)(綠)
- indices(黃、紅) + oligos互補序列(紫、藍)
oligos 互補序列就是為了跟草皮(lawn)小草(oligos)結合
- indices 又名 index,帶有 barcode,每個DNA樣本都會有獨特的條碼。
能同時將不同樣本放在同一個 Flow cell 多樣本大量定序,
這樣子 Pooling 的方式稱為 Sample Multiplexing。
像是大家帶著條碼手環泡在大眾池,這動作英文稱為 pooling,
然後再拿著條碼器(定序)快速逼手環就知道誰是誰了。
帶有Adapters序列片段與oligos結合
聚合酶開始複製,使得草皮上的 oligos 被延長 後也長得與序列片段相同。
被延長後的 oligos 稱為 Hybridized fragment (雜合片段),
再將原序列片段洗去,他不要了,只留下 lawn上的雜合片段。
因為另一端也與 lawn 上 oligos 互補,所以雜合片段會彎腰結合,
形成像是橋 (Bridge) 狀的序列,
接著聚合酶又來複製了,形成兩座 DNA 橋 (Double stranded bridge)
重複第4~5很多很多次,形成上百萬座橋
稱為橋式放大(Bridge amplification)。
然後就會獲得很多根巧克力棒,洗去紫色底座 oligos 上的雜合片段,
留下藍色底座 oligos 上的雜合片段
紫色底座的序列都飛走惹,剩下藍色底座。
在開始定序前,3'端的 oligos 會用一小段序列擋住,
因為這段序列是加上去的,不需要被定序
接下來,帶有四種螢光的dNTP加入到Flow cell中,
只要結合到雜合片段,就會發光!
稱為 Sequencing by Synthesis (SBS) ,因為一邊合成一邊定序。
電腦就會偵測螢光出現的順序,進行讀取,其實超級漂亮的!!
加入一段能辨識indices的primer,
再加入dNTP與聚合酶,
一樣用螢光讀取的方式獲得這些目標序列的 barcode。
稱為 Index Read。
洗去indices的primer 還有各種一切,
留下巧克力棒(Hybridized fragment),Forward 定序讀完了,來讀 Reverse
稱為 Paired-End Sequencing。
因為步驟與 Part C 類似,就不放動畫了,有興趣可以觀看Illumina出品的動畫。

敬請期待,下回分曉。
礙於篇幅,三代定序技術會在介紹完 NGS 資料分析後,於 TGS 分析前介紹。
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
