改編自 (Siegwald, Léa, et al., 2017)
傳統定序分析中,
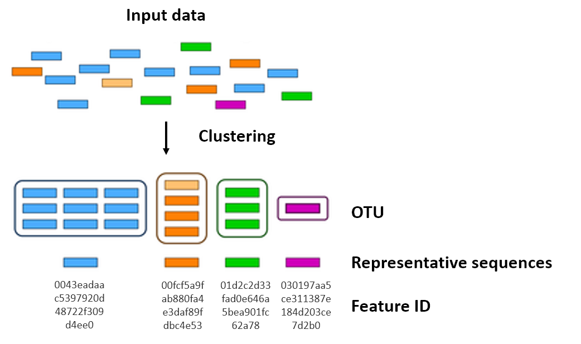
經過品質管制 (Quality control) 會獲得每條約 400~500 bp 的序列上萬條,
並將 97~99% 相似的序列群聚一起 (Clustering),
每一組則稱為 操作分類單元 OTU (Operational Taxonomic Units),
並選出一條可以代表該群的序列稱為代表性序列 (Representative sequences),
就像是組長一樣,給予組長一串編號稱為特徵ID (Feature ID)。
由於利用相似程度群聚的方式,
些微的差異歸類在同一群容易忽略小群體的物種,
(可以觀察上圖的橘色序列有一條是淺橘色)
為了解決上述痛點, DADA2 推出的演算法,
使用了降噪 (Denoised) [Day 08] 得到更高的解析度,
用輕鬆的講法就像是使用了 100% 相似序列進行群聚,
於是 DADA2 作者將 100% 相似群聚的 OTU 取了一個更炫砲的名字:
ASV (Amplicon Sequence Variants),
學習上無論是 OTU 還是 ASV 在輸出的檔案呈現是一樣的,
我們可以打開 [Day 08] 所獲得的檔案rep-seqs-dada2.qzv,
就是基於上述概念所輸出結果: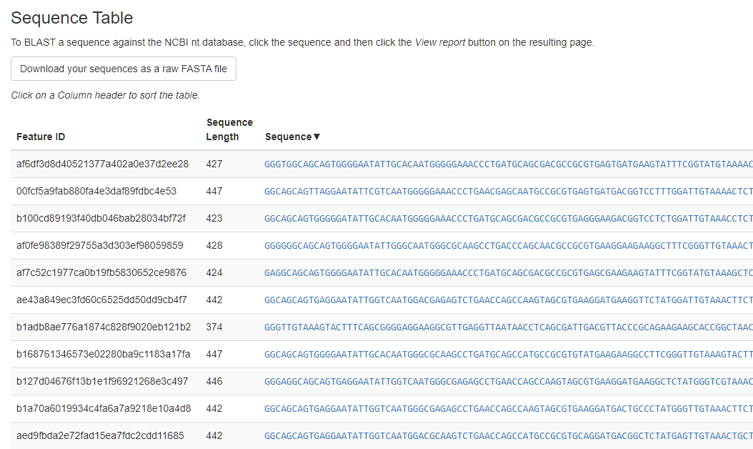
檔案中可見特徵ID (Feature ID) 、長度以及序列,序列帶有的連結可以連到NCBI檢視與序列相關的物種。
品質管制 (Quality control) 完成之後,
獲得了一組組很優良的序列們 (OTU / ASV),
接下來就要來看看他們究竟代表著哪些物種!
幫序列找爸爸媽媽其實有很多種方式,
這裡選擇使用機器學習分類器進行物種分類 (Taxonomy assignment),
沒錯,到第十天我們用到AI了 (?,
不過本系列文著重在應用而非探討,
所以就不為難各位生科人了Q
以下有些簡單的名詞介紹 :
不知道各位是否還聽過查字典比賽,
給一個「生字」讓「訓練」過的「小朋友」翻「字典」找「部首、注音、頁數」,
在尋找序列的所屬的物種名時,
可以想像
分類器 (classifier) 就是小朋友,
微生物資料庫 (microbiota database) 就是字典。
什麼都不說,直接給小朋友一本字典,
他可能連用都不會用,所以需要訓練 (training)。

一位正在受訓練的小朋友。
套用在序列分析的情境 :
給一段「序列」讓「訓練」過的「分類器」翻「 」找「物種名」。
翻「 」 嗯???
分類器畢竟是電腦中的玩意兒,記憶力超強,
所以這位分類器小朋友是天才,那麼 :
訓練好的小朋友,是可以把整本字典背起來的
也就是說訓練好的分類器是不需要資料庫輔助了~
16S 有 V1~V9 的變異區片段,
坊間已有針對特定幾區 (如V3-V4等)
提供 已訓練 (pre-trained) 的分類器,(練好的小朋友)
但要特別注意的是,
假設是做 V3-V4 的定序,
V1-V9 全長訓練好的分類器,
是不一定會比針對V3-V4訓練好的分類器效果來得佳的,
所以不要貪心。
| Greengenes | SILVA | RDP | |
|---|---|---|---|
| 域(Domain) | Bacteria and Archaea | Bacteria, Archaea and Eukarya | Bacteria, Archaea and Fungi (Eukarya) |
| rRNA 區域 | 16S | 16S/23S for Prokaryotes and 18S/28S for Eukarya | 16S rRNA (Bacteria and Archaea) and 28S rRNA (Fungi) |
| 資料庫大小 | 最小 | 最大 | 次之 |
| 適合環境 | 人類 | 人類、動物、土壤、海洋等 | 人類、動物、土壤、海洋、真菌等 |
| 最近版本 | 2013 (v13_8) | 2020 (v138) | 2020 (v18) |
另外,像是 NCBI、OTT 也是常見的微生物資料庫。
Campos, Philip M., et al. (2022), Eldred, L. E., Thorn, R. G., & Smith, D. R. (2021), Balvočiūtė, M., & Huson, D. H. (2017)
本篇所使用的是由 anweshmaile GitHub
所釋出的 Silva 138 V3-V4 已訓練好的分類器,
也是剛好在寫這篇文章時所找到的。
關於已訓練的分類器在 QIIME2 文件中也有提及到,
只是並無 V3-V4 片段,僅有 V1-V9, V4,
若有機會使用時記得到 QIIME2 文件中引用對應的論文 :
QIIME2 官方建議使用者自行訓練分類器,可以達到最好效果 :
Taxonomic classifiers perform best when they are trained based on your specific sample preparation and sequencing parameters, including the primers that were used for amplification and the length of your sequence reads.
實務上若樣本來源常見(土壤、水質、糞便等),
使用其他人訓練好的分類器效果也蠻好的,
本篇所使用的是已訓練好的單純貝氏分類器 (Naive Bayes classifiers)。
本篇使用到的檔案 :
rep-seqs-dada2.qzv
這篇的概念實際上水很深,有興趣的還是推薦看看官方文件,會有更多的收穫。
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
