今天天氣晴朗 走輕鬆路線,
來畫一張大小朋友教授都愛的熱圖 (Heat map) (? :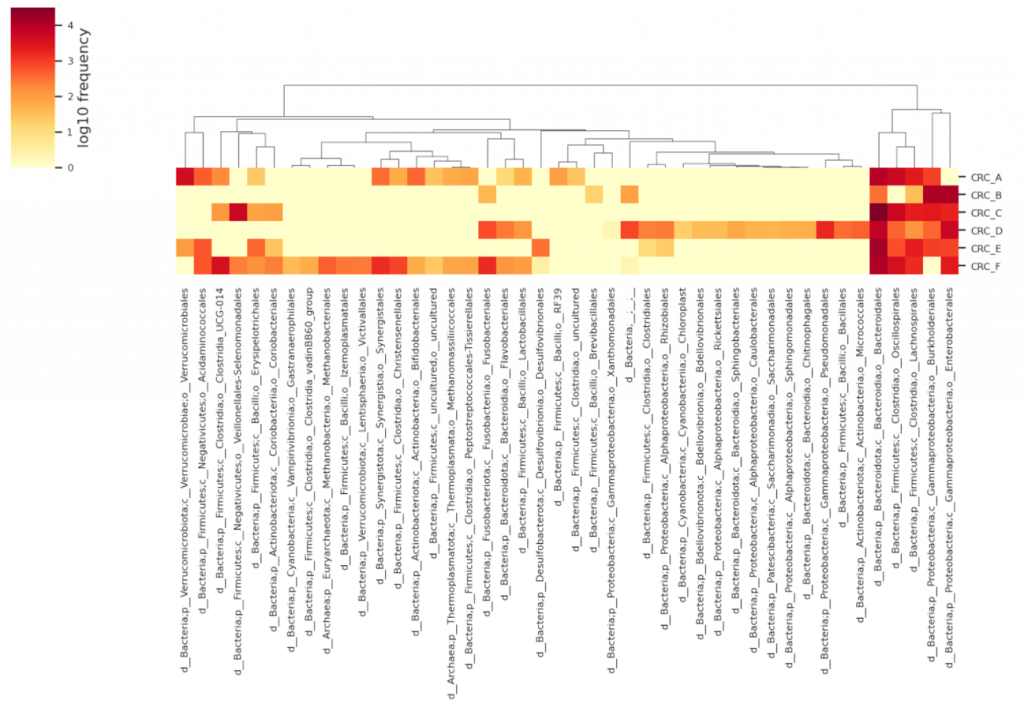
熱圖可以幫助判斷樣本之間的菌種豐富度分布,
能夠快速看出哪些是共同菌,哪些是一些樣本才有的菌種。
如果在 [Day 13] 有好好研究親緣關係樹 (Phylogenic tree) 的 iTOL 結果圖,
會發現許多特徵ID (Feature ID) 會對應到同一物種,
使得在結果圖上同一物種重複出現,
但因為在親緣關係樹是以序列比對 (alignment) 角度出發,
強調的是序列的相似度(距離),是需要存在的結果。
而在熱圖中,會經歷兩個步驟,
先將對應到同一物種的序列進行合併(條數加總),
這過程 QIIME2 將其稱為折疊 (collapse),
再進行繪製。
Collapse : Collapse groups of features that have the same taxonomic assignment through the specified level. The frequencies of all features will be summed when they are collapsed.
進入 qiime2-2022.8 環境後輸入 :
qiime taxa collapse \
--i-table table-dada2-240.qza \
--i-taxonomy taxonomy.qza \
--p-level 4 \
--o-collapsed-table col_table_4.qza
--p-level : 這邊就要決定要繪製的分類階層 (taxonomic rank) :
參數 階層 \ 1 | 界 Kingdom |
\ 2 | 門 Phylum |
\ 3 | 綱 Class |
\ 4 | 目 Order |
\ 5 | 科 Family |
\ 6 | 屬 Genus |
\ 7 | 種 Species |
--o-collapsed-table : 檔案管理方便,若是有輸出不同階層,記得更改檔名。
完成後會顯示 :
'
Saved FeatureTable[Frequency] to: col_table_4.qza
'
qiime feature-table heatmap \
--i-table col_table_4.qza \
--o-visualization heatmap_4.qzv \
--p-color-scheme YlOrRd \
--p-cluster features
--p-color-scheme : 好的熱圖顏色帶你上天堂,一般來說會希望越多的顏色越深,還有更多選擇,以下顯示私心推薦的選項,喜歡就直接記住前面的單字於指令中改動 (若此參數不填,跑出來蠻醜的)。
--p-cluster : 可使用 features、samples、both、none,
根據序列/樣本/兩者/都不要,
進行聚類 (clustering) 排序。
若樣本名稱帶有分組的含意(如 CT_1, CT_2, EXP_3, EXP_4),
可以使用 features 就好,就能快速看出實驗組是否與對照組間有菌相差異。
除了上述參數外,還有一些進階選項 :
例如 --p-method 依照不同階層式分群法 (hierarchical clustering)可以分成 : average、centroid、complete、median、single、ward、weighted。
預設為 average [選填參數]
完成後會顯示 :
'
Saved Visualization to: heatmap_4.qzv
'
同樣將heatmap_4.qzv拖曳到 QIIME2 VIEW :
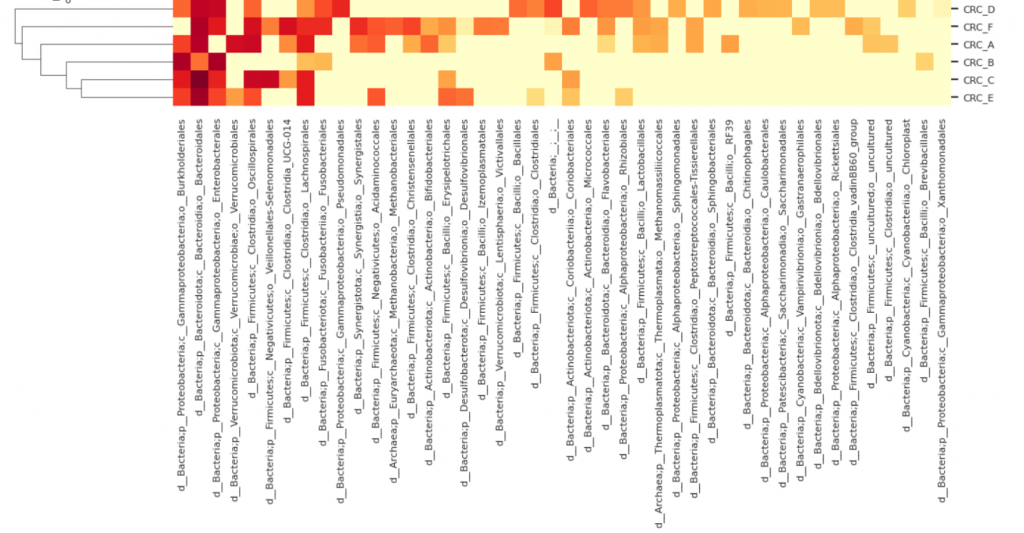
--p-level 4 / --p-cluster samples

--p-level 4 / --p-cluster features
發現差別了嗎? 兩者分別對樣本/特徵ID進行聚類。
本篇使用到的輸入/輸出檔案 :Input : table-dada2-240.qza、taxonomy.qza、col_table_4.qzaOutput: col_table_4.qza、heatmap_4.qzv
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
