後來發現這系列不只有讀者,竟然還有response之後,今天就繼續來談一下Whippet.jl這個工具。首先得先說聲抱歉,最近由於想多花點時間陪陪家人,所以有時候一天能分給這個鐵人賽的時間就相對有限,也因此肯定會有很多地方沒談到。所以我非常歡迎,如果您有興趣,我們可以針對這個點繼續聊。

作者們覺得過去的方法中,有些雖然能大幅降低計算時間跟所需計算資源了,但他們卻只能用在找比較簡單的AS(像intron-exon-intron這種),用來找比較複雜一點的AS(像下圖)就很不準了。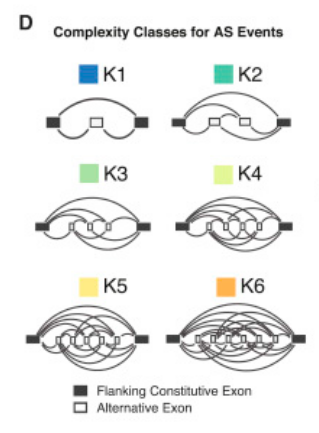
為了公平比較,作者們分別用實際跟模擬的Data以及不同的演算法工具來比較,用實際Data的部份,答案是根據RT-PCR的結果;模擬Data的部份,則是用參考基因序列加上外部工具及自己寫的工具產生模擬資料,結果顯示,Whippet這個演算法不僅錯誤率低,效能也好。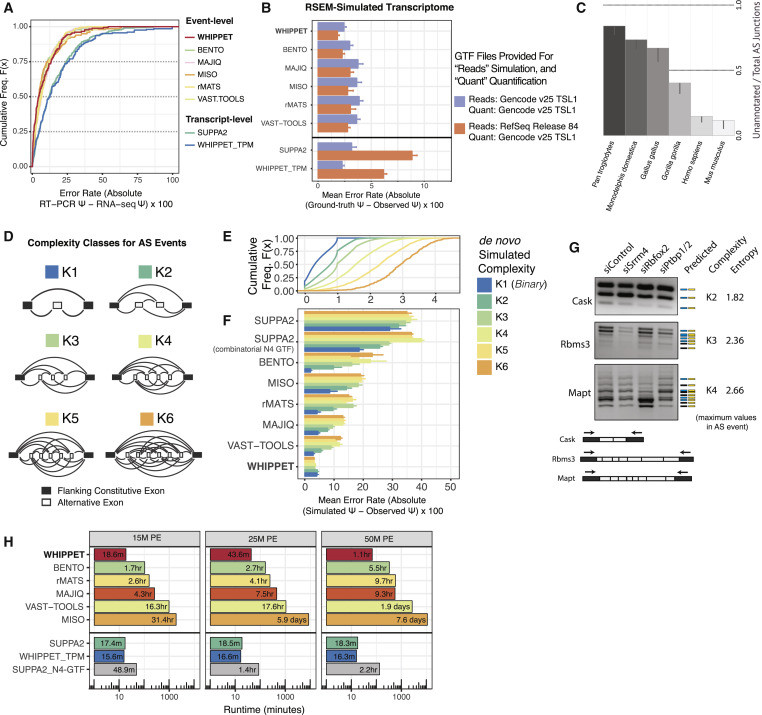
在贏得了與其他工具之爭後,作者們繼續拿Whippet去看看在正常組織中RNA的AS分佈與在腫瘤組織中的AS分佈有何不同。作者們發現,在腫瘤組織中所呈現的AS分佈顯示,複雜度高的AS events會比在正常組織中來的多。經過統計分析後,找到一些基因在腫瘤組織中其AS的數量顯著比在正常組織中的數量高很多,於是經過功能性分析(functional analysis)後,找出這些基因所對應的功能及所參與的生物功能路徑。
在更後續的研究中,作者們利用Whippet找到了更多的AS,同時也發現了裡頭有很多AS其實在演化上挺保守的,說明這些AS扮演著一定的重要功能。
Whippet.jl的使用強烈建議直接使用 Julia v0.6,我自己這兩天過測試之後,如果不想自己跳下去改原作者的code的話,只有用0.6版的Julia才有辦法安裝。如果可以的話,在產生contiguous splice graph index的時候,盡量採用BAM + GTF的方式。
感謝閱讀。

請教一下在實際跑過那麼多 project 後,
Julia 在套件版本相容的問題會不會很麻煩?
或著說,他能有像 python 這樣的 virtual env 可以使用?
目前沒看過有類似python底下的pyenv或是virtual env這種東西,不過我自己會把這些吃不同版本Julia的套件用Docker去包 XD
docker呀,然後再學 Kubernetes 管理嗎(倒...