上一篇我們的基因體時代-AI, Data和生物資訊 Day18-基因變異的檔案格式VCF上一篇介紹當我們取得每個人定序的比對資訊後,就是想要了解他跟其他人的差異,也就是所謂的基因變異,這時候就需要新的資料格式來做紀錄,目前為止,這樣的比較大多是跟參考基因組的序列比較來看差異性,而在人類基因組計畫後,擁有比較穩定的第二代定序工具後,美國便開始有更大型的定序計畫來探討相關議題,因此對於人跟人的差異性有更多的理解,VCF格式便是在執行這類大型定序計畫時為了儲存變異資料而提出來的格式,這格式主要分為兩個部分,第一部分為Header,每一行由#開始,裡面會註明所儲存的資料遵循之VCF版本、以及裡面所使用的欄位細節,第二部則是由8欄所組成的資料本身,每一行為一筆資料。
這邊接著介紹有什麼工具可以用來分析和處理VCF檔案,下面把一份VCF資料長的樣子顯示出來!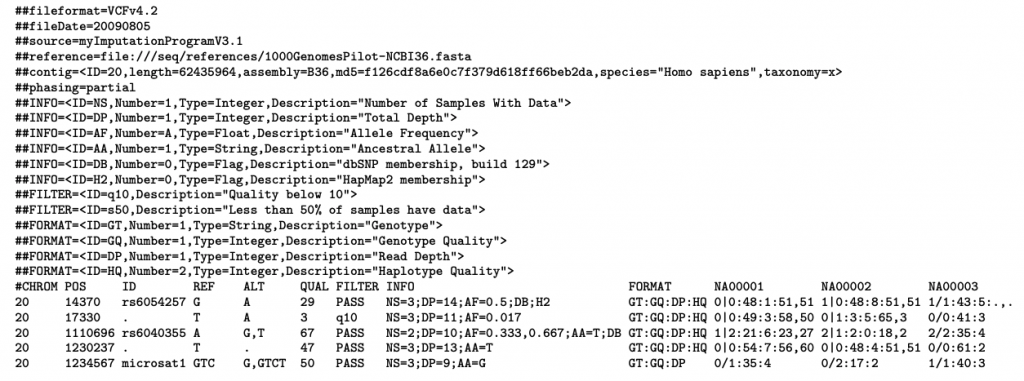
在linux或是unix環境的命令行下,有許多原生的程式是能很高效率地去做字串處理,以下從簡單到複雜來羅列:
這幾個程式其都是歷史悠久的底層程式,可以從中一窺一開始程式都在解決什麼樣類型的問題(其實就是字串處理問題。),zcat是有時候這類生物資訊相關的資料通常都會壓縮過,這時候就可以用zcat,在不解壓縮的情況下看一下裡面的資料,往下head/tail/cut都是單純的工具,head和tail方便你可以只顯示頭幾行或是尾幾行的資料,cut則是可以讓你輕鬆地去把一個欄位資料中,挑選特定的欄位形成新的資料,grep、sed和awk則是相對比較複雜,其每一個都是有其可擴充性,基本上都是搭配所謂的正規表達式(regular expression)的語法在整個程式語法之中。正規表達式其實是在計算機科學中前期很重要的一個概念,可以看一下這部影片。目前在字串處理的語法中基本上都會用到正規表達式,不論是在用R、Python、Javascript、shellscript都會用到,而grep、sed和awk中則是會利用正規表達式來進行相關字串處理。
這邊假如需要好好深入學習的話,推薦下面的書,可以邊查邊做:
從可以熟悉一下命令行指令的語法和觀念,這邊可以先從所謂的wildcard的概念學起,可以幫助往下走
grep本身就是在linux/unix命令行會用到的指令,但因其可以搭配正規表達式(regular expression)來做更複雜的搜尋
從使用grep來熟悉所謂的正規表達式後,就可以往下去觸碰sed,再來是awk,本質上awk相對複雜一點,他本身就是一個完備的程式語言,但觀念上跟字串處理是組合再一起的。

下一篇會來更近一步的介紹這三個工具,他們分別在三個不同層次上用來處理相關問題:
相關閱讀:
Day08-一些常用Linux指令-3-7(cat, tac, head, tail, vi, sed)
https://genome.ucsc.edu/FAQ/FAQformat.html
https://en.wikipedia.org/wiki/BED_(file_format)
這個月的規劃貼在這篇文章中我們的基因體時代-AI, Data和生物資訊 Overview,也會持續調整!我們的基因體時代是我經營的部落格,如有對於生物資訊、檢驗醫學、資料視覺化、R語言有興趣的話,可以來交流交流!
