上一篇我們的基因體時代-AI, Data和生物資訊 Day17-分析定序檔案格式SAM, BAM的工具上一篇接續介紹怎麼來利用和分析BAM/SAM檔案內的基因序列比對資料,其中最主要的工具是samtools,可以利用其瀏覽BAM/SAM檔案,做基本之描述統計分析比對結果,近一步分析檔案內的細節,直接做基因變異之分析,BAM/SAM檔案之排序、合併、格式微調,建立索引或是檔案格式之轉換,當然也是可以使用R裡面Bioconductor或是Python裡面的工具來做處理(雖然這兩個語言環境中也是調用samtools來做運算)。
人類的染色體到底有多少序列,到底人跟人之間差異有多大,人有多少個基因,這些知識其實都是在最近這二十年才比較多理解,從人類基因組計畫的關係,我們開始擁有價格比較親民的定序技術和開發出一系列的分析算法,但只取得一組所謂的人類參考基因組並不能讓我們知道我們人跟人的差異,所以人類基因組計畫後,還有許多更大型的定序計畫,如1000人定序計畫1000 Genome Project,在這個計畫中總共定序2504個人,這些人來自26種族包括來自非洲、東亞、歐洲、南亞、美洲,下面地圖中的地方是這個計畫有收錄的人種來自之地區: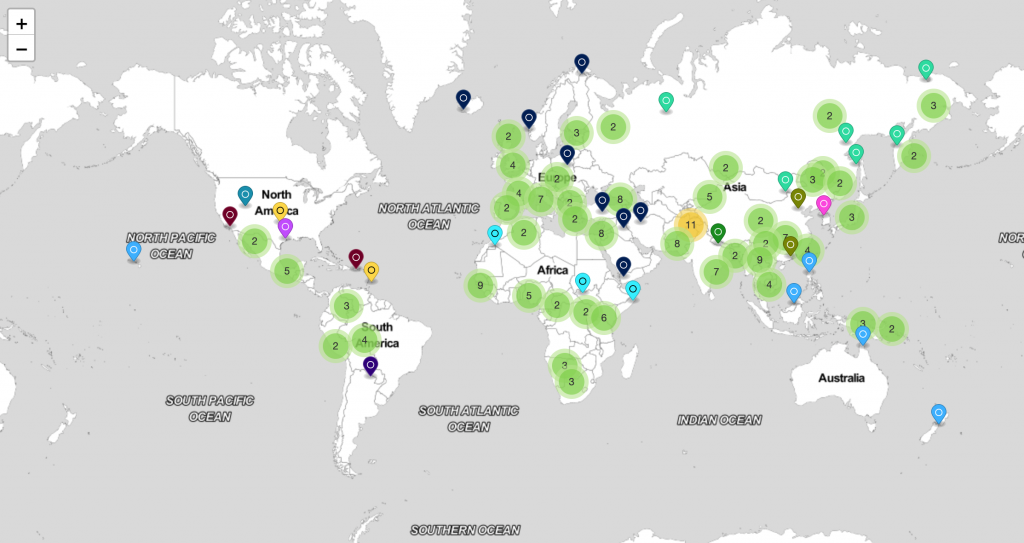
一個人的基因跟目前人類參考基因組比較來看,平均會有4百萬到5百萬的位置之差異,大概有百分之99.9%的變異都是單鹼基的變異,但還是有大概2100到2500個比較長的變異(structural variation, > 500bps),下面這張圖比較了不同種族跟參考基因組之差異,裡面可以看到歐洲人跟人類參考基因組的差異比較少(當然,因為人類參考基因組有80%來自一個歐洲血統的受試者),大部分在一個人之基因序列中觀察到跟人類參考基因組之差異是>0.5%的,只有大概4萬到5萬的位點是在族群中小於0.5%的。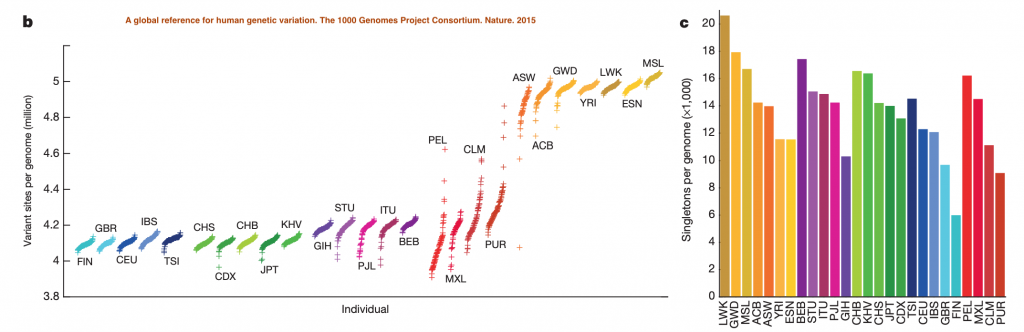
所以有了定序資料的比對資訊BAM,我們就可以從中來分析所謂的變異資訊,這時候輸出的資料也會有個統一格式,叫做Variant Calling Format(VCF),這個格式也是在執行1000 Genome Project時候所提出的,為了解決如何去儲存和方便後續分析所使用的資料格式,一開始是由1000 Genome Project所維持,後來則由Genomic Data Toolkit所維持,詳細的資料規格也可以在此一覽。
範例的VCF檔案長得如下: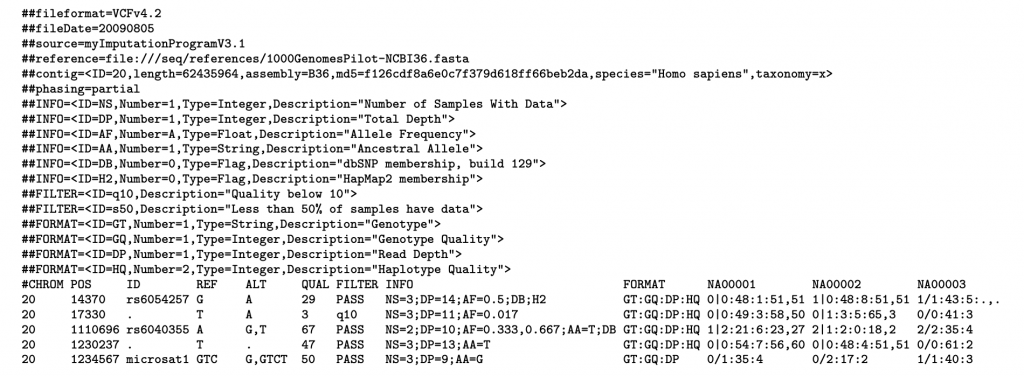
整個資料也可以分成兩個部分:header和variants
header的部分主要是用來記錄這個資料的一些meta資訊如這分資料是使用哪個版本的VCF、裡面所用的樣本模式、等位基因的模式、篩選的方式等等。
variants本身的資料則是由8個欄位所組成,分別是:
*#CHROM:第一個欄位就是註明這個變異為在哪一個染色體上面,通常是根據參考基因組來自於美國或是歐洲系統,有的會用1、2、....,有的則是chr1, chr2.....
*POS:這個變異點第一位置在此染色體的位置,是1-based的系統
*ID:通常是使用rs ID
*REF:對照參考基因組上的序列
*ALT:此變異實際的變化序列
*QUAL:通常是此位點的定序品質,要是這是個缺失變異的話,品質則是−10log10(位點之Phred品質參數)
*FILTER:是否有通過篩選條件
*INFO:這個欄位基本上就是整個資料中變化差異最多的,基本上可以擴充非常多資料,比如加入下表的一些資訊:
閱讀參考:
The Biostar Handbook
Human Molecular Genomics, 15th
Frazer, K. A., Murray, S. S., Schork, N. J., & Topol, E. J. (2009). Human genetic variation and its contribution to complex traits. Nature Reviews Genetics, 10(4), 241–251. doi:10.1038/nrg2554
The 1000 Genomes Project Consortium. A global reference for human genetic variation. Nature 526, 68–74 (2015). https://doi.org/10.1038/nature15393
這個月的規劃貼在這篇文章中我們的基因體時代-AI, Data和生物資訊 Overview,也會持續調整!我們的基因體時代是我經營的部落格,如有對於生物資訊、檢驗醫學、資料視覺化、R語言有興趣的話,可以來交流交流!
