上一篇我們的基因體時代-AI, Data和生物資訊 Day19-分析和處理基因變異的檔案格式VCF的工具上一篇介紹在處理VCF檔案時,可以用的工具,先從最簡單的文字處理工具,如在命令行中的cut, grep, sed, awk,接者往下比較複雜的處理則可以使用bedtools, bcftools, vcftools來做運用。
再開始處理比較複雜的基因數據,就必須要先介紹相關跟基因數據相關的註釋資料格式,可以參考聖塔克魯茲加利福尼亞大學(University of California, Santa Cruz)的基因體瀏覽器網站(UCSC Genome Browser)之頁面。
UCSC genome browser和基因註釋相關的資料格式有什麼關係呢?這邊就要提到一點點歷史和國外生物資訊研究的風氣,相對於國內生物醫學的研究,國外往往講求研究資料的共享,就像是美國國家生物技術資訊中心(National Center for Biotechnology Information)已經是目前世界上最重要的生物資料庫,當人類基因體計畫的開始,其實美國國家生物技術資訊中心就開始把相關資料置放在網站上供世界各地的科學家使用,當時人類基因組的資料庫就是置放在NCBI資料庫GeneBank之中,而除了美國的NCBI資料庫外,歐洲也有另一個生物資訊中心叫做歐洲分子生物學實驗室(European Molecular Biology Laboratory, EMBL),也建立了一個重要的公開資料庫Ensembl,而當時聖塔克魯茲加利福尼亞大學(University of California, Santa Cruz)的研究人員便決定也建立一套相關網站,來提供視覺化以及相關基因數據的瀏覽功能,當時在2000初的版本外觀如下:
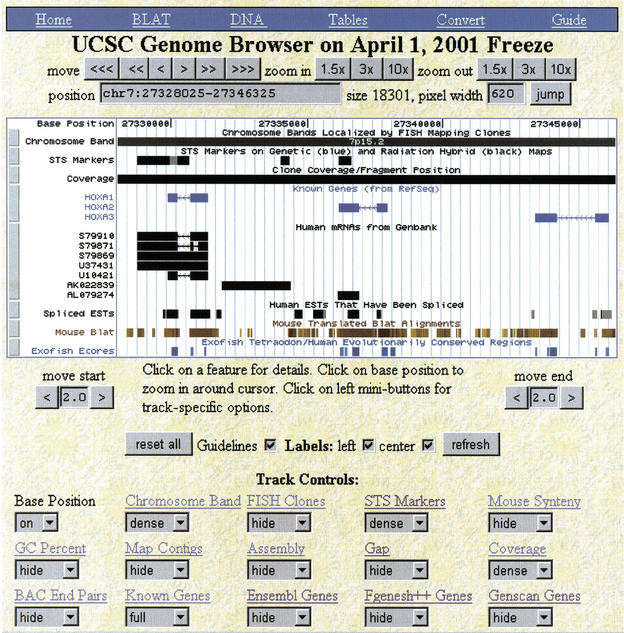
當時,這個網站就叫做UCSC Genome Browser,其初始的視覺化代碼其實就是用來呈現線蟲(C. elegans)定序資料中,基因模型的一個C語言的程式,其可以把資料呈現如下: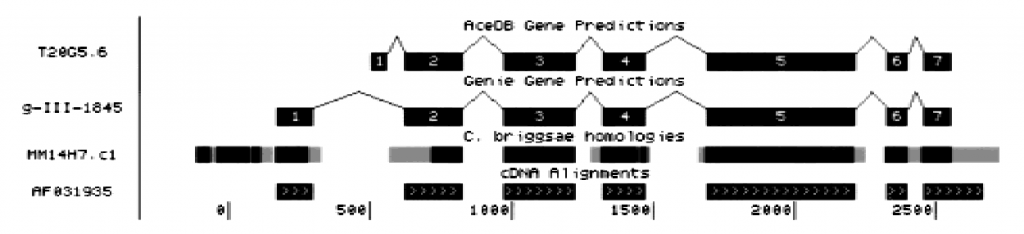
這樣的目的是為了提供科學家一個更好理解基因序列資訊的方式,我想正常人類應該無法從單純的一大串ATCGCGCTAGCTAGCTA中直接理解它的意義,所以基因組瀏覽器的目的就是提供一個有生物意義層次的架構:從染色體、外顯子、基因、代謝路徑等等層次去讓生物學家來瀏覽這些基因序列資料。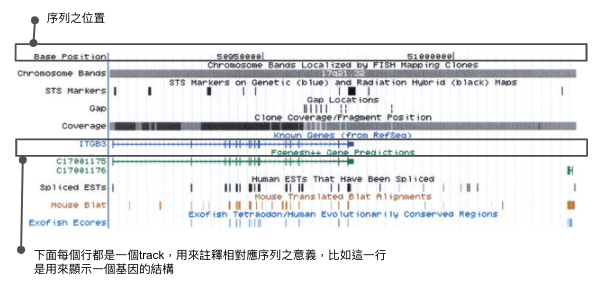
為了呈現這種註釋效果,當時就使用所謂的track的模式,如同上面這張圖,每一個track之間共通的關係就是在序列上的位置,然後其本身可以疊加不同意義,比如上圖中圈起來的track,就是用來註釋一個基因的結構,哪邊是外顯子,哪些是內顯子,而如今這個方法已成為基因註釋的主流方式,在UCSC Genome Browser在基因註解之track已經超過上千種了。
那呈現這樣的視覺化資訊,要搭配相對應的資料格式,這時候就出現了所謂的BED檔案格式(Browser Extensible Data),其便是為了對應這樣視覺話需求的資料,當初最簡單的版本就是3欄的數據,第一欄chrom:染色體名稱,第二欄chromStart:起始位置(0-based index system),第三欄:chromEnd:結束位置(0-based index system),除了這三欄外,還有九個延伸的欄位:
所以目前一份BED格式的資料會長的如下:
track name="ItemRGBDemo" description="Item RGB demonstration" visibility=2 itemRgb="On"
chr7 127471196 127472363 Pos1 0 + 127471196 127472363 255,0,0
chr7 127472363 127473530 Pos2 0 + 127472363 127473530 255,0,0
chr7 127473530 127474697 Pos3 0 + 127473530 127474697 255,0,0
chr7 127474697 127475864 Pos4 0 + 127474697 127475864 255,0,0
chr7 127475864 127477031 Neg1 0 - 127475864 127477031 0,0,255
chr7 127477031 127478198 Neg2 0 - 127477031 127478198 0,0,255
chr7 127478198 127479365 Neg3 0 - 127478198 127479365 0,0,255
chr7 127479365 127480532 Pos5 0 + 127479365 127480532 255,0,0
chr7 127480532 127481699 Neg4 0 - 127480532 127481699 0,0,255
BED檔案,其實就是一群定義好的區域,這區域在基因體上有相對應的位置,那這時候就可以用很多工具來做處理,比如bedtools,這軟體的解說頁很清楚的表達了他怎麼處理不同之BED檔案: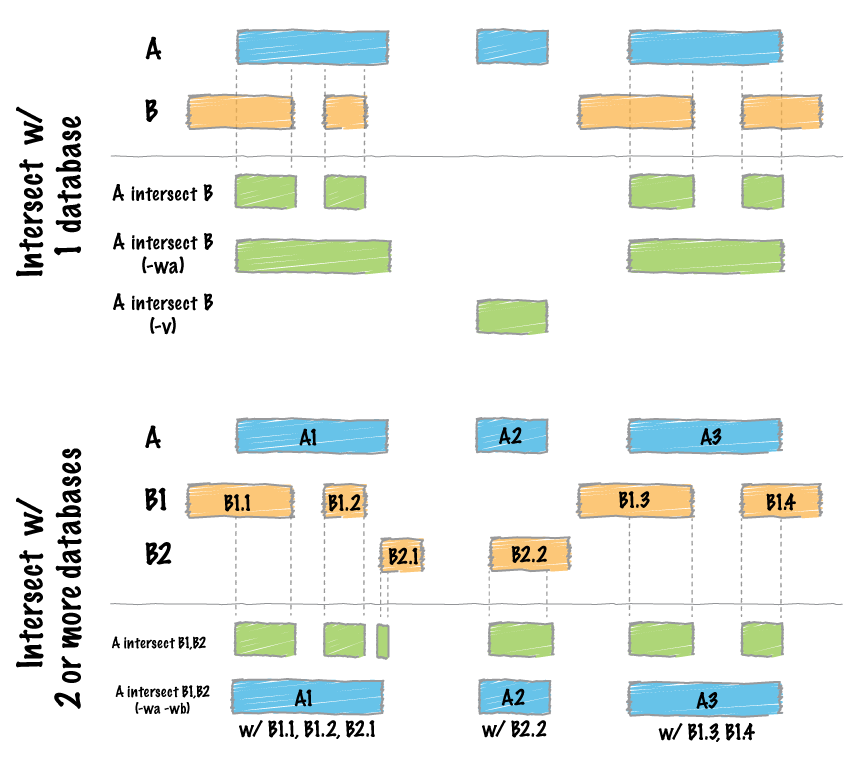
怎麼把這樣的操作轉換成生物意義來解釋呢?
當然,bedtools本身有很多功能,目前有41個函數,這些函數可以區分為幾類功能,如基本區域篩選(intersect, window, closest, coverage, mapgenomecov, merge, cluster, complement, shift, subtract, slop, flank, sort, random, shuffle, sample, spacing, annotate)、多檔案比對(multiinter, unionbedg)、格式轉換(bamtobed, bedtobam, bamtofastq, bedpetobam, bed12tobed6)、BAM處理(multucov, tag, pairtobed, pairtopair)、統計分析(jaccard, reldist, fisher)、雜類(overlap, igv, linkns, makewindows, groupby, expand, split, summary)。
參考閱讀:
Kent WJ, Sugnet CW, Furey TS, et al. The human genome browser at UCSC. Genome Res. 2002;12(6):996-1006. doi:10.1101/gr.229102
User-friendly, scalable tools and workflows for single-cell RNA-seq analysis. Nat Methods. 2021 Apr;18(4):327-328. doi: 10.1038/s41592-021-01102-w. PMID: 33782609; PMCID: PMC8299072.
Towards complete and error-free genome assemblies of all vertebrate species. Nature. 2021 Apr;592(7856):737-746. doi: 10.1038/s41586-021-03451-0. Epub 2021 Apr 28. PMID: 33911273; PMCID: PMC8081667.
Speir ML, Bhaduri A, Markov NS, Moreno P, Nowakowski TJ, Papatheodorou I, Pollen AA, Raney BJ, Seninge L, Kent WJ, Haeussler M. UCSC Cell Browser: Visualize Your Single-Cell Data. Bioinformatics. 2021 Jul 9:btab503. doi: 10.1093/bioinformatics/btab503. Epub ahead of print. PMID: 34244710.
Gonzalez JN, Zweig AS, Speir ML, Schmelter D, et al. The UCSC Genome Browser database: 2021 update Nucleic Acids Res. 2021 Jan 8;49(D1):D1046-D1057. PMID: 33221922 PMCID: PMC7779060
這個月的規劃貼在這篇文章中我們的基因體時代-AI, Data和生物資訊 Overview,也會持續調整!我們的基因體時代是我經營的部落格,如有對於生物資訊、檢驗醫學、資料視覺化、R語言有興趣的話,可以來交流交流!
