實務上由於往往無法從文獻、學長姊(X 中得知,
究竟得到的序列是不是含有 Adapter、Barcode,
於是嘗試不同的剪切片段就是很重要的步驟。
想像在平行時空下,[Day 07] 的我們獲得了三張便條紙,
上頭寫著每條序列只留下 :
0~250 nt 片段 (總長 250 nt,都不切)
qiime dada2 denoise-paired \
--i-demultiplexed-seqs demux.qza \
--p-trim-left-f 0 \
--p-trim-left-r 0 \
--p-trunc-len-f 250 \
--p-trunc-len-r 250 \
--o-table table-dada2-250.qza \
--o-representative-sequences rep-seqs-dada2-250.qza \
--o-denoising-stats stats-dada2-250.qza \
--p-n-threads 8
10~250 nt 片段 (總長 240 nt)
qiime dada2 denoise-paired \
--i-demultiplexed-seqs demux.qza \
--p-trim-left-f 10 \
--p-trim-left-r 10 \
--p-trunc-len-f 240 \
--p-trunc-len-r 240 \
--o-table table-dada2-240.qza \
--o-representative-sequences rep-seqs-dada2-240.qza \
--o-denoising-stats stats-dada2-240.qza \
--p-n-threads 8
30~240 nt 片段 (總長 210 nt)
qiime dada2 denoise-paired \
--i-demultiplexed-seqs demux.qza \
--p-trim-left-f 30 \
--p-trim-left-r 30 \
--p-trunc-len-f 210 \
--p-trunc-len-r 210 \
--o-table table-dada2-210.qza \
--o-representative-sequences rep-seqs-dada2-210.qza \
--o-denoising-stats stats-dada2-210.qza \
--p-n-threads 8
一樣做視覺化輸換 (.qzv),這次只做 stats-dada2.qza。
(這邊指令以總長240 nt 為例)
輸入 :
qiime metadata tabulate \
--m-input-file stats-dada-240.qza \
--o-visualization stats-dada2-240.qzv
完成後會顯示 :
"Saved Visualization to: stats-dada2-240.qzv"
不合格,Non-Chimeric 通過條數過低 : 0~250 nt 片段 (總長 250 nt,都不切)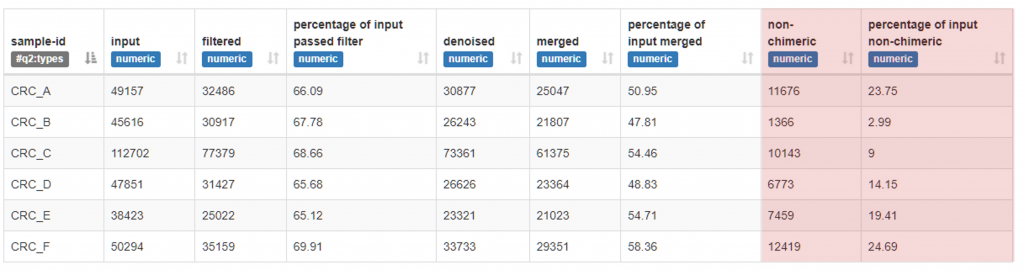
可以發現到如果都沒有切 (或起始切太少),
從 merged 到 non-Chimeric 出現驟降(多數剩下不到1%),
代表原始序列起始端可能含有 Adapter 或 Barcode,
讓 DADA2 誤以為這是 PCR 造成的嵌合體(chimera)
(聚合酶表示無辜) :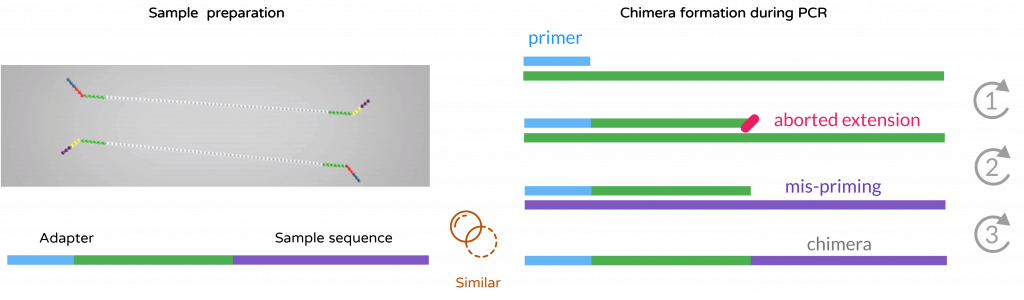
殘餘的 Adapter 未切除乾淨(左),被 DADA2 誤認是大大大大大 Primer的錯誤黏合(右)
圖源參考Illumina, ISB Microbiome Course 2020, flaticon重組繪製。
合格 : 10~250 nt 片段 (總長 240 nt)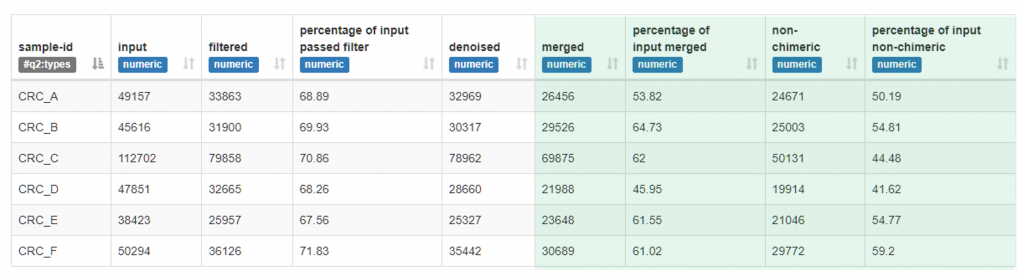
這個很乖,是合格的,實務上通常 Filter 會是掉最多的,後續篩選都不會有過大的降幅。
不合格,Merged 通過條數過低 : 30~240 nt 片段 (總長 210 nt)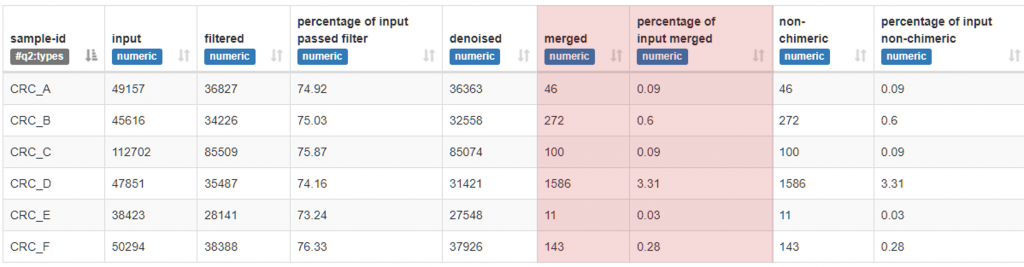
這是剪切過多造成的結果,
從 denoised 到 merged 出現驟降(剩下不到10~30%,甚至 < 1%),
代表原始序列起始端與末端剪接過多,
雙尾定序 (Paired-end) 的特性就是最後會組裝在一起 [Day 05],
若是剪切時切除了兩序列重疊的片段,
就會整個樣本的序列都找不到另一半(單身樂園?,
最後大家都會被 DADA2 狠心淘汰 (Q
生物資訊的資料分析做實驗相同,都是需要不斷試錯,
最後,快快樂樂的拎著10~250 nt 片段 (總長 240 nt) (即[Day 08]產出的三個.qza檔案),
繼續 QIIME2 分析的旅程~
本篇使用到的輸入/輸出檔案 :Input : demux.qzaOutput: stats-dada2-250.qza、stats-dada2-240.qza、stats-dada2-210.qza、stats-dada2-250.qzv、stats-dada2-240.qzv、stats-dada2-210.qzv
本篇文章同步刊載於科學毛怪部落格 PetSci Blog。
