在 [Day 05] 的 16S rRNA 介紹中,
曾經提及 NGS 的技術限制在單條超過 300 bp 品質就會急速下降,
使得 16S 定序中,以 V3-V4 區域(~470 bp)雙尾定序為主,
細心的你如果有注意到,會發現在 [Day 11] 出現的分類柱狀圖,
其物種階層多半停留在 Genus 階層,鮮少見到 Species 階層,
主因就是 V3-V4 對於物種的解析度(resolution)的限制 :

[Day 11] 的分類柱狀圖,分類結果多半停留在 Genus 階層。
若能夠一股作氣定序 V1-V9 (~1500 bp)就皆大歡喜了,
難道沒有更好的方法了嗎 ? 有的,就是第三代定序 !
有別於短片段 (0.25 kb) 在 [Day 03] 提及由 Illumina 幾乎獨佔,
截至目前長片段 (5~1000 kb) 定序
則有 PacBio / Oxford Nanopore 兩大公司激烈競爭中,如同前端框架 React.js 與 Vue.js 之爭一樣火熱,
礙於研究生時間寶貴 (剛好用過 PacBio ),
之後會以 PacBio 當作範例。
(Logsdon, G. A., Vollger, M. R., & Eichler, E. E., 2020)
Pacific Biosciences (PacBio) 提出
將一段髮夾狀 adapters 連接於目標序列兩端,
使其成為環狀結構,稱為 SMRTbell ,再配上引子與聚合酶 :
並將每個 SMRTbell 置於 SMRT cell 上獨立的小洞洞(ZMWs),
讓聚合酶乖乖地合成,
合成過程中同時讀取帶有螢光標定的 dNTP 發出的訊號進行定序 :
因為 SMRTbell 呈現一個 Loop 形狀,這讓聚合酶可以瘋狂轉圈圈合成,
以此增加定序的準確度,最終產生高品質的序列 (HiFi read) :
稱為 Circular Consensus Sequencing (CCS),
另外也有 Continuous Long Read (CLR) 適用於 >50kb 的定序方式。
Reference : PacBio
也推薦讀者可以去看看 Oxford Nanopore ,
是利用電訊號的超酷創新定序方式,
也附上兩家的比較圖 :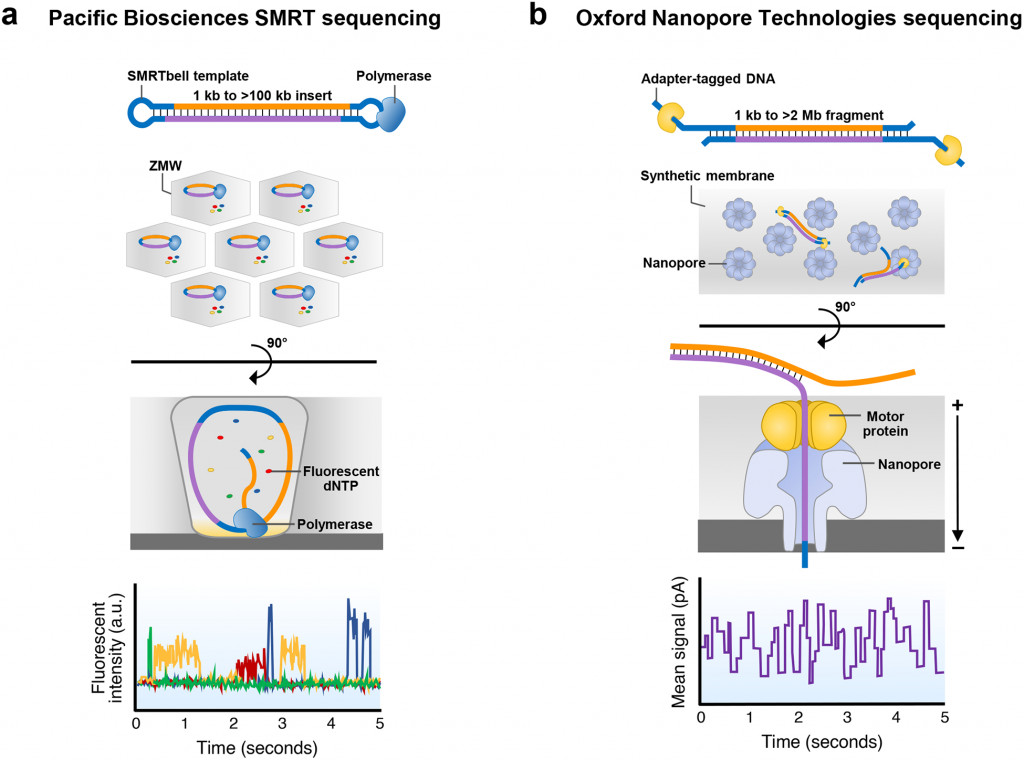
(Logsdon, G. A., Vollger, M. R., & Eichler, E. E., 2020)
畢竟三代定序是近幾年來的新技術,
目前 TGS 平均定序價格仍較次世代高些,
對應的分析軟體也正在快速發展尚未完善,
更別說繁中的技術文章了,這也凸顯本系列文章的價值ヽ(✿゚▽゚)ノ,
之後會採用 PacBio SMRT Sequel II HiFi 16S rRNA Full Length (V1-V9) 做為範例。
