Kyoto Encyclopedia of Genes and Genomes 緣起於 1995 年,由京都大學化學研究所教授金久實提出,最大的特色在於大量人工繪製的生物代謝途徑圖。
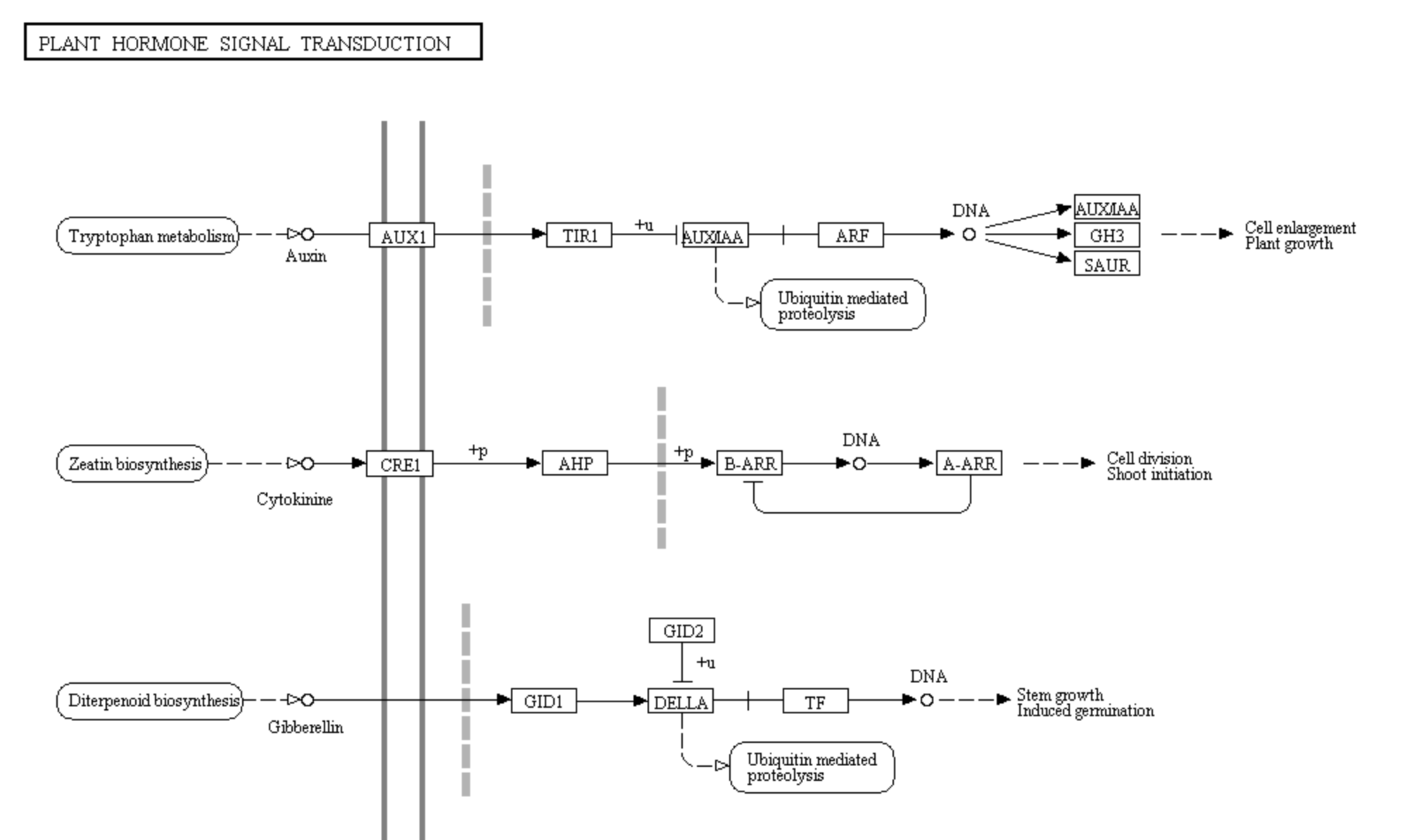
每張生物途徑圖都是基於文獻繪製
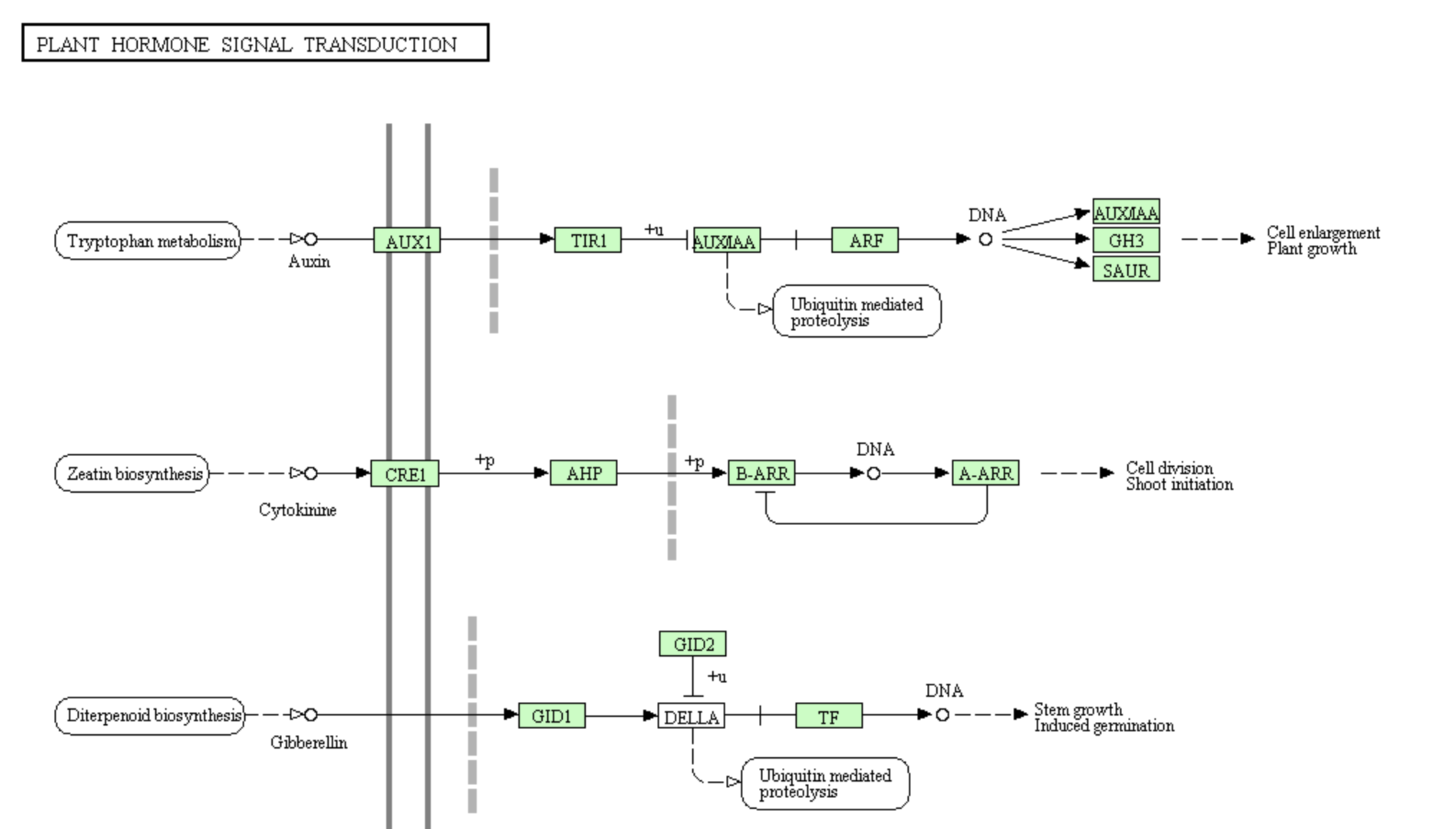
隨著物種的不同,基因體中可能不具有該途徑上的所有基因。因此針對不同的物種,會有一張最完整的途徑圖作為基底,其中代表酵素的方框中會以淺綠底色作為標記,用以表示該物種中已經確定有紀錄的基因。
研究者可能想要一次取得一張人工繪製的生物途徑途中所有的基因知識別代號,KEGG 提供了 REST API 讓研究者用程式存取需要的資訊,以下便提供使用 BioPython 來介接 KEGG API 的簡單腳本。
KEGG API 的網址直接用瀏覽器開啟,就可以看到如下的格式:

此處使用 get 方法,加上途徑圖代號就可以取得一張生物途徑圖上的所有資訊。途徑圖代號就是生物學名縮寫 (苦瓜學名 Mormodica charantia => mcha) 加上途徑圖編號 (04075)。前幾行的 ENTRY (入口)、NAME (名稱)、CLASS (分類)、PATHWAY_MAP (途徑圖) 是最基本的這張途徑圖的基本資訊。DBLINKS 可以看到有連結的資料庫的資訊,比如此處顯示了有關的基因本體論代號。ORGANISM 是物種的學名全名及俗稱。
GENE 才是我們所在意的重點,這部份由左至右分成三段,最左邊的數字前頭加上 LOC 就是 NCBI Gene database 的 Gene Symbol,中間是目前用以稱呼該基因的常見名稱,右邊則是 KEGG 本身將該基因歸類位置的代號。
因此我們的目標就是下列三步驟:
from Bio.KEGG import REST
pathway_list = ["mcha04075"]
# 將想要存取的生物途徑圖代號以字串形式準備在 list 中,實際上要批次存取時比較方便
for pathway in pathway_list:
kegg_id_list = []
# 用 biopython 提供的 REST 之 kegg_get 函式取得需要的資訊,其結果就是瀏覽器中可以看到的樣子,先存在 pathway_file 變數中待會再慢慢讀
pathway_file = REST.kegg_get(pathway).read()
# 準備一個 current_section 變數來確認程式讀到哪個章節了
current_section = None
for line in pathway_file.rstrip().split("\n"):
# 一行一行讀取得的文字,將每一行開頭前 12 個字元存為 section
section = line[:12].strip()
if not section == "":
current_section = section
if current_section == "GENE":
# 終於讀到 GENE 章節,取得該行是第幾個字才是 KO 代號
ko_index = line.index("[KO:")
# KO 代號後的 4~10 個字元就是我們需要的代號
kegg_id = line[ko_index+4:ko_index+10] #K13946
# 將 KO 代號通通存進 list 中,就算有重複也沒有關係
kegg_id_list.append(kegg_id)
# 將變數型態轉換成沒有重複內容的 set 在轉換回 list 型態,就可以把重複的內容剔除掉啦
kegg_id_list = list(set(kegg_id_list))
print(kegg_id_list)
後續再比對 Trinotate 中的 KEGG 欄位就可以畫熱圖啦!
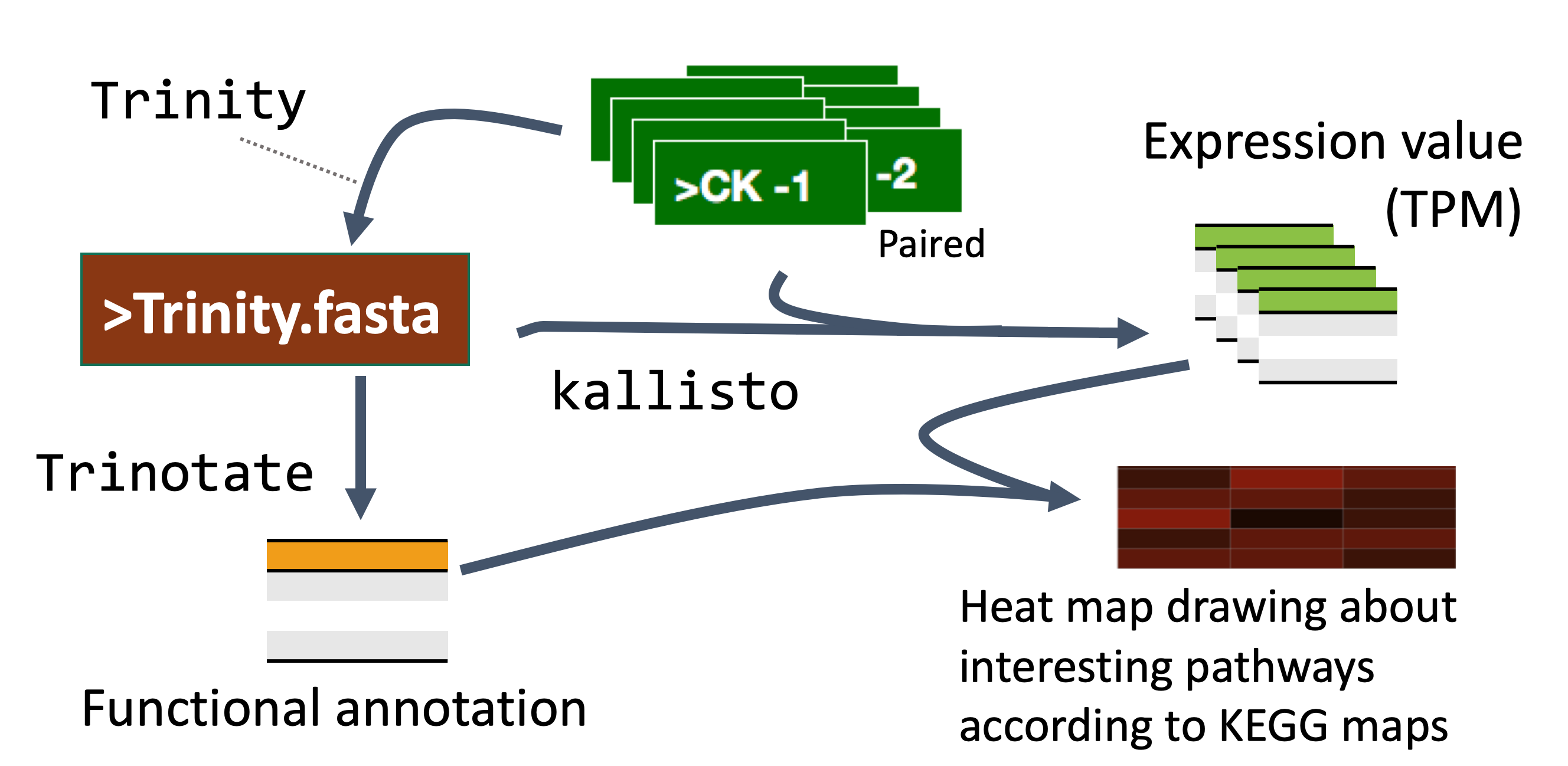
總地來說,人工繪製的生物途徑代謝圖讓讀者最有踏實的安全感,但是實際使用上如果不是研究透徹的模式生物的話,建議還是以參考用途為主或是保守地使用比較好~
如果有誤或是想了解更多請留言告訴我~
KEGG: Kyoto Encyclopedia of Genes and Genomes
KEGG: new perspectives on genomes, pathways, diseases and drugs
關於作者
謝晨 (Chen Hsieh),臺大園藝暨景觀學系研究所碩士。讀碩士前的興趣是懷著寫點程式妄圖解決農業問題的夢想參加比賽,拿了幾個黑客松與 Open Data 創新應用競賽的獎,卻都沒有勇氣將項目經營下去;研究所期間的興趣轉換成讀學術期刊的出刊電子報。靠著這些興趣當選 107 學年的臺大優秀青年,畢業後卻成了無業的實驗室居民。現在在農場旁的研究館辦公室寫點東西,希望可以跟世界分享生物資訊與園藝的樂趣!
感謝選擇匿名的朋友協助校閱初稿與提供意見,也敬請各位讀者不吝指教!
