上一篇我們的基因體時代-AI, Data和生物資訊 Day15- 組裝後的序列檔案格式SAM, BAM上一篇介紹基因定序後的檔案fastq,裡面是儲存一個個DNA序列的檔案,但是並不包含此片段來自哪的資訊,所以必須在經過一個分析流程叫做sequence alignment,這個步驟就必須要把這一個個片段跟參考基因組做比較,看這片段是來自於哪裡的,這時候的檔案資訊就會記錄每一個片段所比對到的位置,這樣的檔案格式就叫做Sequence Alignment Map(SAM),它的二進位壓縮版本就是BAM,而平常大部分都是使用BAM檔,因其可以節省空間。
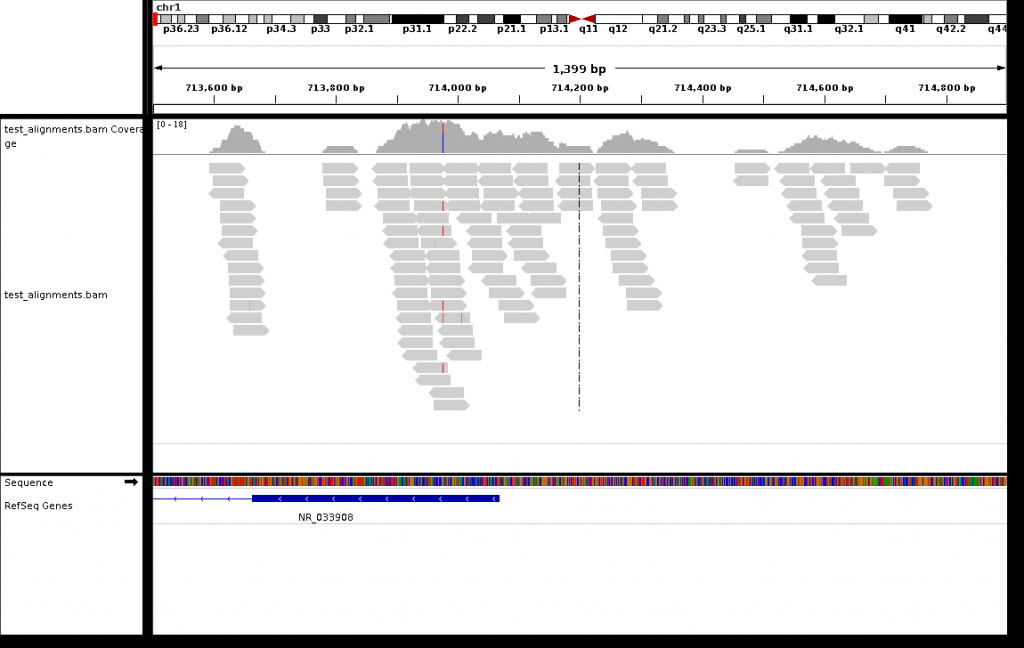
上面這個就是使用IGV來視覺化這個BAM檔看起來的樣子。目前最主流的兩個工具,一個是由美國NIH所開發的Integrative Genomics Viewer (IGV),算是這幾年都有在稍微更新,甚至有推出IGV.js的開發者工具,可以做鑲嵌式的應用。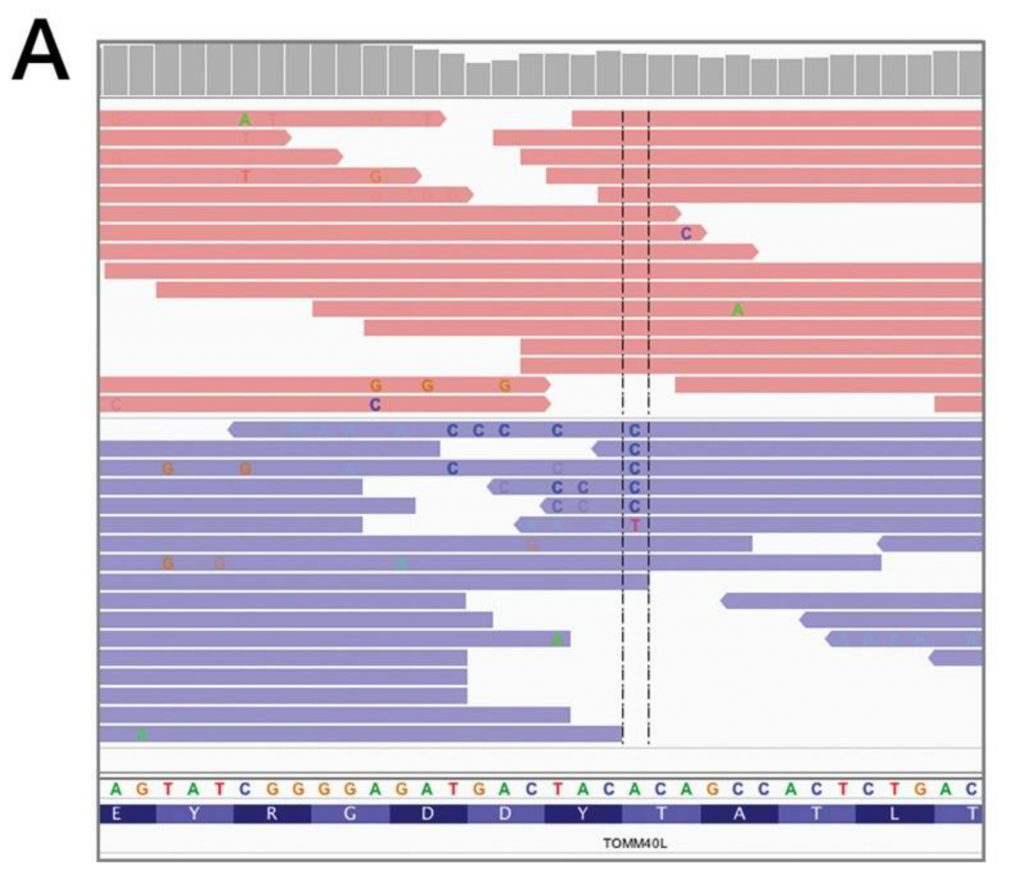
視覺化序列排列的檔案是有其必要的,像是上面這個範例,可以看到中間有一個區域,顯示跟參考序列不同,為C的符號,但是仔細一看,這個區域的片段似乎比對上去的狀況不是很好,很多都是非正確的比對,這就是常見在GC序列比例較多區域會看到的偽陽性的狀況。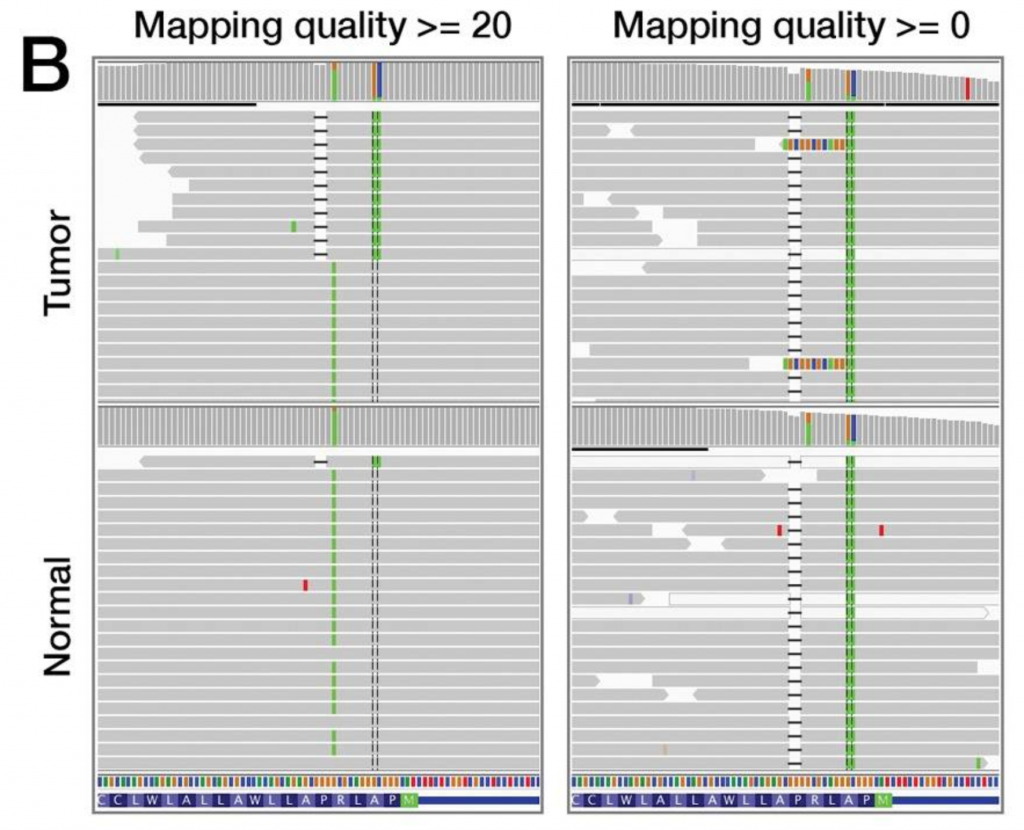
這邊則是另一個例子,左邊是被誤判的,右邊是比較正確的比對結果,這邊可能要帶入一點背景知識,基因的變異有兩種,一種是所謂的天生的叫做Germline mutation,另一種則是後天的變異,通常是指腫瘤細胞上面的變異,叫做somatic mutation,所以要判斷某個跟參考基因組不一樣的變異是先天的還是後天的,就必須要將正常組織和腫瘤組織的定序資料做比較,當變異只在腫瘤細胞上觀察到,那這就是體細胞變異,反之,兩個都有,則是所謂的先天的變異。左邊這個就是先把一些比對品質比較差的片段刪掉後,反而沒有發現某個地方是有基因缺失(deletion),這邊其實就顯示了目前根據參考基因組比對的一些偏差,當你定序的標的其和參考基因組差異越大時,有可能實際定序出來的片段,都無法正確比對到參考基因組上面,這也是目前第二代定序的一個局限所在。
這邊也是一個用視覺化工具發現在基因序列比對時有和真實情況相悖的情境,也顯示人類基因序列的變異是非常多樣的,這個區域是知名的EGFR基因上的一個變異叫做L747_A750delinsP,一個同時有基因缺失和插入的變異,這時候就必須要將他們合併再一起,否則會以為是兩個變異一起發生。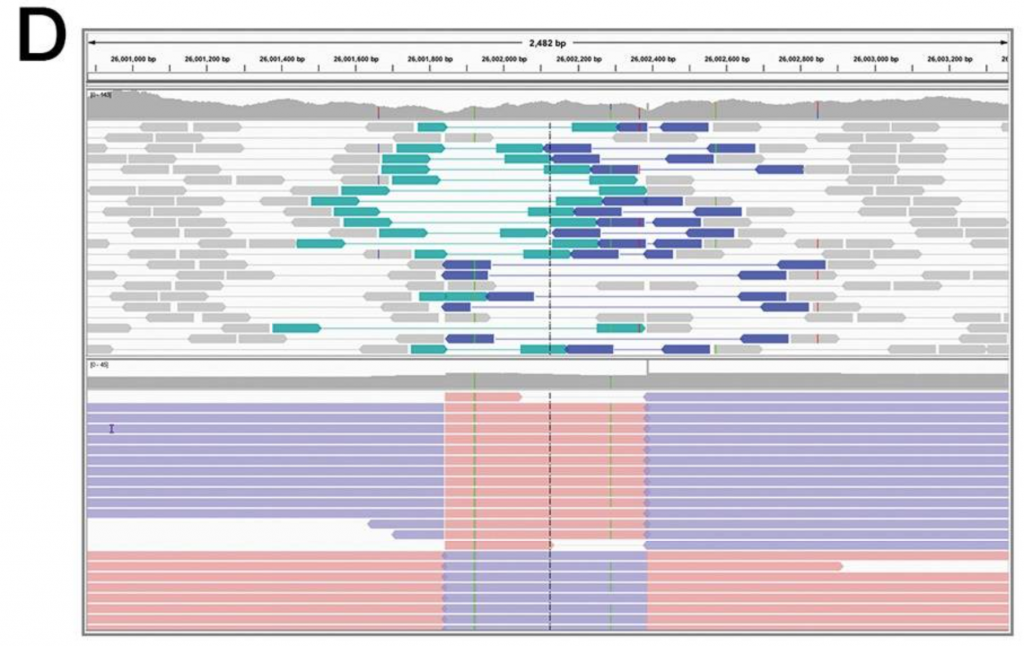
這邊的例子則是顯現第二代定序和第三代定序在找出比較大片段變異的能力差距,上面是第二代定序的資料,每個片段都比較短,下面則是第三代定序,每個片段相對來說都比較常,而這時就能由下面的第三代定序看到有一個500序列長度的反轉,這個就在第二代定序中無法看出。
除了介紹IGV視覺化的樣貌外,也展現了為何需要視覺化的必要性,同時也揭露人類基因變異的多樣性,下面則是另一款由英國Wellcome Sanger institute所開發的軟體叫做Artemis,相對於IGV,Artemis就聚焦在比較單純的BAM視覺化,比較複雜的分析就令闢軟體出來,它的介面相對的就是另一種更linux的風格。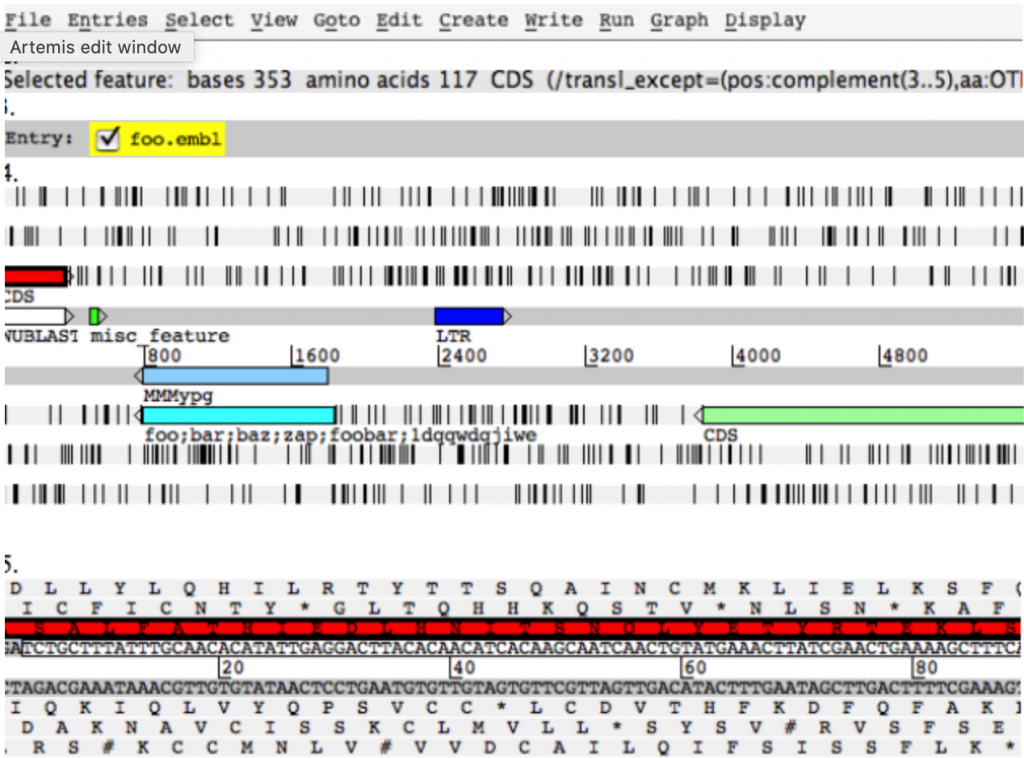
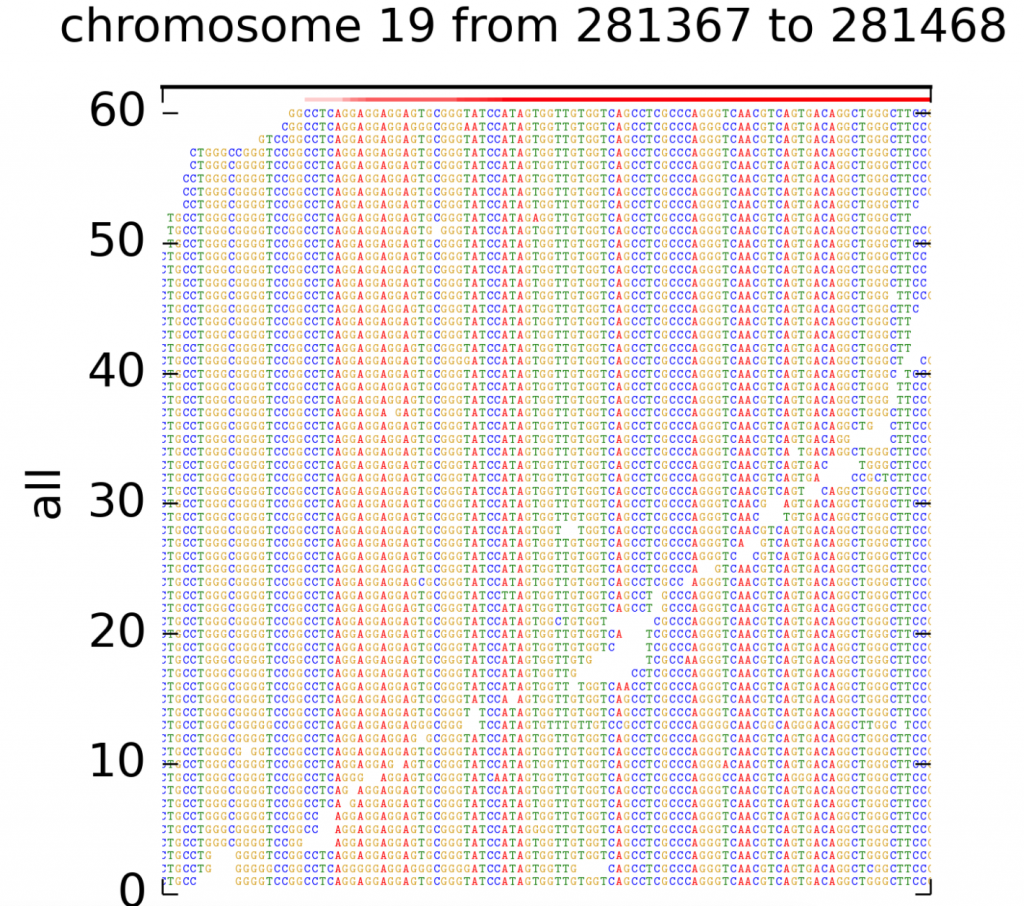
當然,也有一些專門做視覺化的小工具比如DrukBam,他可以幫忙做一些比較遠距的截圖和簡化的功能,可以繪製出如下的圖片。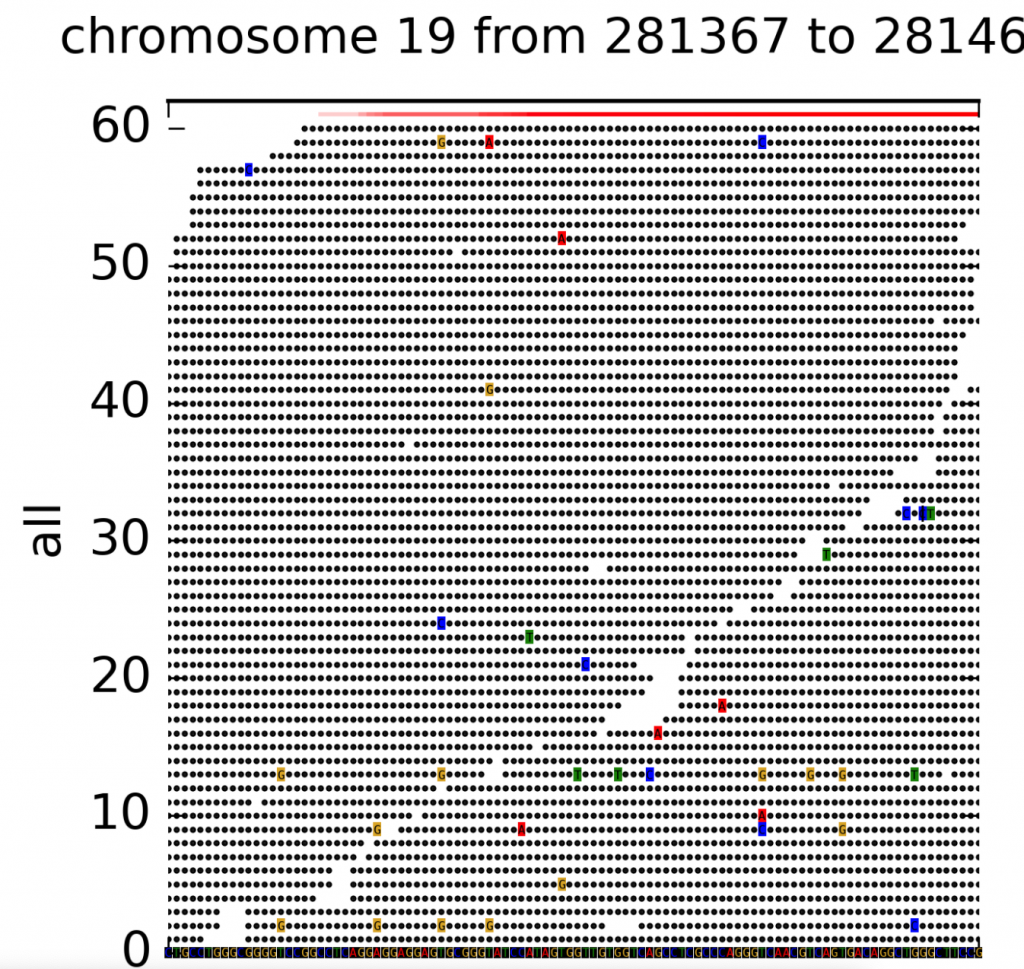
bamview也是一個由英國Wellcome Sanger institute所開發比較單純來看BAM的軟體
James T. Robinson, Helga Thorvaldsdóttir, Aaron M. Wenger, Ahmet Zehir, Jill P. Mesirov. Variant Review with the Integrative Genomics Viewer (IGV). Cancer Research 77(21) 31-34 (2017).
Carver T, Harris SR, Berriman M, Parkhill J and McQuillan JA. Artemis: an integrated platform for visualization and analysis of high-throughput sequence-based experimental data.Bioinformatics (Oxford, England) 2011;28;4;464-9
James T. Robinson, Helga Thorvaldsdóttir, Douglass Turner, Jill P. Mesirov. igv.js: an embeddable JavaScript implementation of the Integrative Genomics Viewer (IGV). bioRxiv 2020.05.03075499.
這個月的規劃貼在這篇文章中我們的基因體時代-AI, Data和生物資訊 Overview,也會持續調整!我們的基因體時代是我經營的部落格,如有對於生物資訊、檢驗醫學、資料視覺化、R語言有興趣的話,可以來交流交流!
