上一篇我們的基因體時代-AI, Data和生物資訊 Day22- 基因註釋資料在Bioconductor中的物件:AnnotationHub在示範間接GenomicRanges的使用前,先介紹一下怎麼在R環境下使用Bioconductor裡面的資料包來取得跟人類基因體相關資訊的內容,以便進一步介紹GenomicRanges的進階用法,那這邊往下帶到可以使用一些工具來視覺化這類的資訊,這邊介紹Gviz。
我們延續上一篇的內容,先使用org.Hs.eg.db和hg19及hg38的資料來搜集跟血型基因RHD相關的資訊,想必大家可能多少都聽過所謂的Rh血型,常常電視劇裡面會提到Rh陰性血非常少見,尤其當Rh陰性孕婦懷了Rh陽性,比如最近火紅的韓劇機智醫師裡面就剛好有這個案例,Rh血型陽性在台灣其實是指其具有RhD基因,其會產生所謂的RhD抗原,這個RhD基因就是我們這邊想要來當作例子。
# load library
library(org.Hs.eg.db)
library(TxDb.Hsapiens.UCSC.hg38.knownGene)
library(TxDb.Hsapiens.UCSC.hg19.knownGene)
library(Gviz)
TxDb.hg38 <- TxDb.Hsapiens.UCSC.hg38.knownGene
TxDb.hg19 <- TxDb.Hsapiens.UCSC.hg19.knownGene
# use AnnotationHub to query gene ID
gene.inform.df <- AnnotationDbi::select(org.Hs.eg.db,
keys = c("RHD"),
keytype = c("SYMBOL"),
columns = c("ENTREZID"))
# use previous acquire gene ID inform to query gene annotation data
# Compare version hg19 and version hg38
hg19.cd.range.inform.df <- AnnotationDbi::select(TxDb.hg19,
keys = gene.inform.df$ENTREZID,
keytype = c("GENEID"),
columns = c("CDSCHROM", "CDSSTART","CDSID",
"CDSEND", "CDSID","CDSSTRAND")) %>%
mutate(WIDTH=CDSEND-CDSSTART)
hg38.cd.range.inform.df <- AnnotationDbi::select(TxDb.hg38,
keys = gene.inform.df$ENTREZID,
keytype = c("GENEID"),
columns = c("CDSCHROM", "CDSSTART","CDSID",
"CDSEND", "CDSID","CDSSTRAND")) %>%
mutate(WIDTH=CDSEND-CDSSTART)
從上上篇的我們的基因體時代-AI, Data和生物資訊 Day21- 基因註釋資料在Bioconductor中物件:IRanges和GenomicRanges內有介紹怎麼在R的環境下利用Bioconductor的兩個專門用來建立基因資訊資料結構的包IRange和GenomicRanges,這邊則是近一步直接用來自於人類基因組資料包取得的資訊來建立物件,以便進一步做分析和比較。
# construct IRange from annotationHub acquired result
hg19.Bloodtype_IRange <- IRanges::IRanges(start = hg19.cd.range.inform.df$CDSSTART,
width = hg19.cd.range.inform.df$WIDTH,
names = hg19.cd.range.inform.df$CDSID)
hg38.Bloodtype_IRange <- IRanges::IRanges(start = hg38.cd.range.inform.df$CDSSTART,
width = hg38.cd.range.inform.df$WIDTH,
names = hg38.cd.range.inform.df$CDSID)
# construct GRanges from IRanges
hg19.Bloodtype_GRanges <- GRanges(seqnames = Rle(hg19.cd.range.inform.df$CDSCHROM),
ranges = hg19.Bloodtype_IRange,
strand = Rle(hg19.cd.range.inform.df$CDSSTRAND),
geneid = hg19.cd.range.inform.df$GENEID,
length = hg19.cd.range.inform.df$WIDTH )
hg38.Bloodtype_GRanges <- GRanges(seqnames = Rle(hg38.cd.range.inform.df$CDSCHROM),
ranges = hg38.Bloodtype_IRange,
strand = Rle(hg38.cd.range.inform.df$CDSSTRAND),
geneid = hg38.cd.range.inform.df$GENEID,
length = hg38.cd.range.inform.df$WIDTH )
在處理這類基因資訊,其實視覺化是很重要的工具,可以快速理解一些資料間的差異,比如這邊的代碼便是用來顯示如何快速裡用Gviz包來畫基因資訊圖,而下面就是用來建立最後要用plotTracks函數畫圖前,要建立的畫軌。
# use Gviz to visualize the gene range data
axTrack <- GenomeAxisTrack()
hg19.bloodTypeTrack <- AnnotationTrack(hg19.Bloodtype_GRanges,
name='hg19 RHD gene',
genome='hg19')
hg38.bloodTypeTrack <- AnnotationTrack(hg38.Bloodtype_GRanges,
name='hg38 RHD gene',
genome='hg38')
hg38.idxTrack <- IdeogramTrack(genome="hg38", chromosome="chr1", name='chr1')
hg19.idxTrack <- IdeogramTrack(genome="hg19", chromosome="chr1", name='chr1')
# plot hg38 version
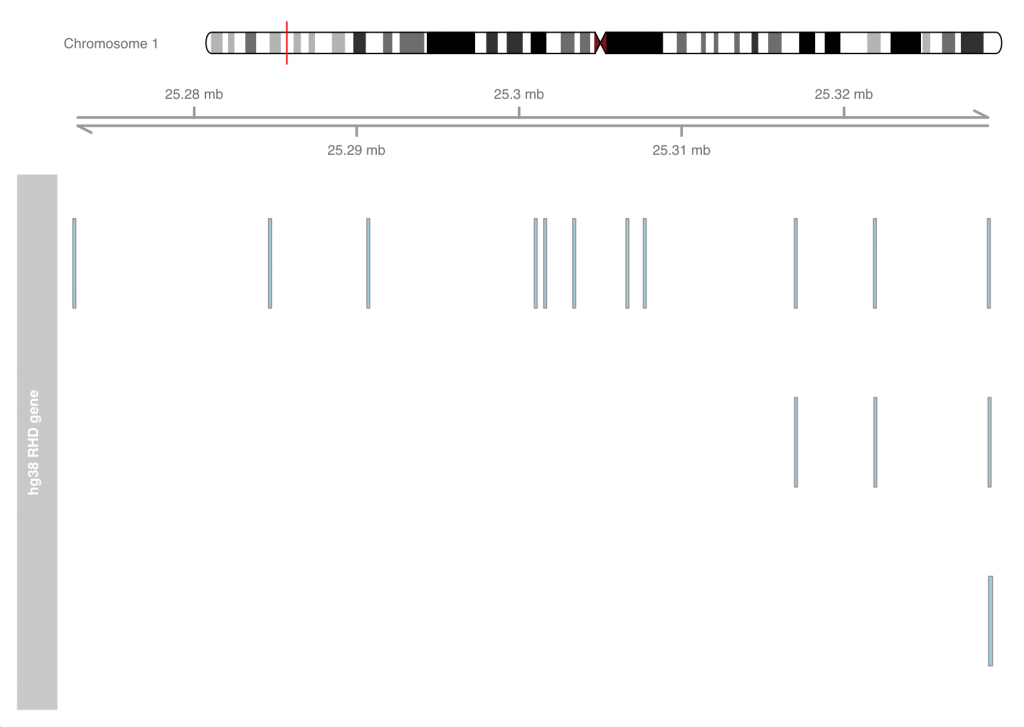
plotTracks(list(hg38.idxTrack, axTrack, hg38.bloodTypeTrack))
# plot hg19 version
plotTracks(list(hg19.idxTrack, axTrack, hg19.bloodTypeTrack))
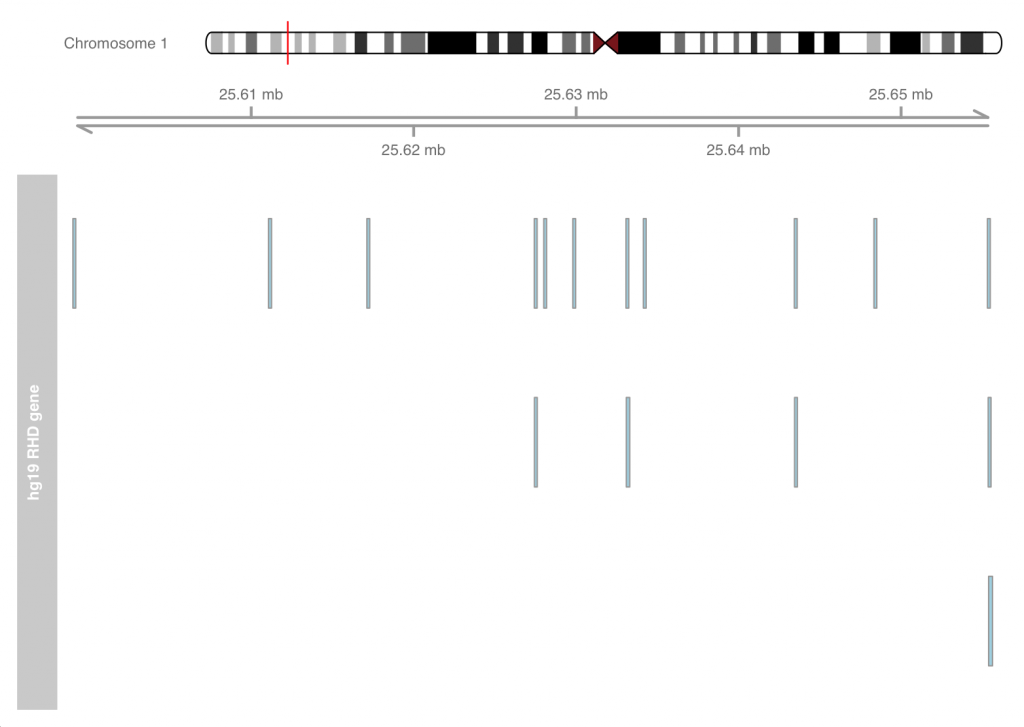
這樣其實馬上就可以看出在不同版本的基因資訊中,在RhD基因的基因轉錄區域資訊其實是有變化的,另外,本質上第一號染色體其實肉眼看也能發現不同,藉由這類視覺化其實能大幅幫助處理相關生物數據的能力!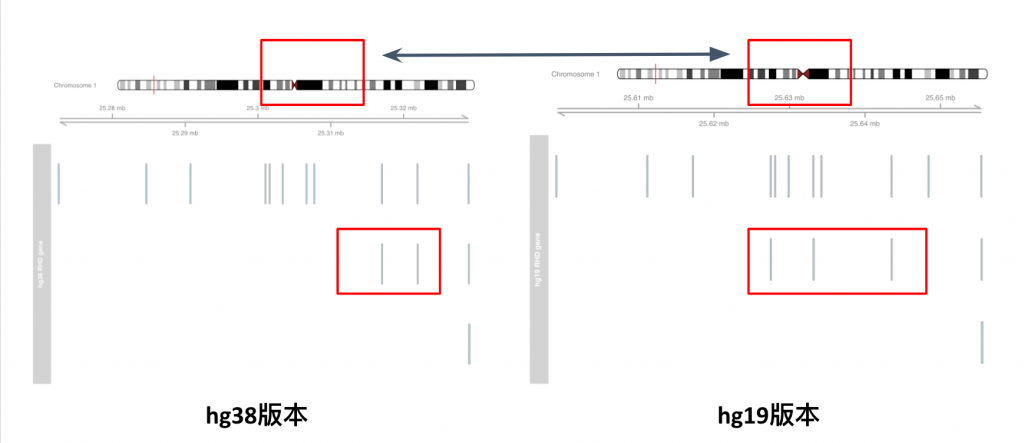
接下來再繼續深化怎麼使用Bioconductor內的工具來使用、分析和視覺化相關基因的資訊!
Chapter 16. Visualizing Genomic Data Using Gviz and Bioconductor. Statistical Genomics. 2016
Ou J, Zhu LJ (2019). “trackViewer: a Bioconductor package for interactive and integrative visualization of multi-omics data.” Nature Methods, 16, 453–454. doi: 10.1038/s41592-019-0430-y, https://doi.org/10.1038/s41592-019-0430-y.
這個月的規劃貼在這篇文章中我們的基因體時代-AI, Data和生物資訊 Overview,也會持續調整!我們的基因體時代是我經營的部落格,如有對於生物資訊、檢驗醫學、資料視覺化、R語言有興趣的話,可以來交流交流!
