實際收案拿到的資料中常常會有遺失值(Missing Value)出現,產生遺失值的原因有很多種,包含個案中離、試驗者拒絕回答、標記不清處等等原因都有可能。
⇒進行資料分析時我們不能直接忽略這些遺失值,直接忽略它們可能會造成以下幾種問題:
在進行遺失值處理前,我們首先要去了解資料遺失值是哪種的缺失:
完全隨機缺失 Missing Completely at Random (MCAR)
隨機缺失 Missing at Random (MAR)
非隨機缺失 Missing Not at Random (MNAR)
完全隨機缺失
就是資料中的遺失值是隨機分布的,這種狀態比較好處理。
隨機缺失
是指特定變數(族群)下,會有缺失資料出現(遺失值是有關連性的)。
非隨機缺失
遺失值的產生是有意義的,(Censored missing的資料,例如數值低於某個臨界值時,不去輸入資料或以 . 代替)。這種遺失值需要去做特別的處理。
遺失值在資料裡的的表現形態有很多種,包括. NA 空白等等,在 R 裡是顯示為NA(Not Available)。
missing_value <- c(1, 2, 3, NA, 4, 5)
is.na( )可以用來檢查是否有遺失值。
> missing_value
[1] 1 2 3 NA 5
> is.na(missing_value)
[1] FALSE FALSE FALSE TRUE FALSE
如果是要檢查資料內的遺失值:
data <- data.frame(
col_a=c( 0, 10, NA, 4, NA),
col_b=c(-15,13,0,-5,-1),
col_c=c( 1, 2, 3, NA, 5)
)
sum(is.na(data)) # 計算缺失值的個數
sum(complete.cases(data)) # 完整資料的資料筆數
which( colSums(is.na(data)) >0) # 有缺失值的欄位,(回傳Column Name 和 Column Index)
complete.cases(data) # 當一筆資料(row)是完整的,回傳TRUE;當一筆資料有遺漏值,回傳FALSE
> data
col_a col_b col_c
1 0 -15 1
2 10 13 2
3 NA 0 3
4 4 -5 NA
5 NA -1 5
> sum(is.na(data)) # 計算缺失值的個數
[1] 3
> sum(complete.cases(data)) # 完整資料的資料筆數
[1] 2
>
> which( colSums(is.na(data)) >0) # 有缺失值的欄位,(回傳Column Name 和 Column Index)
col_a col_c
1 3
> complete.cases(data) # 當一筆資料(row)是完整的,回傳TRUE;當一筆資料有遺漏值,回傳FALSE
[1] TRUE TRUE FALSE FALSE FALSE
接下來介紹的方法主要是對 MCAR、MAR 遺失值的處理方法。
直接移除有遺失值的資料:
remove_data<- data[complete.cases(data), ]
> data
col_a col_b col_c
1 0 -15 1
2 10 13 2
3 NA 0 3
4 4 -5 NA
5 NA -1 5
> remove_data
col_a col_b col_c
1 0 -15 1
2 10 13 2
示範 pairwise 的用各欄的平均值填補缺失位置:
data <- data.frame(
col_a=c( 0, 10, NA, 4, NA, 1, 8, 6, 4, 5),
col_b=c(-15,13,0,-5,-1, 8, -6, 4, 9, -9),
col_c=c( 1, 2, 3, NA, 5, 6, 7, 8, 9, 10)
)
> data
col_a col_b col_c
1 0 -15 1
2 10 13 2
3 NA 0 3
4 4 -5 NA
5 NA -1 5
6 1 8 6
7 8 -6 7
8 6 4 8
9 4 9 9
10 5 -9 10
mean_col1 <- mean(data[, 1], na.rm = T) # column 1 的平均數(na.rm = T 忽略遺失值)
na_c1rows <- is.na(data[, 1]) # column 1 中,有遺失值存在的資料(回傳TRUE/FALSE)
mean_col3 <- mean(data[, 3], na.rm = T) # column 3
na_c3rows <- is.na(data[, 3])
# 用 mean( column 1 ) ,填補第一欄位的遺漏值
data[na_c1rows, 1] <- mean_col1
data[na_c3rows, 3] <- mean_col3
> data
col_a col_b col_c
1 0.00 -15 1.000000
2 10.00 13 2.000000
3 4.75 0 3.000000
4 4.00 -5 5.666667
5 4.75 -1 5.000000
6 1.00 8 6.000000
7 8.00 -6 7.000000
8 6.00 4 8.000000
9 4.00 9 9.000000
10 5.00 -9 10.000000
多重插補 Multiple Imputation
它是從有遺失值的資料集中生成一個完整的資料集,重複去模擬資料的遺失,並試著去填補缺失值。
常見的方法之一就是使用套件mice,它是使用利用 Chained Equations 產生插補值填補,它的優點是能夠差補混和類型(類別、二元類別、連續數值)的資料。
Multivariate Imputation by Chained Equations (mice)示意圖:
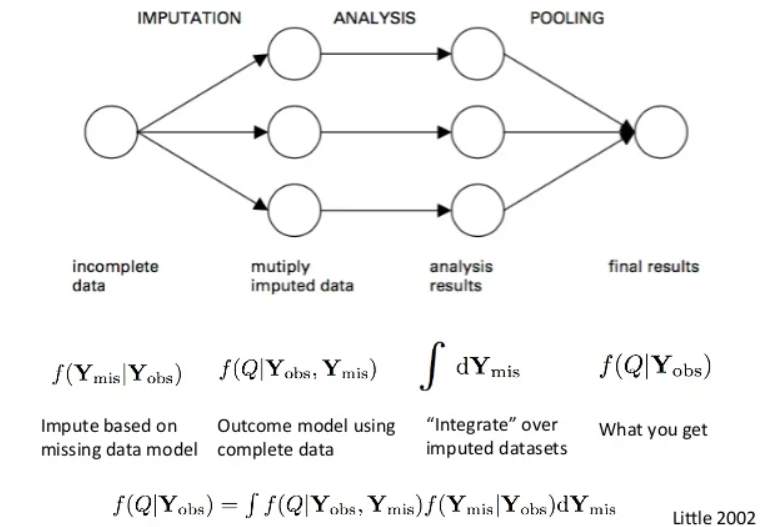
圖片來源:https://hmtb.tistory.com/34
基本步驟如下:
install.packages("mice")
require(mice) # mice套件
imp <- mice(data, m) # 包含m個插補數據集的列表,默認 m = 5
## fit包含m個單獨統計分析結果的列表對象
fit <- with(imp, analysis) # analysis:設定應用於m個插補數據集的統計分析方法
## pooled是一個包含這m個統計分析平均結果的列表對象
pooled <- pool(fit)
summary(pooled)
complete(imp,n) # 可以看見到其中一筆(第n筆)填補完的資料
範例一:
以CART決策樹進行遺失值預測填補
data# 原始資料(有遺失值)
mice.data <- mice(data,
m = 3, # 產生三個被填補好的資料表
maxit = 50, # max iteration
method = "cart", # 使用CART決策樹,進行遺失值預測
seed = 188) # set.seed(),令抽樣每次都一樣
## 填補好的資料:因為m=3,所以會有三個填補好的資料集,可以用以下方式取出
complete(mice.data, 1) # 1st data
complete(mice.data, 2) # 2nd data
complete(mice.data, 3) # 3rd data
## 觀察填補好的資料
df <- complete(mice.data, 1)
head(df)
程式碼參考來源: https://www.rpubs.com/skydome20/R-Note10-Missing_Value
範例二:
以哺乳動物睡眠資料(sleep)為例。
目標: 想知道做夢的時間(Dream)與動物壽命(Span,單位:年)及妊娠期(Gest,單位:天)的線性關係。
install.packages("VIM") # 從VIM套件取得 sleep 資料
library(VIM)
data(sleep, package= "VIM" )
head(sleep)
require(mice) # mice套件
imp <- mice(sleep, seed=123) # 因為會隨機差補所以設定種子數(seed)固定 #默認 m = 5
fit <- with(imp, lm(Dream~Span+Gest)) # 統計分析方法lm()
pooled <- pool(fit)
summary(pooled)
> head(sleep)
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
1 6654.000 5712.0 NA NA 3.3 38.6 645 3 5 3
2 1.000 6.6 6.3 2.0 8.3 4.5 42 3 1 3
3 3.385 44.5 NA NA 12.5 14.0 60 1 1 1
4 0.920 5.7 NA NA 16.5 NA 25 5 2 3
5 2547.000 4603.0 2.1 1.8 3.9 69.0 624 3 5 4
6 10.550 179.5 9.1 0.7 9.8 27.0 180 4 4 4
也可以從 pooled 觀察插補訊息,例如fmi(Fraction of Missing Information)。
> pooled
Class: mipo m = 5
term m estimate ubar b t dfcom df riv lambda
1 (Intercept) 5 2.525630585 5.706649e-02 5.362455e-03 6.350144e-02 59 45.33857 0.11276225 0.10133544
2 Span 5 -0.004576084 1.271602e-04 1.194255e-06 1.285933e-04 59 56.36165 0.01127008 0.01114448
3 Gest 5 -0.003894918 1.962279e-06 6.919705e-08 2.045315e-06 59 53.56957 0.04231634 0.04059837
fmi
1 0.13851753
2 0.04446079
3 0.07451772
df <- complete(imp) # 看填補完的資料
head(df)
> head(df)
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
1 6654.000 5712.0 3.2 0.0 3.3 38.6 645 3 5 3
2 1.000 6.6 6.3 2.0 8.3 4.5 42 3 1 3
3 3.385 44.5 10.9 1.5 12.5 14.0 60 1 1 1
4 0.920 5.7 13.8 2.8 16.5 5.0 25 5 2 3
5 2547.000 4603.0 2.1 1.8 3.9 69.0 624 3 5 4
6 10.550 179.5 9.1 0.7 9.8 27.0 180 4 4 4
還有很多其他方法可以填補遺失值,例如:
impute 套件)pcaMethods 套件)統計諮詢課上課內容, [STAT4008]統計諮詢, 課程系統碼: H240200。
R筆記–(10)遺漏值處理(Impute Missing Value)
https://www.rpubs.com/skydome20/R-Note10-Missing_Value
MULTIPLE IMPUTATION IN STATA
https://stats.oarc.ucla.edu/stata/seminars/mi_in_stata_pt1_new/
Missing Data Analysis with mice. Firouzeh Noghrehchi.
https://web.maths.unsw.edu.au/~dwarton/missingDataLab.html
결측값 데이터 다루기(處理缺失值數據), Jane Jeon
https://hmtb.tistory.com/34
https://github.com/lifeisgouda
