這幾天要來講應用在「分類」問題上的機器學習模型。預計將會介紹到
這幾種模型以及它們在 R 上的操作。
羅吉斯迴歸是線性回歸的延伸,線性回歸的反應變數 (Y, target/ label) 通常是連續型的數值,但羅吉斯迴歸是應用於二元類別變數上,簡單來說就是反應變數 (Y) 只有兩個種類,例如: (0,1) / (A,B)/ (是,否)/ (有,沒有)。
[註] 羅吉斯迴歸只有限制反應變數 (Y) 是二元類別變數。
解釋變數並沒有限制,解釋變數 (X) 不論是連續數值或是類別的變數都能夠建立模型。
Logistic Curve :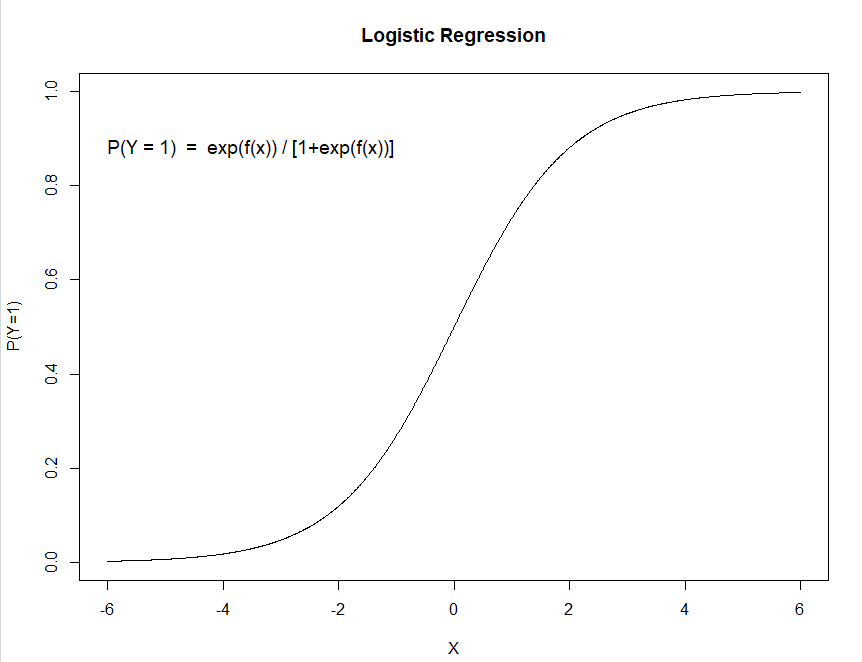
Y 為 「類別1」 的機率: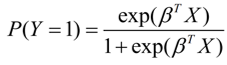
羅吉斯迴歸模型( logit link ):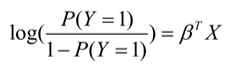
glm(y ~ x , family=, data=) 是 R 裡內建的 Generalized Linear Model( GLM ) 的廣義線性模型函數,使用方式和線性迴歸模型的 lm( )很像,Y 放”反應變數” X 放”解釋變數”。
建立羅吉斯迴歸: glm(Y ~ X, family = “binomial”, data=data)
建構羅吉斯迴歸模型時,要設定 family = “binomial",這樣才能對應到 logit link 的 Link function 上。
[補充] 其他 family type和 link function
以下使用 Diabetes data 糖尿病資料做一個 Logistic regression 的小示範,這裡是示範的程式碼:
首先需要從mlbench這個package裡載入資料。
這個例子裡:
反映變數 Y: {有沒有糖尿病} (1: 沒有;2: 有),
解釋變數 X: 有8個變數,包括 懷孕次數、血糖等等。
在建模之前一樣需要觀察一下資料的型態:
install.packages("mlbench") # Diabetes data
library(mlbench)
data("PimaIndiansDiabetes")
data<- PimaIndiansDiabetes
head(data) # 觀看前6筆資料
summary(data)# 連續型資料:會看到 Qu. #類別型資料:會看到不同類別的資料個數
str(data) # 列出資料內每個欄位的狀態


可以看到糖尿病( Diabetes )是二元的類別(Factor)變數,並且被編碼成 1("neg"), 2("pos")。
為了後續建模不希望有 Overfitting 或 Under fitting 的情況,所以我們先把資料分割為training 跟testing data,這裡舉例為 70% 、 30% 的分割,先以 training data建立模型,再以 testing data 檢驗我們的模型結果。
set.seed(1) # 固定sample()抽樣的結果
train_rows<- sample(x=1:nrow(data), size=ceiling(0.70*nrow(data) ))# 70%的data
train<- data[train_rows,] # 70%當training data
test<- data[-train_rows,]# 30%當testing data
[注意]
⇒ glm( )函數在建羅吉斯迴歸時,需要帶入的( Y )資料為0或1的二元資料,所以要先進行資料處裡。
這裡是將 Y 直接 -1 ,變成了(0: 沒有糖尿病;1: 有糖尿病)的編碼。
如果忘了 0, 1 分別代表什麼意思,可以用contrast()函數做個確認。
#為了放入glm(),將反應變數factor從編碼"1、2" 轉成"0和1"
train$diabetes = as.numeric(train$diabetes) -1
test$diabetes = as.numeric(test$diabetes) -1
contrasts(data$diabetes) # neg: 0, pos: 1
接著建立羅吉斯迴歸模型( Logistic regression model )進行判斷、分類
model_glm = glm(diabetes ~ ., data = train, family = "binomial")
summary(model_glm)
> summary(model_glm)
Call:
glm(formula = diabetes ~ ., family = "binomial", data = train)
Deviance Residuals:
Min 1Q Median 3Q Max
-2.4926 -0.7722 -0.4314 0.8022 2.7578
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -7.792026 0.838851 -9.289 < 2e-16 ***
pregnant 0.140563 0.036508 3.850 0.000118 ***
glucose 0.031374 0.004139 7.580 3.45e-14 ***
pressure -0.015932 0.006096 -2.613 0.008965 **
triceps -0.006229 0.007793 -0.799 0.424104
insulin -0.001126 0.001128 -0.999 0.317953
mass 0.098980 0.018055 5.482 4.20e-08 ***
pedigree 0.812996 0.352517 2.306 0.021096 *
age 0.011988 0.010661 1.124 0.260803
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 705.70 on 537 degrees of freedom
Residual deviance: 531.35 on 529 degrees of freedom
AIC: 549.35
Number of Fisher Scoring iterations: 5
建立完羅吉斯迴歸模型,我們可以去做模型推論:
透過邏輯斯迴歸的logit link,我們可以由exp(係數beta)去解釋發生事件(Y=1, 患有糖尿病)的勝算比。
勝算比(Odds ratio)= exp(係數beta)。
舉例來說:
pregnant的coefficient 為0.14,那exp(0.14)= 1.15,表示每增加一次懷孕次數,得到糖尿病的勝算提高1.15倍。
predict(model, type)在 R 裡建構羅吉斯迴歸時,如果要進行預測時依樣使用函式predict( )。
預設的輸出為type = "link",輸出的值是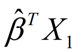 。
。
當我們希望輸出一個 P(Y=1) 的機率值(介於0~1之間)時,
需要更改設定為type = "response",這時輸出的值就會是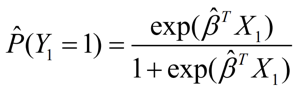 。
。
#predict(model,type,newdata)
#這裡用head()觀察predict()函數前6項輸出結果
head(predict(model_glm)) # predict()預設type="link"
head(predict(model_glm, type = "link"))# 每個樣本分別代入X的預測結果Y
## type='response',機率P(Y=1∣X=x)
head(predict(model_glm, type='response'))
# type=link的數值經過轉換,可以變成type='response'的機率
exp(-0.4363519)/(1+exp(-0.4363519)) #以資料679為例
> head(predict(model_glm, type = "link"))
679 129 509 471 299 270
-0.4363519 -1.4736009 -1.8532542 0.2277766 0.2166410 0.3224093
> head(predict(model_glm, type='response'))
679 129 509 471 299 270
0.3926106 0.1863959 0.1354913 0.5566992 0.5539494 0.5799113
> exp(-0.4363519)/(1+exp(-0.4363519)) #以資料679為例
[1] 0.3926106
得到預測機率後,可以使用ifelse(result > c , 1, 0)給定一個閥值 c 去進行「分類」。
舉例來說: 如果預測機率值大於特定閥值(例如 0.5),會將那筆資料的 Y 分類為 1,其他為0。
#得到機率後,給定一個閥值進行"分類"
result = predict(model_glm, type='response')
class_result = ifelse(result > 0.5,1,0)# 閥值c設定為0.5用來區隔不同類
head(class_result)
> head(result)
679 129 509 471 299 270
0 0 0 1 1 1
如果想對新資料進行預測,使用predict( model, type, newdata) :
#自己假設一筆資料
new_data=data.frame(pregnant=0,glucose=111,pressure=62,triceps=0,
insulin=0,mass=22.6,pedigree=0.142,age=21)
predict(model_glm,type = "response",
newdata = new_data)
class_result = ifelse(result > 0.5,'1: pos','0: neg')
print(class_result)
> result = predict(model_glm,type = "response",newdata = new_data)
> class_result = ifelse(result > 0.5,'1: pos','0: neg')
> print(class_result)
1
"0: neg"
這裡以 training data 建立模型,再對 testing data 進行預測、分類,進而檢驗我們的模型成效。
result = predict(model_glm,newdata=test,type='response')
result = ifelse(result > 0.5,1,0)
table(predicted = result, actual = test$diabetes)
#Accuracy
(143+41)/230
準確率的計算為斜對角數目(分類正確的數目)除以資料總數。
> result = predict(model_glm,newdata=test,type='response')
> result = ifelse(result > 0.5,1,0)
> table(predicted = result, actual = test$diabetes)
actual
predicted 0 1
0 143 31
1 15 41
> #Accuracy
> (143+41)/230
[1] 0.8
install.packages("caret")
library(caret)
confusionMatrix(data=as.factor(result),reference=as.factor(test$diabetes))
Confusion Matrix and Statistics
Reference
Prediction 0 1
0 143 31
1 15 41
Accuracy : 0.8
95% CI : (0.7424, 0.8497)
No Information Rate : 0.687
P-Value [Acc > NIR] : 8.356e-05
Kappa : 0.5051
Mcnemar's Test P-Value : 0.02699
Sensitivity : 0.9051
Specificity : 0.5694
Pos Pred Value : 0.8218
Neg Pred Value : 0.7321
Prevalence : 0.6870
Detection Rate : 0.6217
Detection Prevalence : 0.7565
Balanced Accuracy : 0.7373
'Positive' Class : 0
install.packages("pROC")
library("pROC")
test_prob = predict(model_glm, newdata = test, type = "response")
par(pty = "s") #指定畫布繪製成正方形的大小
test_roc = roc(test$diabetes ~ test_prob, plot = TRUE,
print.auc = TRUE,legacy.axes=TRUE)
#legacy.axes=TRUE 將 x軸改成 1 - Specificity
曲線下的面積稱為 AUC (Area Under Curve),AUC面積越大越好,表示模型配適越好。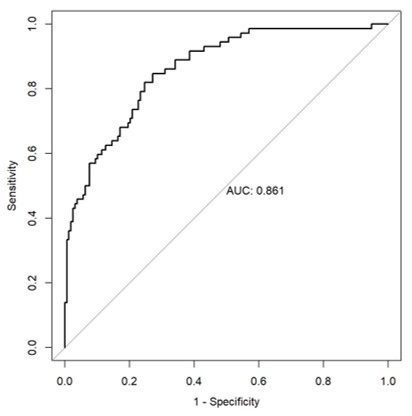
羅吉斯迴歸也同樣能進行正規化,和一般正規化迴歸一樣使用 glmnet() 函式,不過裡面要設定 family = "binomial" 。
glmnet(
family =若y是連續值,設"gaussian" ,
若y是二元分類,設"binomial",
若y是多元分類,設"multinomial",
alpha = 0(Ridge) 、介於0~1介之間(Elastic-net)、1(Lasso),
lambda = 懲罰值 )
An Introduction to Statistical Learning with Applications in R. 2nd edition. Springer. James, G., Witten, D., Hastie, T., and Tibshirani, R. (2021).
