上一篇我們的基因體時代-AI, Data和生物資訊 Day12-基因療法中之腺病毒載體與機器學習 我們分享了另一篇也是利用資料和簡單數據分析來輔助基因治療中的腺病毒相關病毒載體設計的文章。
發現有點拖延到預計規劃的內容,想說只好忍痛先往下,本來預定第二週要開始分享一些分子生物學和基因定序相關的資料格式,但寫了快兩週,還沒有把一些經典的生物領域機器學習應用分享完,可能等之後再找機會分享,但至少讓大家知道所機器學習在生物領域可以應用的地方太多了,不要在千篇一律說什麼胸部X光或是電腦斷層的AI預測了。
為了讓之後分享各種基因定序資料格式或是相關概念比較清楚,這邊先來的背景介紹!

一個人的細胞中,都有23對染色體,染色體是由DNA和結構蛋白所組成的,而DNA則是由四種鹼基組成:A、T、C、G。這些鹼基的排列組合就會決定一個細胞會產生什麼蛋白質、怎麼去調控、怎麼去跟其他細胞互動、怎麼形成整個複雜組織,所以要是能讀取其中的序列,將會幫助我們理解人的疾病和一些生理現象。
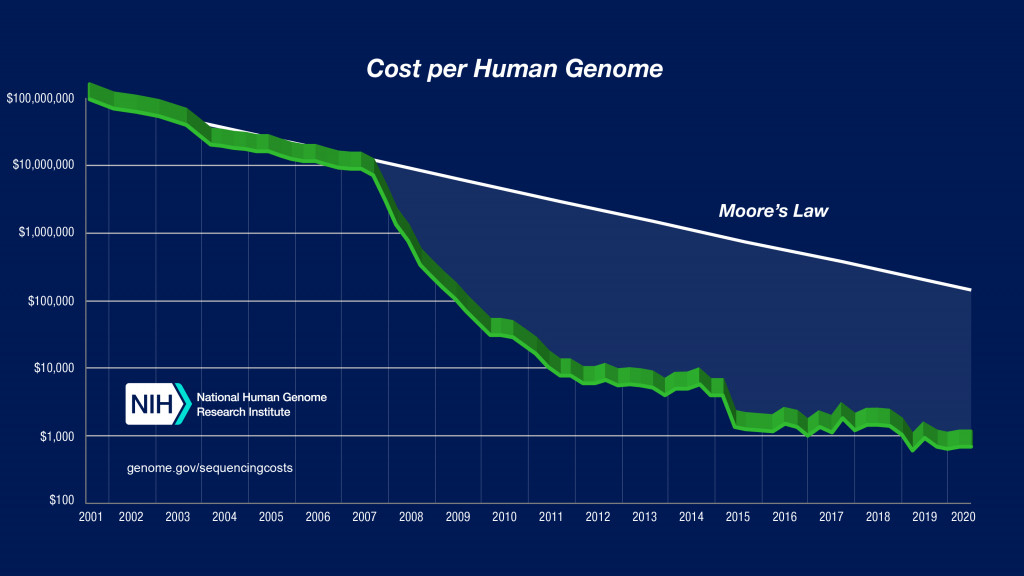
就是因為定序的便宜,所以資料量大增,這部分可以參考第一天的內容我們的基因體時代-AI, Data和生物資訊 Day01- 超越摩爾定律的資料增長
,這也是為何基因體學開始進入所謂資料科學領域的原因,當價錢很貴的時候,資料量相對的產出就少,唯有當價錢便宜才有可能越來越多,直到現在是資料多到分析的人不夠以及儲存空間不足。
fasta格式其實是個非常有歷史淵源的,他是來自於美國NCBI創辦人David J. Lipman,原本這其實是一個演算法的名稱,在1987年的時候所發表,是用來作為基因序列的搜尋引擎所用,也就是拿到一個序列,怎麼知道他來自於哪,或是跟誰很像。
作為最基本的一個生物資訊的資料格式,它可以用來儲存DNA或是蛋白質的序列,他的資料結構非常簡單,就長得像下面這樣:

這個資料基本上就是由兩行組成,第一行是以>開頭,用來描述這個序列是什麼,第二行則是序列本身的內容。
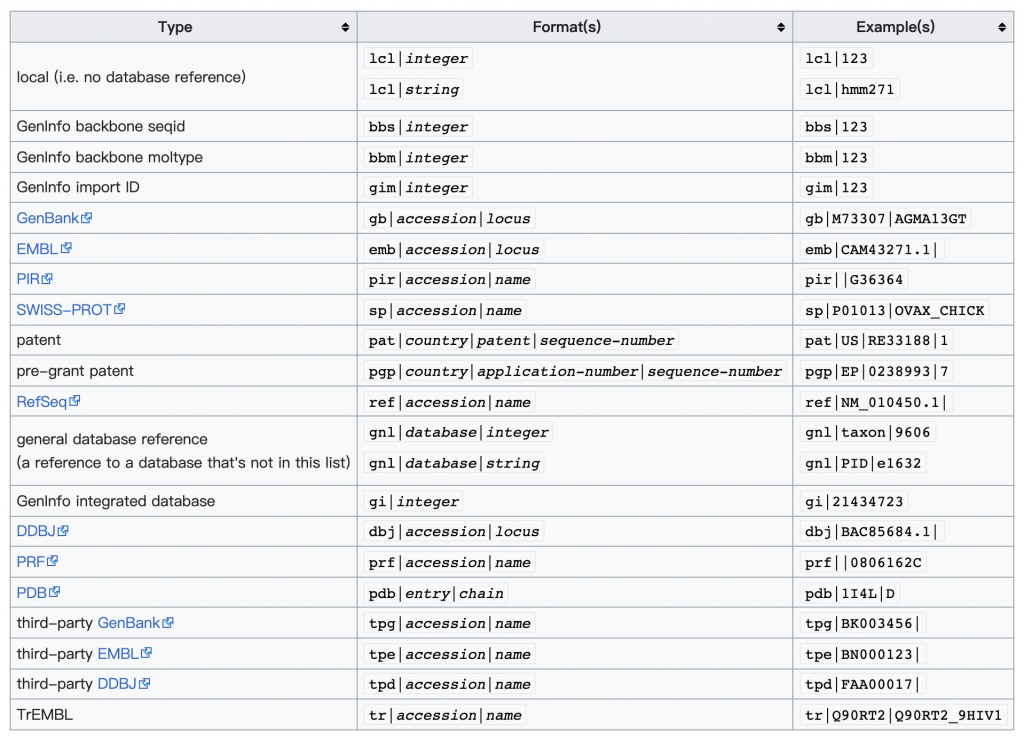
另一方面我們可以根據這個開頭的編碼模式,知道這個資料來自於那個現行生物資料庫!
閱讀參考
Fasta format for nucleotide sequence
https://www.ncbi.nlm.nih.gov/genbank/fastaformat/
Query use format
https://blast.ncbi.nlm.nih.gov/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=BlastHelp
Goodwin, S., McPherson, J. & McCombie, W. Coming of age: ten years of next-generation sequencing technologies. Nat Rev Genet 17, 333–351 (2016). https://doi.org/10.1038/nrg.2016.49
Logsdon, G.A., Vollger, M.R. & Eichler, E.E. Long-read human genome sequencing and its applications. Nat Rev Genet 21, 597–614 (2020). https://doi.org/10.1038/s41576-020-0236-x
Tslil Gabrieli, Hila Sharim, Dena Fridman, Nissim Arbib, Yael Michaeli, Yuval Ebenstein, Selective nanopore sequencing of human BRCA1 by Cas9-assisted targeting of chromosome segments (CATCH), Nucleic Acids Research, Volume 46, Issue 14, 21 August 2018, Page e87, https://doi.org/10.1093/nar/gky411
López-Girona, E., Davy, M.W., Albert, N.W. et al. CRISPR-Cas9 enrichment and long read sequencing for fine mapping in plants. Plant Methods 16, 121 (2020). https://doi.org/10.1186/s13007-020-00661-x
Gilpatrick, T., Lee, I., Graham, J.E. et al. Targeted nanopore sequencing with Cas9-guided adapter ligation. Nat Biotechnol 38, 433–438 (2020). https://doi.org/10.1038/s41587-020-0407-5
Payne, A., Holmes, N., Clarke, T. et al. Readfish enables targeted nanopore sequencing of gigabase-sized genomes. Nat Biotechnol 39, 442–450 (2021). https://doi.org/10.1038/s41587-020-00746-x
這個月的規劃貼在這篇文章中我們的基因體時代-AI, Data和生物資訊 Overview,也會持續調整!我們的基因體時代是我經營的部落格,如有對於生物資訊、檢驗醫學、資料視覺化、R語言有興趣的話,可以來交流交流!
