pb-16S-nf 本身即整合 QIIME2 軟體緣故,所產出的圖檔幾乎相同,
本篇則先介紹資料前處理、品質管制、物種分配,
並提供該檔案路徑檔名,供讀者查閱 :
Overview 確認樣本名稱、數量與條數TGS_sewage/allResults/summary_demux/samples.demux.summary.qzv
Interactive Quality Plot 序列品質查看TGS_sewage/allResults/results/visualize_biom.html
因 TGS Quality Score 過高 (>45 甚至 90 相當常見),
使得 QIIME2介面的縱軸無法相容,
在這裡會使用上述路徑做觀察 :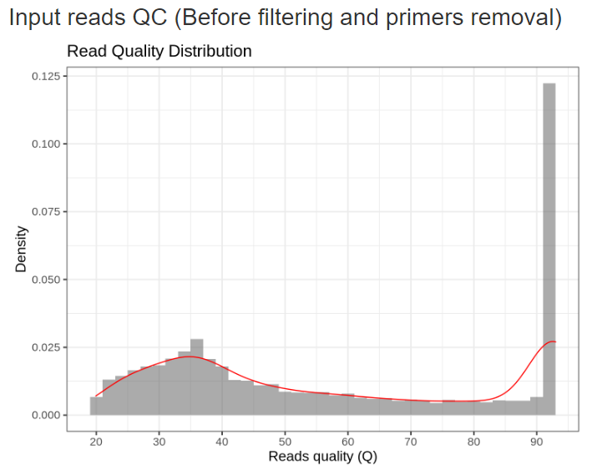
還是可以偷偷看一下,會發現顯示報錯 :
TGS_sewage/allResults/summary_demux/samples.demux.summary.qzv
資料加工壓縮基礎解說 : [Day 07]
TGS_sewage/allResults/results/dada2_stats.qzv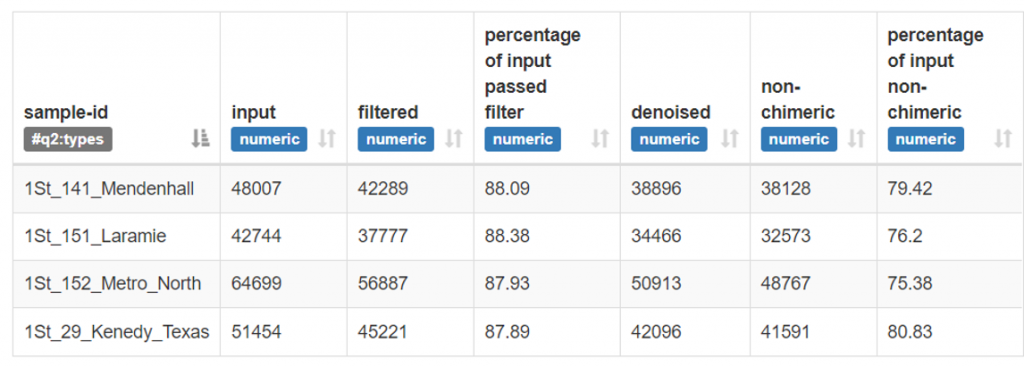
TGS_sewage/allResults/results/dada2_table.qzv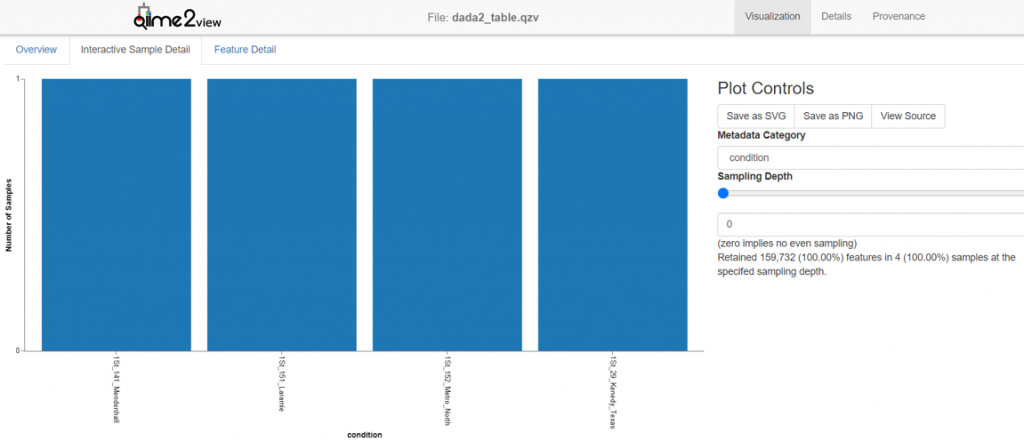
若想試試啟動 QIIME2-2022.8 環境繼續手動分析往下做也可以 !
其原始檔案(.qza) 位於 TGS_sewage/allResults/dada2/資料夾內。
還記得[Day 23] 提到 pb-16S-nf 使用
VSEARCH 與 Naive Bayes classifier 兩種分類器嗎?
因此在物種分類結果上也會有兩種檔案可以參考,
分析者可以觀察分類器的表現決定,也會發現其層級與NGS 二代分析相比,
三代分析多數都能達到種 (Species) 層級 :
TGS_sewage/allResults/results/taxa_barplot.qzv
TGS_sewage/allResults/results/taxa_barplot_vsearch.qzv

TGS_sewage/allResults/results/krona.qzv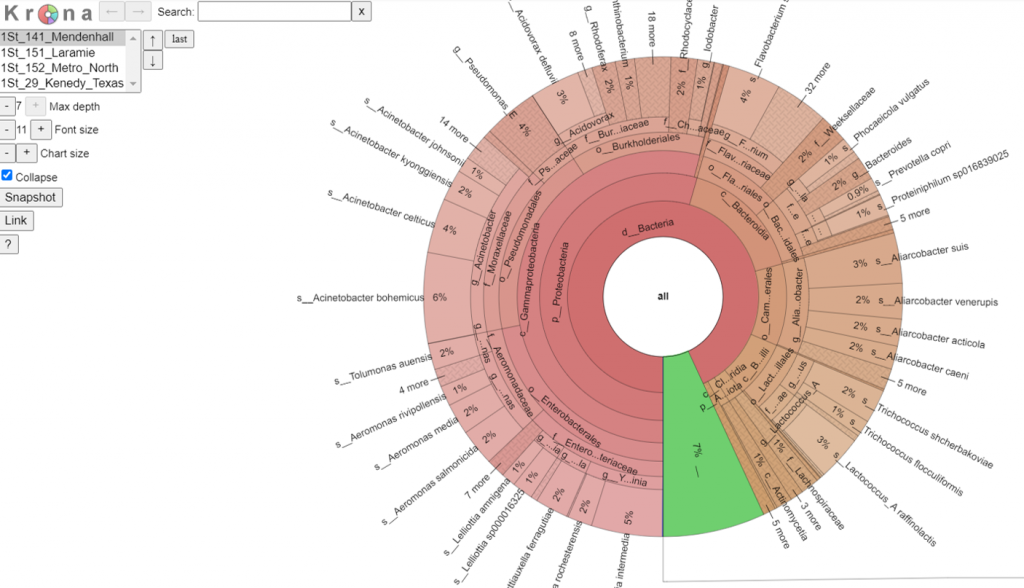
pb-16S-nf 由於流程皆自動化的關係,
在稀疏曲線的取樣深度 (Sampling depth) 也交給了程式,
可在TGS_sewage/allResults/rarefaction_depth_suggested.txt 檢視程式給的值,
以範例檔案來說是 37867 ,經由檢視TGS_sewage/allResults/results/dada2_table.qzv後,
會發現有1個樣本被淘汰了 (意即剩3個樣本進入統計分析與繪製)。
若這樣的結果不是你想要的,如想全部保留,
可以使用--rarefaction_depth指令重新設定取樣深度 :
nextflow run main.nf \
--input TGS_sewage/manifest.tsv \
--metadata TGS_sewage/sample-metadata.tsv \
-profile conda \
--outdir TGS_sewage/allResults \
--dada2_cpu 32 \
--cutadapt_cpu 32 \
--vsearch_cpu 32 \
-resume \
--rarefaction_depth 32346
